5种虾虎鱼类线粒体COI基因序列变异及系统进化
廖 健,张 顺,龙水生,黄承勤,郭昱嵩,王中铎,刘楚吾(广东海洋大学水产学院 南海水产经济动物增养殖广东普通高校重点实验室,广东 湛江 524025)
5种虾虎鱼类线粒体COI基因序列变异及系统进化
廖健,张顺,龙水生,黄承勤,郭昱嵩,王中铎,刘楚吾
(广东海洋大学水产学院 南海水产经济动物增养殖广东普通高校重点实验室,广东 湛江524025)
摘要:获得雷州半岛红树林海区5种虾虎鱼稚幼鱼的45条线粒体COI基因序列,其T、C、A、G碱基平均含量分别为29.9%、28.4%、23.9%、17.8%。45条序列共定义23个单倍型,其中检测到变异位点184个,双带缟虾虎鱼(Tridentiger bifasciatus)单倍型比例最高(80.0%),湖栖鳍虾虎鱼(Gobiopterus lacustris)单倍型比例最低(18.2%)。Kimura 2-parameter遗传距离显示,种间遗传距离为0.137 6~0.263 5,种内遗传距离为0.000 0~0.020 8。NJ进化树及ML进化树均表明,聚为5大支系的虾虎鱼类中,阿部氏鲻虾虎鱼(Mugilogobius abei)与诸氏鲻虾虎鱼(M.chulae)亲缘关系最近,湖栖鳍虾虎鱼与其他4种虾虎鱼的亲缘关系稍远。基于群体内等位基因频率分布的中性检验结果表明,双带缟虾虎鱼(Tridentiger bifasciatus)、小口拟虾虎鱼(Pseudogobius masago)、阿部氏鲻虾虎鱼、诸氏鲻虾虎鱼4个群体存在大量低频等位基因位点,而湖栖鳍虾虎鱼群体以中等频率等位基因为主。
关键词:虾虎鱼;线粒体;COI基因;系统进化
Key words:goby; Mitochondria; COI gene; Phylogenetic
虾虎鱼类是虾虎鱼亚目全部物种的统称,是鲈形目鱼类中最大的类群[1]。其适应力强,分布广泛,从淡水湖泊、溪流到海洋生态系统均有分布,海陆交错的红树林生态系统是虾虎鱼类主要的栖息场所。全球虾虎鱼类主要集中在热带、亚热带、温带水域,个别种类仅在小范围水域出现,如湖栖鳍虾虎鱼(Gobiopterus lacustris)目前仅发现于菲律宾。虾虎鱼类主要摄食钩虾、枝角类、桡足类[2],同时为洄游鱼类幼鱼提供了生物饵料[3],在很大程度上维持红树林生态系统的物质循环与能量传递。然而虾虎鱼类种类繁多,群体间形态高度相似,难以准确进行形态学分类。随着分子生物学的发展,利用基因序列辅助分类已广泛的应用于物种分类,分子数据与传统形态分类的结合较好解决了近缘物种难以界定问题[4-5]。目前,关于虾虎鱼类形态学分类已有较系统研究,而未见虾虎鱼类分子生物学方面的报道。线粒体基因组(mtDNA)是位于真核生物核外的遗传物质,具有结构简单、分子小、进化快以及母系遗传等特征,细胞色素氧化酶I基因是mtDNA上进化速率适中的一段,是系统进化研究的优质分子标记。本研究采集雷州半岛红树林海区的5种虾虎鱼类,通过获取COI基因作为分子标记,初步分析它们的遗传进化关系,为虾虎鱼类的生物学保护和系统进化研究提供依据。
1 材料与方法
1.1材料
虾虎鱼稚幼鱼,于2014年4月至2015年4月用孔径149 μm的手抄网分别采自雷州半岛沿岸的高桥镇红树林保护区,下六镇红莳地、附城镇北家村、调风镇九龙山湿地保护区,用体积分数75%的乙醇固定或鲜活带回实验室,在OLYMPUS体视镜下观察。对于已有成鱼形态的稚幼鱼,根据体形、体纹、头形(吻形、眼)、尾形、透明度、腹鳍是否愈合及有无吸盘等易于辨识的形态特征进行初步分类、标号、拍照(佳能500D)、保存。初步物种鉴定主要参照文献[1,6-7]及FishBase等。最终的中文名、分类地位确认参照《拉汉世界鱼典》[8]。
1.2基因组DNA提取
取-80℃冻存(或新鲜)虾虎鱼肌肉组织0.5 g,采用传统苯酚-氯仿法提取DNA[9],提取的DNA经10 mg/mL琼脂糖凝胶电泳和Nanodrop 2000检测定量合格后,于4℃条件下保存备用。
1.3COI基因扩增和测序
采用通用引物对COI基因进行PCR扩增,正、反向引物序列分别为:Fish F,5′-TCAACCAAC CACAAAGACAATGGCAC-3′;Fish R,5′-TAG ACTTCTGGGTGGCCAAAGAATCA-3′[10]。引物由上海生工生物有限公司合成。反应体系为:10×Buffer(含Mg2+)2.5 μL、2.5 mmol/L dNTP 2 μL、5 μmol/L引物各1 μL、DNA模板1 μL、500 u/L Taq DNA聚合酶0.2 μL,补充ddH2O至25 μL。程序设置为:94 ℃ 1 min 20 s;94 ℃ 30 s,55 ℃ 30 s,72 ℃ 50 s,35个循环;72 ℃ 5 min;4 ℃保存。产物经10 mg/mL琼脂糖凝胶电泳检测,合格产物送至上海生工生物有限公司测序。
1.4数据分析
利用MEGA6.0对所获得的序列进行多序列比对,人工核查后,对所得序列的碱基组成、遗传颠换值、变异位点、单一突变位点、简约信息位点进行统计,计算个体间的Kimura 2-parameter遗传距离。采用Modeltest 软件确定最适合的进化模式[11]。分别采用邻位连接法(Neighbor-Joining,NJ)和最大似然法(Maximum Likelihood method,ML)构建进化树。NJ 法构建进化树Bootstrap 置信值重复抽样1 000次,ML法Bootstrap分析重复抽样100次[12],两种方法均采用Modeltest获得的参数构建系统进化树。利用DAMBE进行替代饱和性检验,并以转换数和颠换数为纵坐标,TN93法校正后的遗传距离为横坐标构建散点图。分子水平自然选择作用的检测采用基于群体内等位基因频率分布的中性检验。采用DnaSP v5软件对单倍型数目、单倍型多样性、核苷酸多样度、平均核苷酸差异进行统计。
2 结果与分析
2.1样本分类
样本经形态学分类,分别为鲻虾虎鱼属的阿部氏鲻虾虎鱼(Mugilogobius abei)和诸氏鲻虾虎鱼(M.chulae)、拟虾虎鱼属的小口拟虾虎鱼(Pseudogobius masago)、缟虾虎鱼属的双带缟虾虎鱼(Tridentiger bifasciatus),样本a仅能确定到鳍虾虎鱼属(Gobiopterus)(图1)。
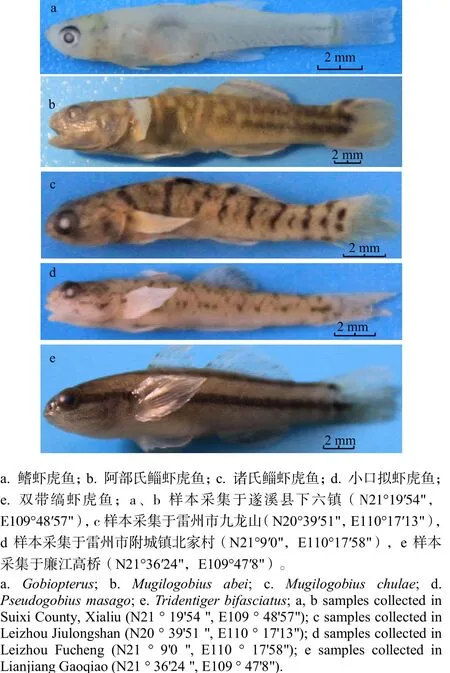
图1 5种虾虎鱼类Fig.1 Five species of Gobiidae
将获得的COI序列与NCBI及BOLD中的数据进行比对,相似度在98%至100%之间,形态鉴定至种的4种虾虎鱼与分子鉴定结果一致,而鳍虾虎鱼属的样本a经条形码鉴定,以及与NCBI 及BOLD数据库结果比对,与Gobiopterus lacustris相似度分别为99%、99.05%,且该属仅有2个种,结合形态结果和分子数据可确定为湖栖鳍虾虎鱼(Gobiopterus lacustris)。对COI序列进行整理归纳,得到5种虾虎鱼的45条COI序列,上传至GenBank获取登录号KT923713-KT923757(表1)。
2.2线粒体COI基因片段序列分析
用于测序的5个物种45个样本经人工核查、对位排列以及剪切得序列长540 bp,编码氨基酸180个,与GenBank中数据进行Blast比较,相似度均大于98%,与BOLD数据库中比较相似度在98.44% ~100%。比较5种虾虎鱼的COI序列显示,在540个核苷酸序列中,未出现插入、缺失及终止密码子。变异位点184个,包括单一突变位点12个,简约信息位点172个。在碱基组成上,T、C、A、G平均含量分别为29.9%、28.4%、23.9%、17.8%。A+T含量(53.8%)高于C+G(46.2%)。这与其他鱼类mtDNA COI基因同源序列的碱基特点相似[13-15]。
2.3群体序列变异情况及遗传距离
分析单倍型数目、单倍型多样性、核苷酸多样度、平均核苷酸差异,结果表明,45个序列测得23个单倍型,单倍型比例为51.1%。5种虾虎鱼中双带缟虾虎鱼单倍型比例最高(80.0%),湖栖鳍虾虎鱼单倍型比例最低(18.2%)(表2)。与单倍型比例相似,双带缟虾虎鱼与小口拟虾虎鱼单倍型多样性较高,分别为0.900±0.161和0.917±0.092,而湖栖鳍虾虎鱼最低,为0.545±0.072。5种虾虎鱼的核苷酸多样度均较低,其中小口拟虾虎鱼最高(0.006 6),湖栖鳍虾虎鱼最低(0.001 0)。平均核苷酸差异仍是小口拟虾虎鱼最高(3.556),而湖栖鳍虾虎鱼依然最低(0.545)。

表2 5种虾虎鱼类线粒体 COI 基因序列分析的统计参数Table 2 Statistical parameters of mtDNA COI gene in the five species of Family Gobiidae
利用MEGA6.0计算5种虾虎鱼45条COI基因部分序列的Kimura 2-parameter模式最大似然法估计转换颠换值为1.58。并对5个虾虎鱼物种23个单倍型的45条序列进行遗传距离分析,结果显示,平均种间遗传距离为0.075 9~0.255 2,其中湖栖鳍虾虎鱼与双带缟虾虎鱼遗传距离最大(0.255 2),阿部氏鲻虾虎鱼与诸氏鲻虾虎鱼遗传距离最小(0.075 9)。平均种内遗传距离在0.001 0~0.006 7,其中湖栖鳍虾虎鱼种内遗传距离最小(0.001 0),小口拟虾虎鱼遗传距离最大(0.006 7)(表3)。转换/颠换数对遗传距离散点图(图2)表明,随遗传距离的增加,转换、颠换数均增大,颠换数增加速率大于转换数,具有较强的系统发育信号,可用于系统进化分析。

表3 5种虾虎鱼类线粒体COI基因序列平均遗传距离Table 3 Average genetic distance of mtDNA COI sequences among the five species of Family Gobiidae
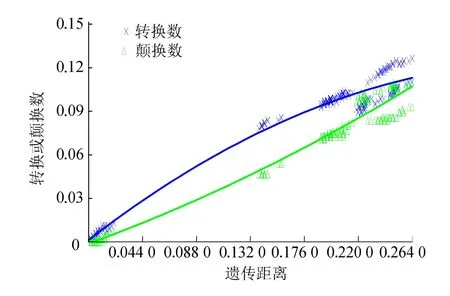
图2 5种虾虎鱼的45条COI基因碱基替换饱和分析Fig.2 Substitution saturation in the COI gene of the five species of Family Gobiidae
2.4分子系统进化
以COI基因部分序列为标记,用鲈形目的长棘银鲈(Gerres filamentosus)作外群,构建的NJ及ML系统进化树见图3。图3表明,基于COI基因部分序列构建的NJ与ML进化树拓扑结构基本保持一致,5种虾虎鱼聚为5个支系,与外群分开,同种虾虎鱼不同个体聚为一支。NJ树和ML树均显示阿部氏鲻虾虎鱼(MA)与诸氏鲻虾虎鱼(MC)亲缘关系较近,先聚为1支,再与小口拟虾虎鱼(PM)聚为1支,然后与双带缟虾虎鱼聚为1支,而与湖栖鳍虾虎鱼的亲缘关系最远。
2.5中性检验
检测5种虾虎鱼群体是否偏离中性进化,结果显示,双带缟虾虎鱼、小口拟虾虎鱼、阿部氏鲻虾虎鱼、诸氏鲻虾虎鱼的Tajima’s D值均为负(表4),表明它们存在大量的低频等位基因位点,推测可能是由负选择(Negative selection)、轻微分离的有害突变或群体扩张所引起。而湖栖鳍虾虎鱼的Tajima’s D值为正,表明该种群内由中等频率的等位基因占主导,可能是平衡选择的结果,或是种群大小在经历瓶颈时稀有等位基因丢失所致。但5种虾虎鱼类的检验结果均显著(P < 0.05),不符合中性进化假设。

图3 基于COI基因的部分序列构建的5种虾虎鱼类NJ(a)和ML(b)系统发生树Fig.3 Reconstruction of NJ(a)and ML(b)phylogenetic trees of the five species of Gobiidae Family based on mitochondrial COI gene fragments

表4 5种虾虎鱼类mtDNA COI基因序列的中性检验结果Table 4 Neutrality tests of the five species of Gobiidae Family
3 讨 论
3.1系统进化分析
mtDNA在遗传进化分析中起重要作用。据所受选择压力不同,mtDNA中各区域的进化速率不同。如进化速率较快的D-loop适于群体遗传学及近缘物种或种内群体分析[16],相对保守的COI、12S rRNA、16S rRNA等适于种属间或分类阶元较高的物种间探讨[17-18]。本研究以COI为标记获得5种虾虎鱼45条序列,从遗传距离来看,诸氏鲻虾虎鱼种内分化程度最高,而湖栖鳍虾虎鱼种内分化程度最低。诸氏鲻虾虎鱼与湖栖鳍虾虎鱼亲缘关系最远,与阿部氏鲻虾虎鱼亲缘关系最近。从分子进化树来看,诸氏鲻虾虎鱼与阿部氏鲻虾虎鱼进化关系最近,而湖栖鳍虾虎鱼位于系统发育树的基部位置,表明其在进化上较为原始或较早分化出来。依据《中国动物志》[1]记载,鲻虾虎鱼属的阿部氏鲻虾虎鱼与诸氏鲻虾虎鱼的胸鳍、腹鳍、尾鳍鳍条数较多,鳃部有假鳃,鳃耙短小,是较特化、较晚形成的物种。而湖栖鳍虾虎鱼胸鳍、腹鳍、尾鳍鳍条相对较少,下颌显著向上突出,长于上颌,鳃部无假鳃,具多个近祖特征,是较原始种[1]。由此看来,用COI基因序列标记分析的虾虎鱼进化结果与传统形态学分类研究一致,证明线粒体COI基因在虾虎鱼类系统进化分析研究上的可行性。
3.2物种地理分布问题
鲈形目是硬骨鱼类较大的一支,而虾虎鱼又是鲈形目鱼类中最大的类群。由于种类繁多,关于虾虎鱼的研究仅见局部水域研究和零星报道。国内比较系统的虾虎鱼类研究成果主要有伍汉霖等[7]的《中国动物志》。该文献记述了中国所产虾虎鱼类的9科5亚科106属307种(含1新种),描述了研究简史、形态特征、生态学特征、区系组成与地理分布、分类系统与演化和经济利用等。但由于编集委员会资料收集、样本采集等有限,难免存在疏漏。本研究采集的5种虾虎鱼中,个别种类在地理分布上存在争议。例如广泛存在于雷州半岛的湖栖鳍虾虎鱼在该文献中无记录,该文献指出产于中国的鳍虾虎鱼属仅大鳞鳍虾虎鱼,同时FishBase指明,湖栖鳍虾虎鱼仅在菲律宾吕宋岛的淡水湖泊里有记录,而Lui Smiguel G A等[10]调查菲律宾最大淡水湖Laguna de Bay资源时也指出湖栖鳍虾虎鱼为该区域特有种。本研究采集的湖栖鳍虾虎鱼样本表明鳍虾虎鱼属在中国不仅存在一个物种。另外,《中国动物志》指出鲻虾虎鱼属的诸氏鲻虾虎鱼仅分布于香港而不产于台湾。而Akihito记述该鱼产于台湾根据是的他人记录,而非亲自采集,持疑。笔者采集到诸氏鲻虾虎鱼11尾,表明该物种在中国雷州半岛也有分布。
贺亮同学帮助采样,张艳苹等在数据处理及论文写作给予指导,广东海洋大学海洋生物技术研究室全体成员在实验技术上给予帮助,谨致谢忱。
参考文献
[1]伍汉霖,钟俊生.中国动物志[M].北京:科学出版社,2008:1-951.
[2]韩东燕,薛莹,纪毓鹏,等.胶州湾六丝钝尾虾虎鱼的摄食生态特征[J].应用生态学报,2013,24(5):1446-1452.
[3]罗刚,庄平,章龙珍,等.长江口中华鲟幼鱼的食物组成及摄食行为[J].应用生态学报,2008,19(1):144-150.
[4]王义权,许群山,彭宣宪,等.通过 Cyt b基因同源序列比较评估厦门文昌鱼的分类学地位[J].动物学报,2004,50(2):202-208.
[5]DOIUCHI R,NAKABO T.Molecular phylogeny of the stromateoid fishes(Teleostei:Perciformes)inferred from mitochondrial DNA sequences and compared with morphology-based hypotheses[J].Mol Phylogen Evolut,2006,39:111-123.
[6]成庆泰,郑葆珊.中国鱼类系统检索[M].北京:科学出版社,1987:1-1458.
[7]中国科学院动物研究所,中国科学院海洋研究所,上海水产学院.南海鱼类志[M].北京:科学出版社,1962:1-1184.
[8]伍汉霖,邵广昭,赖春福.拉汉世界鱼典[M].台湾:水产出版社,1999:1-1028.
[9]AQUINO L M G,TANGO J M,CANOY R J C,et al..DNA barcoding of fishes of Laguna de Bay,Philippines[J].Mitochondrial DNA,2011,22(4):143-153.
[10]孟玮,杨天燕,海萨,等.基于线粒体COI基因序列的亚东鲑DNA条形码研究[J].水产学杂志,2010,23(1):2010-2014.
[11]POSADA D,CRANDALL K A.Modeltest:testing the model of DNA substitution[J].Bioinformatics,1998,14(9):817-818.
[12]FELSENSTEIN J.Confidence limits on phylogenies:an approach using the bootstrap[J].Evolution,1985,39:783-791.
[13]彭士明,施兆鸿,侯俊利,等.银鲳3个野生群体线粒体COI 基因的序列差异分析[J].上海海洋大学学报,2009,18(4):398−402.
[14]张凤英,马凌波,施兆鸿,等.两种鲷属鱼类线粒体COI基因片段序列的比较[J].上海水产大学学报,2006,15(4):403−408.
[15]毕潇潇,高天翔,肖永双,等.4种鳕鱼线粒体16S rRNA、COI和Cyt b基因片段序列的比较研究[J].南方水产,2009,5(3):46−52.
[16]胡文革,段子渊,王金富,等.新疆3种雅罗鱼线粒体 DNA控制区序列的差异和系统进化关系[J].遗传学报,2004,31(9):970-975.
[17]APOSTOLIDISA A P,MAMURISB Z,TRIANTAPHYLLIDISA C.Phylogenetic relationships among four species of Mullidae(Perciformes)inferred from DNA sequences of mitochondrial cytochrome b and 16S rRNA genes[J].Biochemical Systematics and Ecology,2001,29(9):901–909.
[18]THACKER C E.Molecular phylogeny of the gobioid fishes(Teleostei:Perciformes:Gobioidei)[J].Molecular Phylogenetics & Evolution,2003,26(3):354–368.
(责任编辑:刘庆颖)
Sequence Variation and Molecular Phylogeny of Mitochondrial COI Gene Segments from Five Species of Gobiidae Family
LIAO Jian,ZHANG Shun,LONG Shui-sheng,HUANG Cheng-qin,GUO Yu-song,WANG Zhong-duo,LIU Chu-wu
(Key Laboratory of Aquaculture in South China Sea for Aquatic Economic Animal of Guangdong Higher Education Institutes,Fisheries College,Guangdong Ocean University,Zhanjiang 524025,China)
Abstract:45 mitochondrial COI gene sequences of five kinds of goby juveniles from Mangroves of Leizhou region were obtained,and the average content of T,C,A,G were 29.9%,28.4%,23.9%,17.8%,respectively.A total of 23 haplotypes were found with 184 variable nucleotide positions,and the haplotype proportion of Tridentiger bifasciatus was the highest(80.0%),and that of Gobiopterus lacustris was the lowest(18.2%).The inter- and intra-specific genetic distances(Kimura two-parameter,K2P)ranged from 0.137 6 to 0.263 5,and 0.0000 to 0.020 8,respectively.Based on Neighbor-Joining and Maximum Likelihood phylogenetic tree,among the gobies clustered into five branches,Mugilogobius abei and M.chulae are in the closest relationship,while the relationship between Gobiopterus lacustris and the other four populations is a little far.The neutrality tests indicated that G.lacustris was dominated by moderate-frequency allelic loci,while there were a large number of low-frequency allelic loci within the other four populations.
通信作者:王中铎(1980-),男,副教授。email:aduofa@gmail.com
基金项目:国家自然科学基金项目(31201996);广东省高等学校优秀青年教师培养计划项目(Yq2013089);广东省高等学校优秀青年教师培养计划资助项目(Yq2013091)
收稿日期:2015-10-26
doi:10.3969/j.issn.1673-9159.2016.01.002
中图分类号:Q754
文献标志码:A
文章编号:1673-9159(2016)01-0007-06
第一作者:廖健(1990-),男,硕士研究生,从事南海经济鱼类发育生物学研究。email:liaojian05@outlook.com

