花发育干细胞信号转导及基因调控研究进展
李玉军,赵 燕,汤 宾,张学文
(湖南农业大学生物科学技术学院,长沙410128)
花发育干细胞信号转导及基因调控研究进展
李玉军,赵 燕,汤 宾,张学文*
(湖南农业大学生物科学技术学院,长沙410128)
摘 要:植物干细胞的调控是一个动态的、网络化的过程。近年来植物干细胞相关研究已经有了较大进展,不断有新的参与干细胞调控的基因和机理被发现。综述了近年来植物干细胞调控相关研究进展,重点针对信号转导途径和植物花器官发育干细胞调控网络进行了阐述,归纳出较清晰的基因调控路径和模型,便于认识植物花器官发育中干细胞的时空调控方式。
关键词:花发育;干细胞;信号通路;基因调控
植物干细胞又称为胚性细胞,是一类具有自我更新和分化能力的细胞。植物发育过程的重大事件都涉及对干细胞的调控,包括植物胚胎发育、根的生长、叶的起始、花的发育以及胚珠的形成。这些事件中对干细胞的调控方式大体是相同的,都需要在分化的组织中抑制干细胞相关基因的表达使其进入分化发育模式,而在确定的位置继续维持干细胞的活性。
花发育干细胞调控是一个复杂的网络式调控过程。细胞分裂素是干细胞重要的内源信号,而最终信号调控的关键是靶向WUSCHEL(WUS)基因[1]。WUS基因最早由Laux发现并确定其功能。WUS是特异性维持茎尖、花分生组织结构和功能完整性的一转录因子,保守性的抑制干细胞分化因子的表达,WUS通过调控干细胞的分裂维持干细胞数量的相对稳定[2,3]。目前对植物干细胞的调控研究都指向对WUS基因的相关调控研究上,在花发育过程中WUS更多的是作为负调控角色激活其他花发育参与因子,反馈抑制干细胞的活动维持花发育模式[4,5]。WUS的调控涉及多个方面,包括基因表达所需的所有过程:内源信号的产生、信号的传递和信号的转导、信号调节作用、转录过程、翻译过程以及其mRNA的降解(转录后调控)等,这一系列过程都将影响WUS的表达,形成对植物干细胞的时空调控网。但近年来诸多研究表明,与干细胞调控相关因子并非都作用于WUS维持干细胞数量的平衡及分裂模式[6],可能还存在多条并行的干细胞调控途径,WUS也并非唯一能使细胞维持干细胞特征的因子,这也说明植物干细胞的调控比预期的更加复杂。
1 CLV-WUS干细胞信号调控通路
1.1 胞外信号肽研究
体细胞胚胎发生的研究发现,胚细胞与周边细胞存在物理性隔离现象,即干细胞群外周包围着一层较厚的细胞壁与相邻细胞形成显著的界线[7]。花分生组织静止中心之上的中心区由三层细胞形成嵌套结构:L1层细胞参与防御机制的建立,L2层细胞涉及DNA修复和端粒的维护,L3层细胞负责维护分生组织内的离子平衡。其中CLV-WUS反馈调节机制就发生在这三层嵌套结构的细胞中[8]。CLV-WUS反馈调节环路是最早被发现的与WUS调控相关的信号通路,CLV-WUS为受体蛋白激酶信号通路[2,9],CLAVATA3(CLV3)是分泌型的多肽,分泌到质外后其C端经多种蛋白酶的剪切加工以及糖基化修饰,形成具有多对内二硫键的糖肽结合在细胞膜上作为胞外信号受体[10,11]。WUS信号传递通路中除了CLV3作为胞外信号受体外,与CLV3同属多肽配体家族的CLEs也具有同样的功能,多个CLEs基因的过表达可以在一定程度上修复clv3的突变表型[12,13]。CLEs偶联多种环境响应信号以及生理胁迫信号参与一系列植物生理反应信号途径[14]。在拟南芥花发育过程中有19个CLEs成员参与其发育过程,其中CLE4、CLE10、CLE17、CLE27参与花的发育过程,这些基因的缺失突变或多或少的导致分生组织干细胞调控的混乱,出现不同程度的花发育异常[15]。这说明CLEs在一定程度与CLV3存在功能冗余,即CLEs可能作为CLV3的补充性信号受体存在,以完善植物系统性修复机制,降低不可控因素对植物的危害,保证植物体自身正常的生长生殖发育的进行。此外,Engstrom研究揭示GARS家族转录因子HAIRY MERISTEM(HAM)参与CLV3和干细胞表达的空间位置信息的监控过程,Atham1,2,3导致CLV3和WUS表达部位的细胞层整体下移;有趣的是ant ail6中的CLV3和WUS的分布位置发生了互换,且ant/ail6、ham都表现分生组织分布向周边区域扩展现象,但HAM与ANT/AIL6是否存在交互作用目前尚不清楚[16]。Zhou最近的研究证实HAM与WUS存在互作关系,HAM和WUS交互作用于相同的靶基因推动下游调控事件的进行,促进干细胞的增值;两者重叠表达模式中显现的差异是多元干细胞微环境建立的基础,关于分生组织干细胞产生的调控机理还需要进一步的研究[17]。
1.2 信号跨膜转导复合物研究
参与CLV-WUS通路跨膜信号转导中间体大多以复合物形式存在。目前已揭示的参与这一过程的复合物有5类:CLV1复合体由具有跨膜结构的受体蛋白激酶CLV1和CLE2配体形成[18,19];CLV2信号传导复合物由CLV2和CORYNE/(CRN/SOL2)组成,其中CRN/SOL2CRN是不具备胞外结构的受体蛋白激酶[20]。CRN/SOL2复合物作为CLV2胞内结构域的补充协同CLV2完成信号转导以及与其他信号传递复合物互作实现信息共享,crn/sol2出现与clv相似的突变表型,花分生组织WUS的表达范围扩大,雌蕊群异常扩张,形成更多的雄蕊和花瓣,crn clv1出现明显的表现累加效应,sol2在拟南芥花的第4轮长出更多的心皮,同时Miwa的研究表明,CRN/SOL2复合体还参与CLEs的信号转导过程[21,22]。信号由胞内到核内的传递过程涉及两个途径:一条是CLV1和CLV2复合物自磷酸化激活蛋白激酶MAPKs引起级联反应,最终将信号传递给核内靶基因WUS[23];另一条传递路径涉及下游的一个中间信号传递复合体POLTERGEIST(POL1/PLL1),POL1/PLL1编码蛋白磷酸酶,POL1/PLL1在体外磷脂PI(4)P诱导下结合到质膜上与CLV1、CLV2复合物进行互作,通过对目标蛋白的酰基化和烷基化修饰作用完成信号的靶向传递,POL1/ PLL1的过表达导致分生组织干细胞的累积[24~26]。Song研究发现,pol1/pll1可以抑制clv突变表型,Yu的进一步观察发现,pol1几乎完全抑制clv1的突变表型,而对clv2的突变性状只能起到部分抑制效果,这表明CLV1、CLV2共同作用于POL1/PLL1复合体参与的信号传导通路,同时也提示CLV2复合物可能参与独立于CLV1的未知信号传递通路,所以clv1、clv2突变表型弱于clv3[27,28]。
其他三个信号传递复合物是BARELY ANY MERISTEM(BAM)、RECEPTOR-LIKE2/TOADSTOOL2(RKP2/TOAD2)和ERECTA(ER)。Young等发现,bam1bam2bam3表现出与clv1类似的突变表型;进一步研究表明,BAM复合物与CLV3存在相互作用,同时BAM也参与CLEs的信号转导过程,说明BAM是独立于CLV1存在的信号转导复合物[29,30]。最新研究发现,BAM和CLV1之间表现的功能冗余实际上是因为在正常情况下CLV1抑制BAM的表达,BAM可能是CLV1表达调控机制的一环,保证CLV-WUS信号通路的相对稳定[31]。RKP2/TOAD2编码受体蛋白激酶。有研究发现,RKP2/TOAD2也是独立的参与CLV3的信号转导复合体之一,rpk2花分生组织突变表型弱于clv,rpk2-2在花的基部产生新的花序,rpk2-4通常会在心皮中形成更多的雌蕊,RKP2/TOAD2与CLV的双突变体性状显现出较clv更严重的干细胞累积和WUS表达部位的异位现象[32,33]。Urano等证实RKP2/TOAD2与异三聚体G蛋白共同组成信号模块将CLV3的信号传递到胞内,拟南芥中转录编码G蛋白的基因AGB1的缺失突变体agb1表现出与clv相似的干细胞分布区域扩大表型,agb1 clv2双突变体较clv2的心皮变短而数目增多[34]。BAM和RKP2/TOAD2都是广泛性的信号传递中间体,同时可以接收CLV3和CLEs信息,参与包括干细胞调控在内的诸多植物生理信号传导过程,BAM和RKP2/TOAD2可能在CLV-WUS干细胞调控信号跨膜转导过程中起补充作用[35]。ERECTA(ER)是富含亮氨酸的类受体蛋白激酶,ER具备实现信号由胞外到胞内传递功能的跨膜结构域和胞外结构域。目前的研究表明,ER作为第二条受体蛋白激酶信号通路参与花分生组织细胞中WUS表达的调控,间接影响花原基形成确定[36,37]。Uchida等研究证实,ER参与的信号调控影响了干细胞的分化,从而导致分生组织中的干细胞积累,CLV3参与干细胞的分裂过程,clv3因为干细胞分裂活动的失控而出现干细胞累积[38]。ER和CLV3可能参与对分生组织内细胞分裂素缓冲机制的建立,在多种信号作用下实现对干细胞数量的精确调控[39]。
细胞信号调控通路对干细胞调控是非细胞自主性的。正常花发育过程中干细胞信号调控通路(图1)首先是保证信号的有效传递,响应外界各种物理和生理性胁迫信号以及内源信号,然后将这些信号进行综合、解析、处理,传递到胞内使干细胞分裂分化,从而保证干细胞在时间和空间上处于最适宜状态,使植物各项发育活动正常进行。

图1 植物干细胞调控的信号转导Fig.1 The pathways of stem cell signal transduction in plant
2 干细胞调控网与花器官形成的时空性
2.1 以AP2为核心偶联细胞信号调控通路的干细胞调控网
大量的研究表明,分生组织与相应的发育器官的细胞命运,取决于发育细胞的空间位置信息而非遗传信息,对特定细胞命运的确立依赖对特定细胞群的一系列精确而有序的调控,从而保证器官发育的正确有序进行。在花发育初期,干细胞因WUS在中心区域的小范围内被激活,而CLV-WUS反馈调节环实现了干细胞在中心区域的数量和空间位置的稳态。在随后的花发育事件中对干细胞的空间、时间调控是花器官各原基正确起始的关键,参与花发育干细胞调控的相关因子形成了两个相对独立的调控网,花发育前中期形成了以APETALA 2(AP2)为核心偶联细胞信号调控通路的干细胞调控网。SQUINT(SQN)是细胞亲环素CYP40的同源基因,早期的研究认为SQN参与对AG的表达调控,Prunet等发现rbl-1 sqn-1 ult-1第四轮花器官中的AGAMOUS(AG)表达量下降[40]。最近的研究证实,SQN是microRNA172的上游调控基因,SQN的缺失突变体表型与miRNA172具有相似之处,都出现花序发育异常,还发现ap-2的表型弱于sqn,sqn-4 ag-6较ag-6产生更多的花器官,且花萼花瓣的相间出现。这表明SQN通过miRNA172影响了花同源转换基因的表达,间接影响了干细胞的活动[41]。巧合的是,sqn的诸多表型与表型较弱的clv相似,而在clv背景下SQN的缺失对clv的影响却很小,因此SQN可能参与对CLV的调控,而miRNA165/166-HD-Zip III-CLV是目前已证实涉及对CLV的调控通路,所以笔者推测SQN也可能影响microRNA165/166的表达。ARGONAUTE(AGO)编码EIF2C蛋白,研究发现AGO1和AGO10参与对miRNA165/166调控以及miRNA172对AP2的调控过程,其中AGO1作为miRNA的效应蛋白通过PIWI和PAZ结构域与HESO1(miRNA甲基化转移酶)相互作用,结合到miRNA的3′端,形成对miRNA甲基化保护性机制[42~44];Zhu等则证实AGO10通过与AGO1的竞争性结合作用解除对miRNA165/166的甲基化,并作为诱导剂维持miRNA165/166在分生组织干细胞中的表达,因此AGO10的缺失导致分生组织的发育受阻甚至消失,ago10-13表现出雌蕊原基的异位表达,心皮膨大变短,ag-10 ago10-13出现更为严重的花突变表型,花器官变少,心皮膨大更明显甚至导致异位心皮的暴露[45]。Williams等阐明了JABBA(JBA)、miRNA 166、HD-ZipⅢ型转录因子之间的调控路径,JBA在RNaseⅢ和RNA解旋酶的协同作用下导致miRNA 166的过表达从而使PHB、PHV、CAN的表达下调,造成jba-1D出现花分生组织簇生、花序异常、雌蕊发育不良等现象[46]。POWERDRESS (PWR)编码具有SANT结构域的蛋白,Yumul等发现PWR促进miRNA172和CRC的表达,导致干细胞累积和心皮发育异常。ag-10 pwr-1的雌蕊柄伸长,较ag-10心皮变短且内含异位心皮致使心皮的膨大,pwr-1和pwr-2的心皮顶端变宽;pwr-1、pwr-2、Col的相关基因的定量分析比较发现miRNA172和CRC的表达明显下调[47]。
Wu等证实miRNA172参与了对AP2的负调控过程,miRNA172的过表达导致第2、3轮花器官的形成出现混乱[48]。Wollmann等认为miRNA172通过监管AP2的表达使第2、3轮花器官形成过程中AP2和AG的活动处在相对平衡状态,以保证花瓣和雄蕊形态和位置的正确性,pAP3∶MIM172的部分雄蕊转换成花瓣,pAP3∶amiR-AP2出现异常形态的花瓣[49]。在其他多个物种的相关研究也证实了miRNA172对AP2的表达调控是保守性的,miRNA172的缺失都会导致花发育的异常[50]。Grigorova等和Sitaraman等在miRNA172与AP2之间发现一个由SEUSS/LEUNIG(SEU/LUG)以及SPOROCYTELESS(SPL)构成的反馈调节环,在第一轮花器官的形成过程中AP2招募SEU和LUG抑制miRNA172的表达,使AP2的表达上调为第二轮花器官的形成做前期准备[51,52]。在SEU和LUG存在的植物细胞瞬时表达体系中,Sridhar等发现AG的顺式作用元件驱动的报告基因可以响应APETALA1 (AP1)和SEPALLATA3(SEP3)介导的转录抑制活动,这表明AP1、SEP3是SEU和LUG的靶向DNA结合基因[53]。miRNA165的过表达导致HD-Zip III型转录因子(PHB、PHV、REV、ATHB-8,ATHB -15)的转录水平下调,并影响生长素合成以及生长素响应基因的表达,HD-ZipⅢ过表达植株心皮极性的建立出现异常,着生异位心皮,心皮内胚珠暴露,phb phv can还出现了多心皮现象[54,55]。Jia等利用短串联目标模拟技术(STTM)对miRNA165/166的研究发现,miRNA165/166通过HD-Zip III影响非生物胁迫信号途径,STTM165/166通过抑制miRNA165/166上调HD-ZipⅢ的转录水平,其拟南芥转基因植株因雄蕊变短而导致不育[56]。
PHABULOSA(PHB)、PHAVOLUTA(PHV)、CORONA(CNA)是相互之间存在功能冗余的HD-ZipⅢ型转录因子。该转录因子包含4个功能结构域,其中HD结构域参与对靶基因的表达调控。Lee认为PHB、PHV、CNA三者是通过非WUS调控途径对干细胞进行调控,但其机制目前尚不清楚[57]。也有资料表明HD-ZipⅢ型转录因子作用于生长素响应因子ARF影响分生组织区域内的生长素浓度和分布,进而参与干细胞的调控;Lee等推测PHB、PHV、CNA三者可能与CLV表达相关,HD-ZipⅢ型转录因子串联CLV-WUS信号通路间接参与花发育干细胞的调控[58];Landau等证实HD-ZipⅢ也可以协同ER参与花分生组织细胞的信号传导通路[59]。总之,HD-ZipⅢ型转录因子对干细胞的调控途径是多样性的。在HD-ZipⅢ的调控通路中同时还存在反馈调节机制,Brandt对rev-5、rev-6表达分析时发现AGO10表达受到抑制,phb phv rev分生组织中几乎没有AGO10的表达,而phb phv rev/+出现AGO10表达的上调,这说明HD-ZipⅢ型转录因子REVOLUTA(REV)与AGO10的表达调控相关,HD-ZipⅢ形成了对自身表达的反馈调节环[60]。JAIBA(JAB)是植物特异性HD-ZipⅡ型转录因子,参与植物的避荫反应和激素信号通路过程,对花的形态建成有重要影响。Turchi等发现这类转录因子在光途径介导下实现了对细胞内生长素的分布和浓度的调控,从而影响分生组织干细胞的分裂和分化,参与干细胞稳态模式的建立[61]。JAB还参与植物生殖器官的发育过程,其T-DNA插入突变体和反义抑制转基因植株都表现出不同程度的雌蕊、胚珠发育受阻、花药发育不良以及产生较少花粉粒[62]。Liu等证实A类基因AP2可通过调控生长素响应因子ARF3影响局部分生组织干细胞的活性,并抑制C类基因的表达,从而规范花萼花瓣原基的空间位置[63]。AP2的表达受抑制的突变体会出现花器中萼片原基被其它原基替代的现象[64]。
2.2 以AG为核心的干细胞调控网
拟南芥C类基因AG在花器官的形成过程中扮演着重要角色,花发育中后期形成了以AG为核心的干细胞调控网。ULTRAPETALA1(ULT1)编码具有SAND结构域的蛋白,是trxG复合物的主要组成亚基。早期的研究普遍认为ULT1与LEAFY(LFY)协同调控AG的表达,保证花发育事件在空间上的稳定性[65,66]。Engehorn等的研究从多方面的比较证实ULT1和LFY对AG的调控是相对独立的过程。对Col、lfy-6、ult1-1、ult1-1 lfy-6花中的AG表达水平的定量分析比较发现,lfy-6、ult1-1 lfy-6 中AG的表达水平明显下调,而AG在ult1-1和野生型拟南芥中的表达水平相当;同时还发现两者表达的时间和区域也具有差异,LFY是在第3花期的干细胞周边区域表达,而ULT1主要在第6花期的干细胞分布区域表达,LFY可能是在第3花期促进AG的转录水平的上调,ULT1则在第6花期通过解除PcG甲基化抑制作用提高AG的表达水平[67]。Bao等鉴定了2个BRI1-LIKE(BRL)T-DNA插入突变体发现了严重的花突变表型,进一步的研究发现,BRL是通过直接作用于AG启动子上的顺式作用元件促进AG的表达,同时还发现blr-4、blr-5在高温胁迫下导致突变表型加剧。这一发现说明AG的调控可能串联逆境响应途径[68]。Das等和Maier等发现,bZIP转录因子PERIANTHIA(PAN)通过调控AG的表达影响花发育干细胞的命运,pan导致花器官的数目变成5的基数,在短日照下pan显现出与ag相似的表型,而pan2 ag4未出现表型累加效应,因此Maier推测AG、PAN、WUS之间组成了一个负反馈调节环调控干细胞活动[69,70]。PAN通过限定AG表达的空间分布影响整个花器官的发育,perianthia-1的心皮发育出现异常,pan clv的花器官数目增多,seu pan双突变体导致AG在第1轮花器官的异位表达,出现花萼转换成花瓣现象[71]。
CRABS CLAW(CRC)是YABBY家族转录因子成员,在许多种子植物的相关研究中都证实CRC参与了心皮极性的建立和蜜腺的发育过程[72,73]。Mayo等发现CRC协同JAB参与花分生组织和雌蕊的发育过程[74]。Lee等在对LFY和MADSbox基因结合位点进行遗传表达分析时发现,LFY和MADS-box基因对CRC的表达具有重要作用,MADSbox基因通过其保守的结合位点调控CRC在蜜腺的表达[75]。KNUCKLES(KNU)、SUPERMAN(SUP)同属于C2H2锌指蛋白。锌指蛋白在拟南芥中是一个庞大的群体,它们主要参与植物形态建成和环境胁迫应答。C2H2锌指蛋白主要通过锌指结构结合DNA螺旋的大沟,进而对靶基因进行调控[76]。KNU和SUP的T-DNA插入突变体出现花器官异位表达,但二者在干细胞调控网中的地位有差异,KNU是AG的下游作用基因,而SUP与AG很可能处在同一调控层面,SUP在ag背景下的双突变体的花发育突变性状并未得到进一步的凸显[77,78]。Nibau等发现,SUP影响赤霉素在干细胞中的浓度和分布,这意味着SUP是通过独立于WUS的另一条未知途径对花发育干细胞进行调控[79]。Zhao等对黄瓜同源SUP的研究表明,SUP抑制雄蕊原基与心皮原基之间干细胞的活性,促进雄蕊形态建成和心皮边界的确定[80]。SUP的表达时空性分析发现,SUP在不同花期调控不同的基因群体,在花发育的早期SUP特异性参与雄蕊原基的形成,而花发育中期参与雌蕊原基细胞的分化工作[81]。
综上所述,对干细胞直接或间接作用的因子可归类为三个调控群体:直接作用因子、非编码RNA和对非编码RNA以及AG、AP2调控的基因群。这些调控因子形成了在调控方式、时间和对象上相对独立的两个调控网:以AP2为核心偶联细胞信号调控通路的干细胞调控网和以AG为核心的调控网。其中以AP2为核心偶联细胞信号调控通路调控网,偶联激素信号响应途径和环境胁迫响应途径(例如温度、光周期等),还可能涉及细胞非自主性调控机制,在花发育的前、中期形成对花器官发育的时空信息调控体系。以AG为核心的调控网则通过表观遗传调控机制和分子动力学调控机制,在花发育中、后期形成对花发育事件起作用的时空调控信息网。这两个干细胞调控网相互交织形成复杂的、精确的花发育事件的调控网络(图2)。
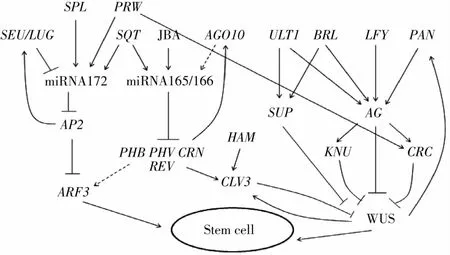
图2 花发育干细胞基因调控网络Fig.2 The gene regulation network of stem cell in floral development
3 H3K27me3甲基化调控机制与花发育干细胞的时空调控
在拟南芥生长发育过程中H3K27me3作用的靶基因大约有7000个。花发育干细胞调控过程中的表观遗传主要涉及组蛋白甲基化机制,其调控的路径大多与AG相关,目前涉及与花发育干细胞相关的甲基化/去甲基化调控路径有3条。其一是直接由AG通过招募PcG在H3K27me3作用下将WUS甲基化抑制WUS的表达,这条途径对WUS抑制作用是相对温和的,主要在花的4~6时期发挥作用[82]。这个过程涉及TOPOISOMERASE1a (TOP1a)Ⅰ型拓扑异构酶的协同作用,TOP1a可以释放DNA复制时的扭曲力保持核小体的稳定性以及使其密度变小,以利于PcG与靶基因DNA结合完成对靶基因的甲基化[83]。Dinh的研究揭示TOP1a可以通过DNA甲基化和H3K9me3沉默转座子活性来维持染色体的稳定性。Liu等最新的研究也表明,TOP1a通过改变核小体的分布来影响PcG介导的组蛋白修饰,而决定是否促进PCG与靶向基因的结合关键在于TOP1a的5′端一个功能区[84]。因此有学者推测TOP1a通过促进PcG介导的H3K27me2抑制WUS的表达,因此ag-10 top1a-2花分生组织异位表达以及top1a-1出现花序混乱现象。TOP1a也可以在DNA的复制阶段通过调控WUS的拷贝数来影响WUS后续的表达水平。第二条途径是AG调控KNU表达过程涉及的组蛋白甲基化机制。KNU最早是通过对EMS诱变突变体knu -1的基因定位而被发现,KNU编码C2H2锌指蛋白,对knu-1突变体的形态学观察发现其雄蕊、心皮出现异位表达现象和雌蕊柄伸长的现象。Sun将35S::KNU-AR转入ag-1形成的转基因植株中,发现其花分生组织中央的圆顶形干细胞团发育恢复正常,这证实了KNU是AG下游对WUS进行调控的基因[85]。然而AG-KNU的表达调控在时间上出现了延迟,GUS组织表达特异性分析表明,KNU在5 ~6花期开始表达,而AG的表达是在第3花期开始的[86]。针对这种现象以及基于Petruk发现在细胞分裂的DNA复制过程中DNA的甲基化会被忠实的复制保留现象,Sun提出了甲基化稀释原理,即已被甲基化的KNU需要AG通过与PRC2的组件ENDOSPERM 1(FIE)的竞争占据FIE在KNU启动区域的特异性结合位点PERs,从而抑制甲基化忠实保留机制,在经过大约2 d时间进行2次细胞分裂后,KNU才有足够多的未被甲基化的DNA拷贝启动自身表达,行使相关功能[87,88]。事实上AG的表达模式与KNU类似,AG-WUS反馈调节通路中AG的表达相对WUS的表达也是延迟的,延迟的时间也大致相同约2 d,WUS激活AG的表达涉及一个PcG抑制因子LFY参与,AG在花发育早期也可能处于甲基化抑制状态,LFY解除PcG对AG的甲基化抑制状态,激活AG的表达[89]。PcG复合物中的两个重要甲基转移酶基因CURLY LEAF(CLF)和SWINGER(SWN)的功能性缺失同样会影响干细胞的表观遗传调控体系[90,91]。
4 展望
花的发育涉及到一个复杂的调控网络。目前植物花的发育研究已深入到了表观遗传学,特别是涉及WUS表观遗传调控的研究工作已经有了较大进展,不断有新的参与干细胞调控的基因和调控机理被发现。其中朱健康揭示一个组蛋白乙酰化酶IDM1参与植物去甲基化作用机制的建立,这个发现对现有的干细胞甲基化调控体系造成了冲击[92]。WUS和HAM交互作用的证实和AINTEGUMENTA (ANT)和AINTEGUMENTA-LIKE(AIL)基因作用机理的进一步揭示,都需要对干细胞调控体系进行重新归纳总结;同时花发育过程中的某些突变性状仍然无法给出合理的解释,需要建立更为合理的调控体系给新出现的调控基因和调控方式预留位置。另一方面,对干细胞调控的量化也存在困难,WUS基因表达的mRNA降解过程的相关研究也十分缺乏,而WUS基因的表达量以及干细胞的数量的精确调控是实现花器官正确定位的保证,例如han出现分生组织的发育延迟,细胞分裂模式的异常,HANABA TANARU(HAN)通过监控干细胞的数量和位置信息参与不同花器官边界的确定,因此需要更多的研究才能对干细胞的数量和WUS基因的表达实现网络化的构建和量化描述[93,94]。植物的生殖发育是植物生活史上的重要事件,在种子植物发育中花器官发育在生殖发育过程中无疑占有尤为重要的地位。生殖器官发育的复杂性取决于花器官形成的精确性要求,这种精确性要求是对干细胞和WUS的位置信息、时钟信息、量化信息三者的协调统一性的调控。
本文梳理了近年来植物干细胞调控相关研究进展,构建了较为清晰的、多方位的植物花发育干细胞调控网络,旨在多方面的探讨、整合相关研究形成的关于植物花发育干细胞的时空调控模式,为今后的研究提供思路和框架性认识。
参考文献:
[1] Naseem M,Srivastava M,Dandekar T.Stem-cell-triggered immunity safeguards cytokinin enriched plant shoot apexes from pathogen infection[J].Frontiers in Plant Science,2014,5(5):1-5.
[2] Yadav RK,Perales M,Gruel J,et al.WUSCHEL protein movement mediates stem cell homeostasis in the Arabidopsis shoot apex[J].Genes&Development,2011,25 (19):2025-2030.
[3] Ikeda M,Mitsuda N,Ohme-Takagi M.Arabidopsis WUSCHEL is a bifunctional transcription factor that acts as a repressor in stem cell regulation and as an activator in floral patterning[J].The Plant Cell,2009,21(11):3493 -3505.
[4] Yadav RK,Perales M,Gruel J,et al.Plant stem cell maintenance involves direct transcriptional repression of differentiation program[J].Molecular Systems Biology,2013,9(1):1-13.
[5] Yadav RK,Tavakkoli M,Reddy GV.WUSCHEL mediates stem cell homeostasis by regulating stem cell number and patterns of cell division and differentiation of stem cell progenitors[J].Development,2010,137(21):3581-3589.
[6] Daum G,Medzihradszky A,Suzaki T,et al.A mechanistic framework for noncell autonomous stem cell induction in Arabidopsis[J].Proceedings of the National Academy of Sciences,2014,111(40):14619-14624.
[7] Fransz PF,Schel JHN.An ultrastructural study on the early development of Zea mays somatic embryos[J].Canadian journal of botany,1991,69(4):858-865.
[8] Yadav RK,Tavakkoli M,Xie M,et al.A high-resolution gene expression map of the Arabidopsis shoot meristem stem cell niche[J].Development,2014,141(13):2735 -2744.
[9] Durbak AR,Tax FE.CLAVATA signaling pathway receptors of Arabidopsis regulate cell proliferation in fruit organ formation as well as in meristems[J].Genetics,2011,189(1):177-194.
[10]Shinohara H,Matsubayashi Y.Arabinosylated glycopeptide hormones:new insights into CLAVATA3 structure [J].Current Opinion in Plant Biology,2010,13(5):515-519.
[11]Ohyama K,Shinohara H,Ogawa-Ohnishi M,et al.A glycopeptide regulating stem cell fate in Arabidopsis thaliana [J].Nature Chemical Biology,2009,5(8):578-580.
[12]Müller R,Borghi L,Kwiatkowska D,et al.Dynamic and compensatory responses of Arabidopsis shoot and floral meristems to CLV3 signaling[J].The Plant Cell,2006,18(5):1188-1198.
[13]Ni J,Clark SE.Evidence for functional conservation,sufficiency,and proteolytic processing of the CLAVATA3 CLE domain[J].Plant Physiology,2006,140(2):726 -733.
[14]Song XF,Guo P,Ren SC,et al.Antagonistic peptide technology for functional dissection of CLV3/ESR genes in Arabidopsis[J].Plant Physiology,2013,161(3):1076 -1085.
[15]Jun JH,Fiume E,Roeder AHK,et al.Comprehensive analysis of CLE polypeptide signaling gene expression and overexpression activity in Arabidopsis[J].Plant Physiology,2010,154(4):1721-1736.
[16]Engstrom EM.HAM proteins promote organ indeterminacy:but how[J].Plant Signaling&Behavior,2012,7 (2):227-234.
[17]Zhou Y,Liu X,Engstrom EM,et al.Control of plant stem cell function by conserved interacting transcriptional regulators[J].Nature,2015,517:377-380.
[18]Clark SE,Running MP,Meyerowitz EM.CLAVATA3 is aspecific regulator of shoot and floral meristem development affecting the same processes as CLAVATA1[J]. Development,1995,121(7):2057-2067.
[19]Lenhard M,Laux T.Stem cell homeostasis in the Arabidopsis shoot meristem is regulated by intercellular movement of CLAVATA3 and its sequestration by CLAVATA1 [J].Development,2003,130(14):3163-3173.
[20]Bleckmann A,Weidtkamp-Peters S,Seidel CAM,et al.Stem cell signaling in Arabidopsis requires CRN to localize CLV2 to the plasma membrane[J].Plant Physiology,2010,152(1):166-176.
[21]Zhu Y,Wang Y,Li R,et al.Analysis of interactions among the CLAVATA3 receptors reveals a direct interaction between CLAVATA2 and CORYNE in Arabidopsis [J].The Plant Journal,2010,61(2):223-233.
[22]Miwa H,Betsuyaku S,Iwamoto K,et al.The receptorlike kinase SOL2 mediates CLE signaling in Arabidopsis [J].Plant and Cell Physiology,2008,49(11):1752-1757.
[23]Betsuyaku S,Takahashi F,Kinoshita A,et al.Mitogenactivated protein kinase regulated by the CLAVATA receptors contributes to shoot apical meristem homeostasis [J].Plant and Cell Physiology,2011,52(1):14-29.
[24]Müller R,Bleckmann A,Simon R.The receptor kinase CORYNE of Arabidopsis transmits the stem cell-limiting signal CLAVATA3 independently of CLAVATA1 [J].The Plant Cell,2008,20(4):934-946.
[25]Gagne JM,Gish LA,Clark SE.The role of the acyl modification,palmitoylation,in Arabidopsis stem cell regulation[J].Plant signaling&Behavior,2010,5(8):1048 -1051.
[26]Song SK,Clark SE.POL and related phosphatases are dosage-sensitive regulators of meristem and organ development in Arabidopsis[J].Developmental Biology,2005,285(1):272-284.
[27]Gagne JM,Clark SE.The Arabidopsis stem cell factor POLTERGEIST is membrane localized and phospholipid stimulated[J].The Plant Cell,2010,22(3):729-743.
[28]Song SK,Lee MM,Clark SE.POL and PLL1 phosphatases are CLAVATA1 signaling intermediates required for Arabidopsis shoot and floral stem cells[J].Development,2006,133(23):4691-4698.
[29]DeYoung BJ,Bickle KL,Schrage KJ,et al.The CLAVATA1-related BAM1,BAM2 and BAM3 receptor kinase -like proteins are required for meristem function in Arabidopsis[J].The Plant Journal,2006,45(1):1-16.
[30]Shimizu N,Ishida T,Yamada M,et al.BAM 1 and RECEPTOR-LIKE PROTEIN KINASE 2 constitute a signaling pathway and modulate CLE peptide-triggered growth inhibition in Arabidopsis root[J].New Phytologist,2015,208(4):1104-1113.
[31]Nimchuk ZL,Zhou Y,Tarr PT,et al.Plant stem cell maintenance by transcriptional cross-regulation of related receptor kinases[J].Development,2015,142(6):1043 -1049.
[32]Kinoshita A,Betsuyaku S,Osakabe Y,et al.RPK2 is an essential receptor-like kinase that transmits the CLV3 signal in Arabidopsis[J].Development,2010,137(22):3911-3920.
[33]Perales M,Reddy GV.Stem cell maintenance in shoot apical meristems[J].Current Opinion in Plant Biology,2012,15(1):10-16.
[34]Urano D,Jones AM.Heterotrimeric G protein-coupled signaling in plants[J].Annual Review of Plant Biology,2014,65:365-384.
[35]Sawa S,Tabata R.RPK2 functions in diverged CLE signaling[J].Plant Signaling&Behavior,2011,6(1):86-88.
[36]Mandel T,Moreau F,Kutsher Y,et al.The ERECTA receptor kinase regulates Arabidopsis shoot apical meristem size,phyllotaxy and floral meristem identity[J].Development,2014,141(4):830-841.
[37]Meng X,Wang H,He Y,et al.A MAPK cascade downstream of ERECTA receptor-like protein kinase regulates Arabidopsis inflorescence architecture by promoting localized cell proliferation[J].The Plant Cell,2012,24 (12):4948-4960.
[38]Uchida N,Shimada M,Tasaka M.Modulation of the balance between stem cell proliferation and consumption by ERECTA-family genes[J].Plant Signaling&Behavior,2012,7(11):1506-1508.
[39]Uchida N,Shimada M,Tasaka M.ERECTA-family receptor kinases regulate stem cell homeostasis via buffering its cytokinin responsiveness in the shoot apical meristem[J].Plant and Cell Physiology,2013,54(3):343 -351.
[40]Prunet N,Morel P,Thierry AM,et al.REBELOTE,SQUINT,and ULTRAPETALA1 function redundantly in the temporal regulation of floral meristem termination in Arabidopsis thaliana[J].The Plant Cell,2008,20(4):901-919.
[41]Prunet N,Morel P,Champelovier P,et al.SQUINT promotes stem cell homeostasis and floral meristem termina-tion in Arabidopsis through APETALA2 and CLAVATA signalling[J].Journal of Experimental Botany,2015,66 (21):6905-6921.
[42]Ji L,Liu X,Yan J,et al.ARGONAUTE10 and ARGONAUTE1 regulate the termination of floral stem cells through two microRNAs in Arabidopsis[J].PLoS Genet,2011,7(3):e1001358.
[43]Shao C,Dong A,Ma X,et al.Is Argonaute 1 the only effective slicer of small RNA-mediated regulation of gene expression in plants[J].Journal of Experimental Botany,2014,65(22):6293-63102.
[44]Ren G,Xie M,Zhang S,et al.Methylation protects microRNAs from an AGO1-associated activity that uridylates 5′RNA fragments generated by AGO1 cleavage [J].Proceedings of the National Academy of Sciences,2014,111(17):6365-6370.
[45]Zhu H,Hu F,Wang R,et al.Arabidopsis Argonaute10 specifically sequesters miR166/165 to regulate shoot apical meristem development[J].Cell,2011,145(2):242 -256.
[46]Williams L,Grigg SP,Xie M,et al.Regulation of Arabidopsis shoot apical meristem and lateral organ formation by microRNA miR166g and its AtHD-ZIP target genes [J].Development,2005,132(16):3657-3668.
[47]Yumul RE,Kim YJ,Liu X,et al.POWERDRESS and diversified expression of the MIR172 gene family bolster the floral stem cell network[J].PLoS Genetics,2013,9 (1):e1003218.
[48]Wu G,Park MY,Conway SR,et al.The sequential action of miR156 and miR172 regulates developmental timing in Arabidopsis[J].Cell,2009,138(4):750-759.
[49]Wollmann H,Mica E,Todesco M,et al.On reconciling the interactions between APETALA2,miR172 and AGAMOUS with the ABC model of flower development[J]. Development,2010,137(21):3633-3642.
[50]Varkonyi-Gasic E,Lough RH,Moss SMA,et al.Kiwifruit floral gene APETALA2 is alternatively spliced and accumulates in aberrant indeterminate flowers in the absence of miR172[J].Plant Molecular Biology,2012,78(4-5):417-429.
[51]Grigorova B,Mara C,Hollender C,et al.LEUNIG and SEUSS co-repressors regulate miR172 expression in Arabidopsis flowers[J].Development,2011,138(12):2451-2456.
[52]Sitaraman J,Bui M,Liu Z.LEUNIG_HOMOLOG and LEUNIG perform partially redundant functions during Arabidopsis embryo and floral development[J].Plant Physiology,2008,147(2):672-681.
[53]Sridhar VV,Surendrarao A,Liu Z.APETALA1 and SEPALLATA3 interact with SEUSS to mediate transcription repression during flower development[J].Development,2006,133(16):3159-3166.
[54]Zhou GK,Kubo M,Zhong R,et al.Overexpression of miR165 affects apical meristem formation,organ polarity establishment and vascular development in Arabidopsis [J].Plant and Cell Physiology,2007,48(3):391-404.
[55]Zhong R,Ye ZH.Regulation of HD-ZIPⅢgenes by microRNA 165[J].Plant Signaling&Behavior,2007,2 (5):351-353.
[56]Jia X,Ding N,Fan W,et al.Functional plasticity of miR165/166 in plant development revealed by small tandem target mimic[J].Plant Science,2015,233:11-21.
[57]Lee C.A WUSCHEL-independent stem cell specification pathway is repressed by PHB,PHV and CNA in Arabidopsis[J].PloS One,2014,10(5):1-19.
[58]Lee C,Clark SE.Core pathways controlling shoot meristem maintenance[J].Developmental Biology,2013,2 (5):671-684.
[59]Landau U,Asis L,Williams LE.The ERECTA,CLAVATA and classⅢHD-ZIP pathways display synergistic interactions in regulating floral meristem activities[J]. PloS One,2015,10(5):1-11.
[60]Brandt R,Xie Y,Musielak T,et al.Control of stem cell homeostasis via interlocking microRNA and microProtein feedback loops[J].Mechanisms of Development,2013,130(1):25-33.
[61]Turchi L,Carabelli M,Ruzza V,et al.Arabidopsis HDZipⅡtranscription factors control apical embryo development and meristem function[J].Development,2013,140(10):2118-2129.
[62]Carabelli M,Turchi L,Ruzza V,et al.Homeodomain-Leucine ZipperⅡfamily of transcription factors to the limelight:central regulators of plant development[J]. Plant Signaling&Behavior,2013,8(9):e25447.
[63]Liu X,Dinh TT,Li D,et al.AUXIN RESPONSE FACTOR 3 integrates the functions of AGAMOUS and APETALA2 in floral meristem determinacy[J].The Plant Journal,2014,80(4):629-641.
[64]Byzova MV,Franken J,Aarts MG,et al.Arabidopsis STERILEAPETALA,a multifunctional gene regulating inflorescence,flower,and ovule development[J].Genes&Development,1999,13(8),1002-1014.
[65]Carles CC,Fletcher JC.The SAND domain protein ULTRAPETALA1 acts as a trithorax group factor to regulate cell fate in plants[J].Genes&Development,2009,23 (23):2723-2728.
[66]Winter CM,Austin RS,Blanvillain-Baufume S,et al.LEAFY target genes reveal floral regulatory logic,cis motifs,and a link to biotic stimulus response[J].Developmental Cell,2011,20(4):430-443.
[67]Engehorn J,Moreau F,Fletcher JC,et al.ULTRAPETALA1 and LEAFY pathways function independently in specifying identity and determinacy at the Arabidopsis floral meristem[J].Annals of Botany,2014,114(7):1497 -1505.
[68]Bao X,Franks RG,Levin JZ,et al.Repression of AGAMOUS by BELLRINGER in floral and inflorescence meristems[J].The Plant Cell,2004,16(6):1478-1489.
[69]Das P,Ito T,Wellmer F,et al.Floral stem cell termination involves the direct regulation of AGAMOUS by PERIANTHIA[J].Development,2009,136(10):1605 -1611.
[70]Maier AT,Stehling-Sun S,Wollmann H,et al.Dual roles of the bZIP transcription factor PERIANTHIA in the control of floral architecture and homeotic gene expression[J].Development,2009,136(10):1613-1620.
[71]Wynn AN,Seaman AA,Jones AL,et al.Novel functional roles for PERIANTHIA and SEUSS during floral organ identity specification,floral meristem termination,and gynoecial development[J].Frontiers in Plant Science,2014,434(5):1-13.
[72]Fourquin C,Primo A,Martínez-Fernández I,et al.The CRC orthologue from Pisumsativum shows conserved functions in carpel morphogenesis and vascular development[J].Annals of Botany,2014,114(7):1535-1544.
[73]Sun W,Huang W,Li Z,et al.Characterization of a Crabs Claw gene in basal eudicot species Epimedium sagittatum (Berberidaceae)[J].International Journal of Molecular Sciences,2013,14(1):1119-1131.
[74]Zúiga-Mayo VM,Marsch-Martínez N,de Folter S.The class II HD-ZIP JAIBA gene is involved in meristematic activity and important for gynoecium and fruit development in Arabidopsis[J].Plant Signaling&Behavior,2012,7(11):1501-1503.
[75]Lee JY,Baum SF,Alvarez J,et al.Activation of CRABS CLAW in the nectaries and carpels of Arabidopsis[J]. The Plant Cell,2005,17(1):25-36.
[76]Dinkins RD,Tavva VS,Palli SR,et al.Mutant and overexpression analysis of a C2H2 single zinc finger gene of Arabidopsis[J].Plant Molecular Biology Reporter,2012,30(1):99-110.
[77]Persikov AV,Wetzel JL,Rowland EF,et al.A systematic survey of the Cys2His2 zinc finger DNA-binding landscape[J].Nucleic Acids Research,2015,43(3):1965 -1984.
[78]Hiratsu K,Ohta M,Matsui K,et al.The SUPERMAN protein is an active repressor whose carboxy-terminal repression domain is required for the development of normal flowers[J].FEBS Letters,2002,514(2):351-354.
[79]Nibau C,Di Stilio VS,Wu H,et al.Arabidopsis and Tobacco SUPERMAN regulate hormone signalling and mediate cell proliferation and differentiation[J].Journal of Experimental Botany,2011,62(3):949-961.
[80]Zhao J,Liu M,Jiang L,et al.Cucumber SUPERMAN has conserved function in stamen and fruit development and a distinct role in floral patterning[J].PloS One,2014,9 (1):e86192.
[81]Airoldi CA.Determination of sexual organ development [J].Sexual Plant Reproduction,2010,23(1):53-62.
[82]Liu X,Kim YJ,Müller R,et al.AGAMOUS terminates floral stem cell maintenance in Arabidopsis by directly repressing WUSCHEL through recruitment of Polycomb Group proteins[J].The Plant Cell,2011,23(10):3654 -3670.
[83]Dinh TT,Gao L,Liu X,et al.DNA topoisomerase 1α promotes transcriptional silencing of transposable elements through DNA methylation and histone lysine 9 dimethylation in Arabidopsis[J].PloS Genetics,2014,11(9):e1005452.
[84]Liu X,Gao L,Dinh TT,S et al.DNA topoisomerase I affects polycomb group protein-mediated epigenetic regulation and plant development by altering nucleosome distribution in Arabidopsis[J].The Plant Cell,2014,26 (7):2803-2817.
[85]Payne T,Johnson SD,Koltunow AM.KNUCKLES(KNU)encodes a C2H2 zinc-finger protein that regulates development of basal pattern elements of the Arabidopsis gynoecium[J].Development,2004,131(15):3737-3749.
[86]Sun B,Xu Y,Ng KH,et al.A timing mechanism for stem cell maintenance and differentiation in the Arabidopsis floral meristem[J].Genes&Development,2009,23 (15):1791-1804.
[87]Sun B,Looi LS,Guo S,et al.Timing mechanism dependent on cell division is invoked by Polycomb eviction in plant stem cells[J].Science,2014,343:1248559.
[88]Petruk S,Sedkov Y,Johnston DM,et al.TrxG and PcG proteins but not methylated histones remain associated with DNA through replication[J].Cell,2012,150(5):922-933.
[89]Deng W,Buzas DM,Ying H,et al.Arabidopsis Polycomb Repressive Complex 2 binding sites contain putative GAGA factor binding motifs within coding regions of genes [J].BMC Genomics,2013,14(1):593.
[90]Chen N,Zhou WB,Wang YX,et al.Polycomb-group histone methyltransferase CLF is required for proper somatic recombination in Arabidopsis[J].Journal of Integrative Plant Biology,2014,56(6):550-558.
[91]Wu MF,Sang Y,Bezhani S,et al.SWI2/SNF2 chromatin remodeling ATPases overcome polycomb repression and control floral organ identity with the LEAFY and SEPALLATA3 transcription factors[J].Proceedings of the National Academy of Sciences,2012,109(9):3576-3581.
[92]Qian W,Miki D,Zhang H,et al.A histone acetyltransferase regulates active DNA demethylation in Arabidopsis [J].Science,2012,336:1445-1448.
[93]Zhao Y,Medrano L,Ohashi K,et al.HANABA TARANU is a GATA transcription factor that regulates shoot apical meristem and flower development in Arabidopsis[J].The Plant Cell,2004,16(10):2586-2600.
[94]Zhang X,Zhou Y,Ding L,et al.Transcription repressor HANABA TARANU controls flower development by integrating the actions of multiple hormones,floral organ specification genes,and GATA3 family genes in Arabidopsis[J].The Plant Cell,2013,25(1):83-101.
The Research Progress of Stem Cell Signal Transduction and Gene Regulation in Floral Development
LI Yujun,ZHAO Yan,TANG Bin,ZHANG Xuewen*
(College of Bio-science and Technology,Hunan Agricultural University,Changsha,Hunan 410128,China)
Abstract:Plant stem cell regulation is a dynamic process and in complex network.Plant stem cell research has been a great progress in recent years,new genes and mechanisms involved in stem cell regulation were found constantly.This paper summarized the research progress on plant stem cell regulation in recent years.It focuses on the signal transduction pathways and elaborates the plant stem cell regulation network in flower organ development.The gene regulation model is summarized for easy understanding of stem cells regulation.
Keywords:floral development;stem cell;signal transduction;gene regulation
基金项目:湖南省教育厅重点实验室项目(15K060)。
作者简介:李玉军(1990-),男,硕士研究生,Email:15974267467@163.com。*通信作者:张学文,教授,Email:xwzhang@hunau.edu.cn。
收稿日期:2015- 11- 24
文章编号:1001-5280(2016)02-0200-10
DOI:10.16848/j.cnki.issn.1001-5280.2016.02.23
中图分类号:Q945.4
文献标识码:A

