山羊酪氨酸相关蛋白1(TYRP1)基因密码子偏好性分析
李丽莎, 李祥龙,2*, 周荣艳, 李兰会
(1.河北农业大学, 河北 保定 071000; 2.河北科技师范学院, 河北 秦皇岛 066004)
山羊酪氨酸相关蛋白1(TYRP1)基因密码子偏好性分析
李丽莎1, 李祥龙1,2*, 周荣艳1, 李兰会1
(1.河北农业大学, 河北 保定 071000; 2.河北科技师范学院, 河北 秦皇岛 066004)
为TYRP1基因选择合适的表达系统及该基因编码蛋白的结构和功能的研究提供依据,运用CodonW软件和Usage Codon在线程序分析山羊酪氨酸相关蛋白1基因密码子偏性,比较不同物种TYRP1基因密码子使用偏性,并比较基于同义密码子相对使用度形成的聚类和基于编码区构建的系统发育结果。结果表明:山羊偏好使用的密码子有27个,以A/T结尾密码子的同义密码子相对使用度值明显偏高。山羊与藏羚羊、黑猩猩、家猫、家牛、家犬、家兔、绵羊、双峰驼、羊驼和野猪在密码子使用上相似,与家马不同。基于同义密码子相对使用度的聚类与基于编码区构建的系统发育结果不同,但TYRP1基因编码区形成的亲缘关系更可靠。
山羊; 酪氨酸相关蛋白1; 密码子偏好性
在动物中,黑色素是最普遍的色素,同时也是哺乳动物唯一能够合成的色素[1]。动物的黑色素主要分为两大类:真黑色素(黑色、棕色或蓝灰色)和褐黑色素(微红色、黄色或褐色)[2-3]。黑素生成通路的关键酶由酪氨酸酶基因家族编码,家族成员分别是酪氨酸酶(TYR)、酪氨酸相关蛋白1(TYRP1)和酪氨酸相关蛋白2(TYRP2)也称为多巴色素异构酶(DCT)[4]。TYRP1基因是第一个克隆成功的色素基因,随后在许多物种中也被成功克隆。TYRP1基因在黑色素合成过程中起调控作用,并对黑色素细胞的发育、生存与功能有调节作用[5-7]。同时,该基因对稳定酪氨酸酶活性具有重要意义,并能直接影响黑色素的生成[8]。毛色是山羊绒/毛/羔皮及裘皮的重要质量性状[9],影响山羊毛色候选基因一直都受到广泛关注,TYRP1基因在真黑色素形成过程中的关键作用已成为研究的热点。
mRNA是蛋白质合成的模板,mRNA与蛋白质之间的联系是通过三联密码的破译实现。已知61种密码子共编码20种氨基酸,每种氨基酸至少由1种密码子进行编码,最多可由6种密码子进行编码,这样对应同一氨基酸的密码子称为同义密码子(synonymous codon)。在编码蛋白质的过程中,同义密码子的使用概率不同[9-10],将基因优先使用的密码子称为最优密码子(optimal codon),此现象称为密码子偏性[11]。大量研究发现,不同物种的基因对密码子的使用具有明显的偏性[12],并且不同功能的基因的密码子偏性也存在较大的差异[13]。产生这种现象的因素有很多,如基因的表达水平[14-15]、翻译起始效应[16-17]、碱基的组分[18]、某些二核苷酸出现的频率[19-20]、GC含量[21-22]、基因长度[23]、tRNA的丰度[24-25]、蛋白质的结构[26]、蛋白质疏水水平[27]及密码子-反密码子间结合能的大小[28]等。对密码子偏性的研究为揭示有关物种间或某一物种的基因家族间的基因进化规律,了解转录和翻译进程中的调控机制,预测外源基因的最适宿主及通过改良外源基因以提高其表达水平等提供理论依据[29]。研究表明,利用聚类分析方法[30]研究不同基因在密码子偏性上的相似性具有可行性。笔者通过CodonW软件和Usage Codon在线程序分析山羊TYRP1基因的密码子偏好性,并与不同物种TYRP1基因密码子偏好性进行比较,为TYRP1基因选择合适的表达系统及该基因编码蛋白的结构和功能的研究提供依据。
1 材料与方法
1.1 目的基因的获取
山羊TYRP1基因的核苷酸序列全长1 614 bp,编码538个氨基酸。藏羚羊、黑猩猩和家马等物种的TYRP1基因完整编码区核苷酸序列数据均来源于NCBI上的(http://www.ncbi.nlm.nih.gov/)Genbank数据库(表1)。共计12个物种12条TYRP1基因编码区核苷酸序列。
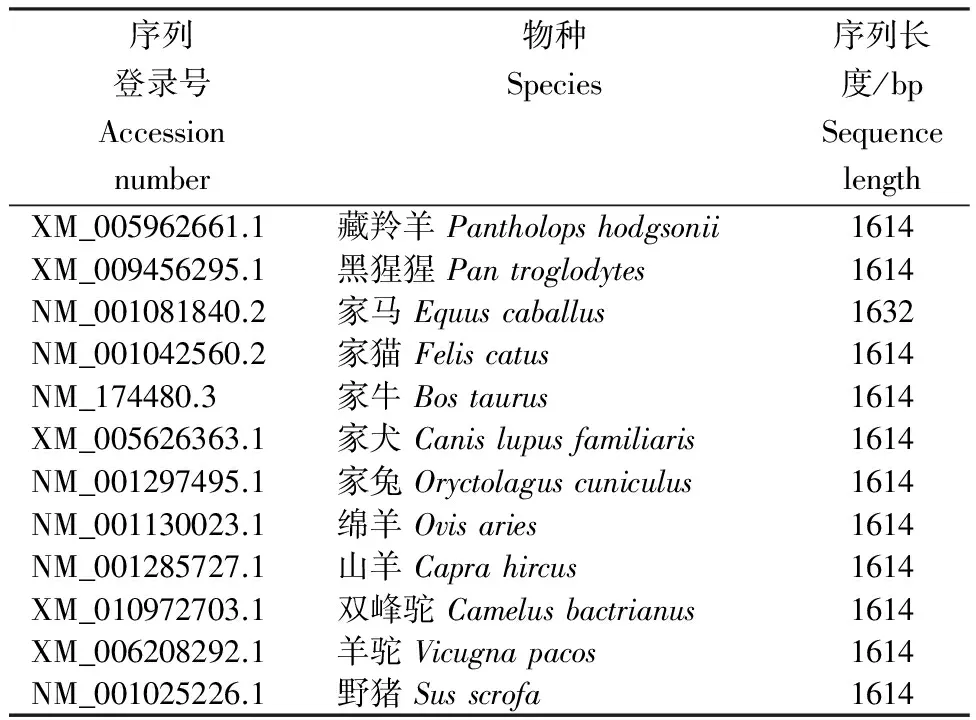
表1 TYRP1基因的完整编码区来源
1.2 RSCU 的计算
同义密码子相对使用度(Relative synonymous codon usage,RSCU)是指某一特定的密码子在编码对应氨基酸的同义密码子中的相对概率。氨基酸的使用和密码子的丰度不影响该值,RSCU值能直观地反映出密码子使用偏好性的程度[30]。当RSCU=1时,表明密码子使用无偏好性;当RSCU>1时,表明该密码子的使用频率较高。根据值大小判断偏好程度,反正亦然。Fraction表示各个密码子在编码该氨基酸的密码子中所占的比例,编码各氨基酸的密码子各比例之和为1。Frequency表示该密码子在编码基因总密码子中出现的频率,即在1000个密码子中出现的次数。利用Usage Codon在线程序(http://www.geneinfinity.org/sms/sms_codonusage.html)分析山羊TYRP1基因的Fraction和Frequency值,利用CodonW软件分析不同物种的RSCU值。
1.3 ENc、GC含量、 GC3s、FOP和CBI 的计算
有效密码子数(Effective number of codons,ENc)是一个能反映同义密码子非均衡使用的偏好程度的量化值[31],该值的范围为20~61,越靠近20表明偏好性越强。该值同时能确定内源基因表达量的相对高低,ENc值越大,对应的内源基因表达量越低,反之亦然。GC含量和密码子第三位的GC含量(GC3s)可以反映出碱基组成是否表现出明显的偏倚性,以0.5为一个界限,当二者均大于0.5时,表明该基因倾向于G、C碱基的使用。最优密码子使用频率(Frequency of optimal codons,FOP)可以确定基因密码子的最佳密码子,为蛋白的高表达改造提供依据,该值范围是0~1。密码子偏爱指数(Condon bias index,CBI)[32]反映一个具体基因中高表达优越密码子的组分情况:当CBI=1时,表明所有密码子均为偏向使用;当CBI=0时,表明所有密码子完全随机使用;CBI也可为负值。利用CodonW软件计算不同物种的ENc、GC、GC3s、FOP和CBI值。
1.4TYRP1基因的聚类分析方法
利用SPSS19.0软件对不同动物TYRP1基因密码子使用偏性的欧式平方距离系数进行聚类分析,在聚类的过程中,以每个物种的TYRP1基因作为一个对象,不考虑色氨酸、甲硫氨酸和3个终止密码子,剩余59个密码子的相对使用度作为变量,不同物种基因间的距离规定为同义密码子相对使用度的欧式平方距离[32]。计算2个基因间密码子使用偏性的欧式距离系数公式如下:
利用Clustal W 2.1软件对不同物种TYRP1基因编码区编码核苷酸序列进行序列比对,再由MEGA[34]软件按邻接法[35]构建系统发育树,该系统发育树的可靠性经1000次重复的自举检验法检验。
2 结果与分析
2.1 有效密码子数的 (ENc)、GC、CG3s、FOP和CBI
经软件计算,山羊TYRP1基因的ENc值、GC值、CG3s值、FOP值和CBI值分别为52.71、0.485、0.467、0.444、0.040。可见ENc值偏大,表明山羊TYRP1基因在编码氨基酸时出现的频率较为一致;山羊TYRP1基因CDS区GC含量较低,其GC3s值也较低,表明山羊偏好使用以A、T结尾的密码子,而且在完整的CDS区中A+T含量大于G+C含量;FOP值和CBI值均略微偏低,二者再次表明山羊TYRP1基因中高表达优越密码子的使用偏低。
2.2 密码子的偏好性
由表2可知,在山羊TYRP1基因的密码子中,27个密码子的RSCU值大于1,其中RSCU值大于1.5的密码子共10个,以A、T结尾的有13个密码子,分别是GCT、GAT、GAA、TTT、GGA、ATT、CCA、CCT、AGA、TCA、TCT、ACA、ACT,且这些密码子的Fraction值也较大,为山羊TYRP1基因的偏好密码子,其终止密码子也使用了TAA;而其余以G、C结尾的密码子的RSCU值大部分低于A、T结尾的密码子的RSCU值。对RSCU值小于1的密码子分析发现,大多数是以G、C结尾,且这些密码子的Fraction值也较低。
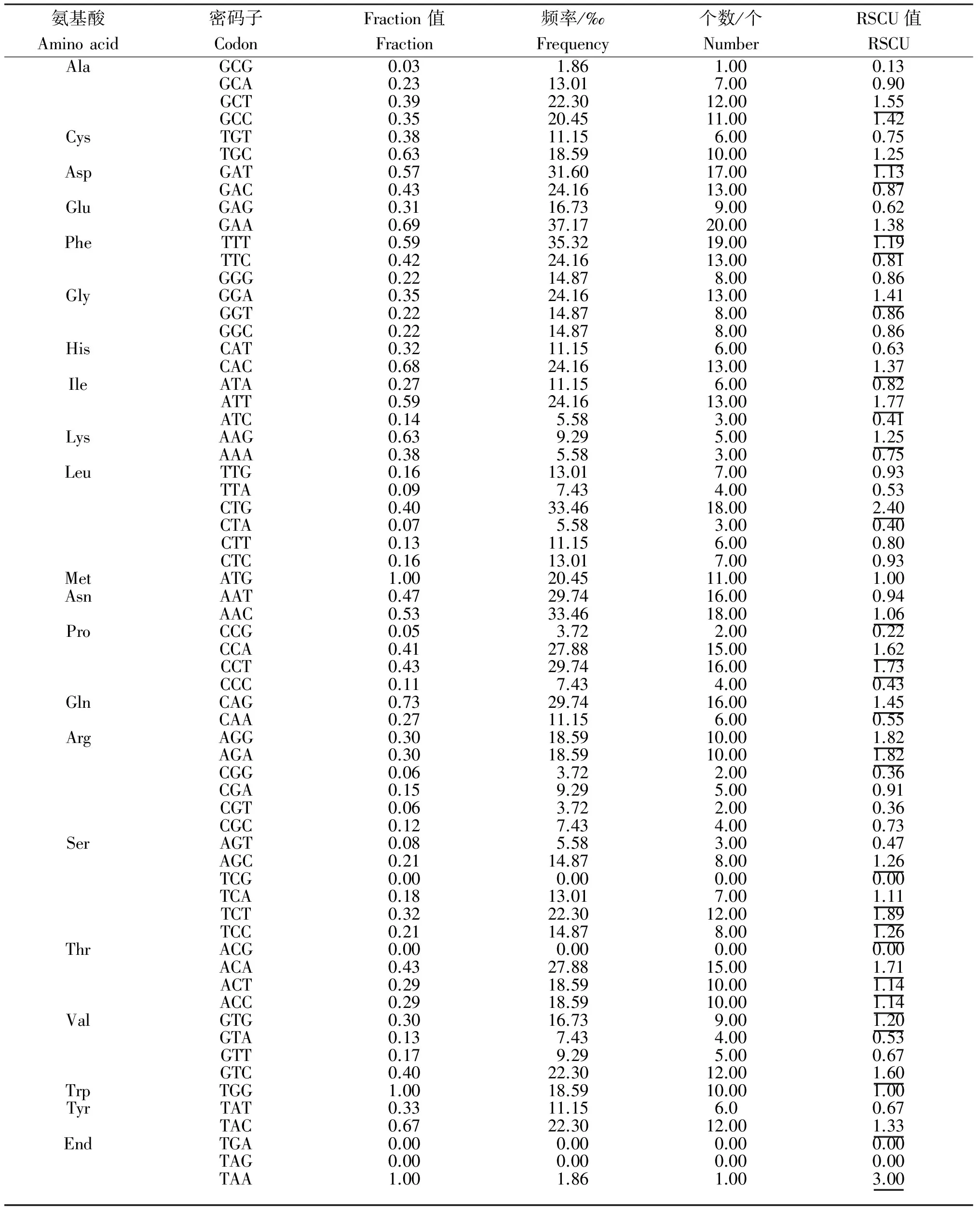
表2 山羊TYRP1 基因的密码子偏好性
注:大于1的RSCU值用下划线表示(下同)。
Note: The data with underline mean that the value of RSCU>1.The same below.
2.3 与其他动物TYRP1 基因密码子偏好性的比较
由表3可看出,藏羚羊、黑猩猩、家猫、家牛、家犬、家兔、绵羊、双峰驼、羊驼和野猪在密码子使用上与山羊相似,这些物种的ENc值均小于55;而家马的ENc值为55.10,大于55。表明山羊、藏羚羊和黑猩猩等11个物种的各密码子在编码氨基酸时出现的频率较为一致,且TYRP1基因表达水平一般,而家马的基因表达水平偏低。所有物种的GC含量和GC3s值均较为接近,且都低于0.5,提示这些物种TYRP1基因对A、T均有一定的偏好性,其中偶蹄目的GC含量和GC3s值大于其他物种。所有物种的FOP值均小于0.5。黑猩猩、家猫、家犬、家兔和野猪的CBI值均为负值,说明这几个物种的最佳密码子数目少于期望值。
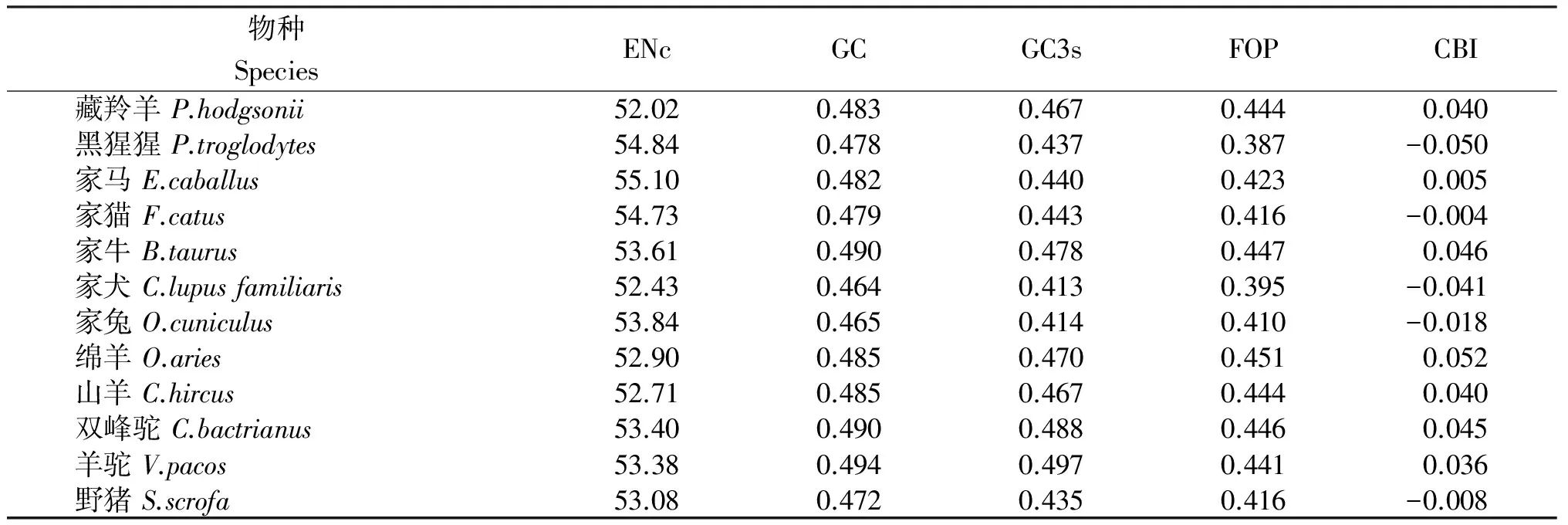
表3 不同物种TYRP1 基因的ENc、GC、GC3s、FOP 和CBI值
2.4TYRP1 基因的聚类
由表4、表5可知,家马和家猫与其他物种的距离差异不大,家犬、家兔分别和双峰驼、羊驼这2个偶蹄目动物的距离较远,同时黑猩猩和双峰驼之间的距离也较远,但同是偶蹄目的山羊、绵羊、藏羚羊和家牛之间的距离较近,揭示出基于各物种之间的欧式距离系数建立起来的聚类分析可能与动物学分类一致。
由图示可知,这些物种总共形成了2大类,一类全为偶蹄目动物的绵羊、山羊、藏羚羊、家牛、双驼峰和羊驼,说明这些物种对密码子的用法较为相似;而家犬、家兔、黑猩猩、家马、家猫和野猪聚成另一类,说明基于同义密码子相对使用度的聚类并没有反映出食肉目、奇蹄目、灵长目和兔形目这些哺乳动物的亲缘关系,比如说同为食肉目的家犬和家猫没有聚在一起,家犬和兔形目动物家兔首先聚在一起,这一聚类结果和基于各物种TYRP1基因的编码区形成的系统发育树(图示)不一致,树中分支位置的数值几乎全部大于75,表明置信度较高,因此由编码区构建的系统发育树更符合动物学分类,更接近这12个物种的真实分类系统,表明TYRP1基因编码区的差异大小更能反映物种间的亲缘关系。
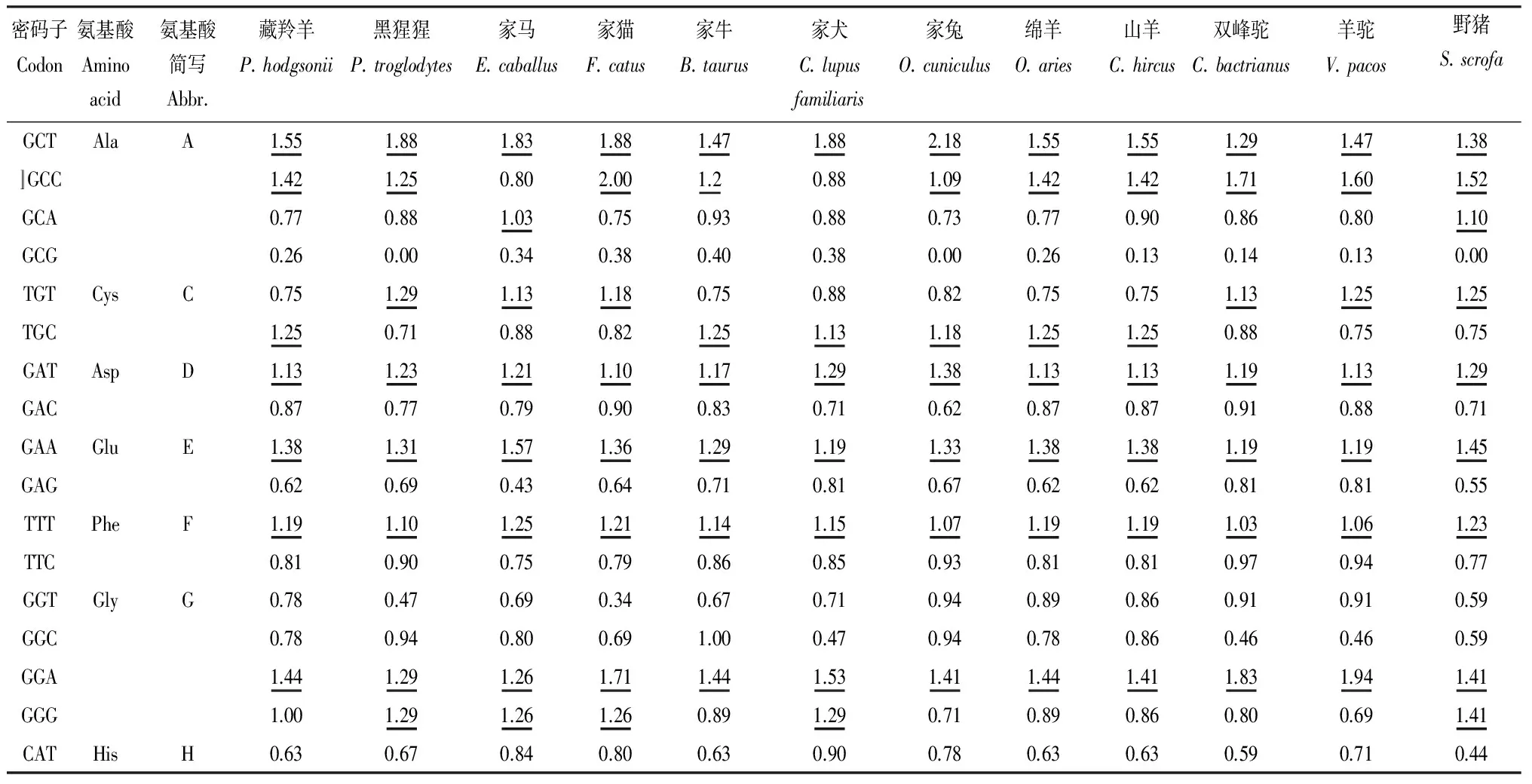
表4 不同物种TYRP1 基因的RSCU 值
续表4
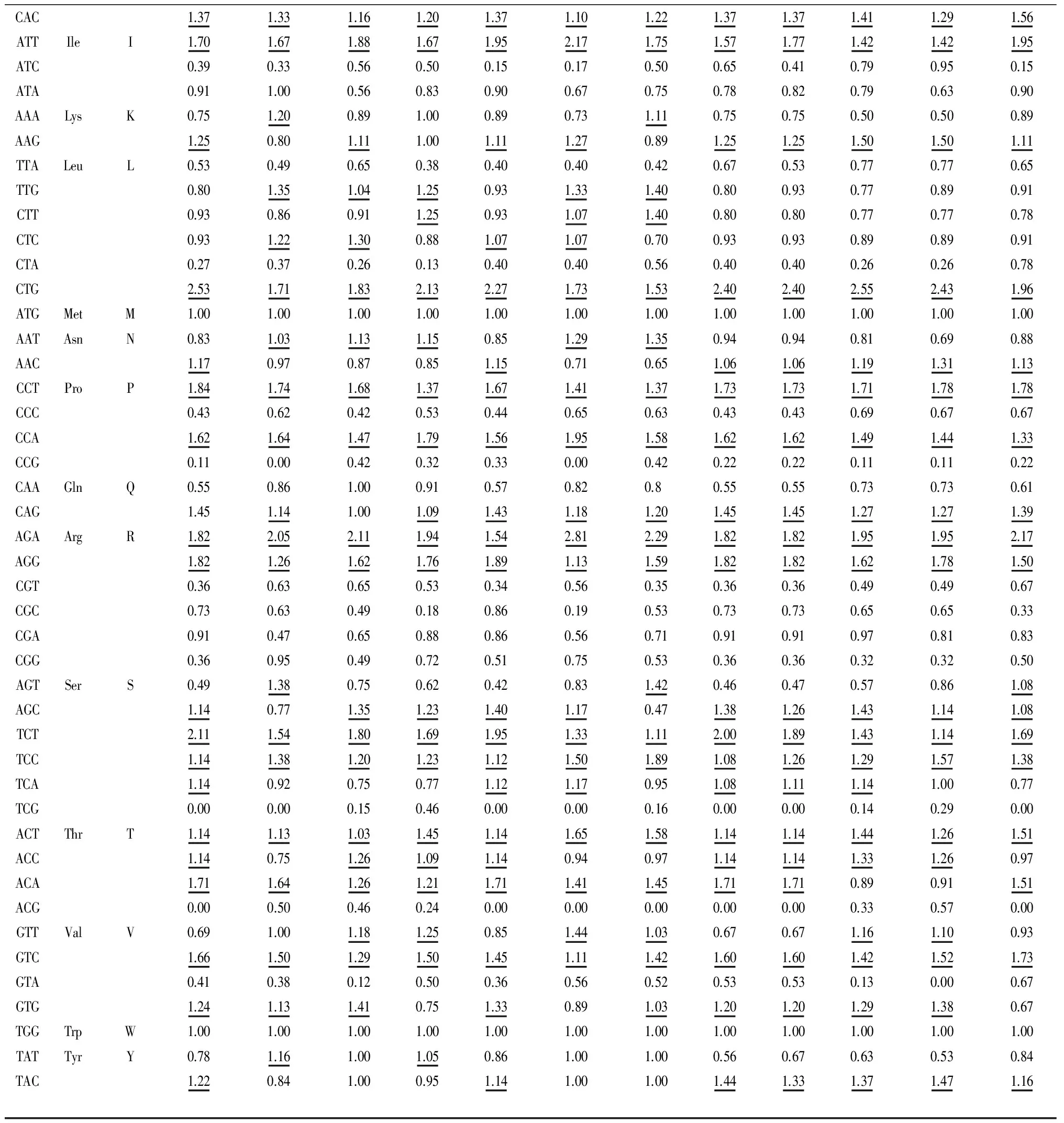
CAC1.371.331.161.201.371.101.221.371.371.411.291.56ATTIleI1.701.671.881.671.952.171.751.571.771.421.421.95ATC0.390.330.560.500.150.170.500.650.410.790.950.15ATA0.911.000.560.830.900.670.750.780.820.790.630.90AAALysK0.751.200.891.000.890.731.110.750.750.500.500.89AAG1.250.801.111.001.111.270.891.251.251.501.501.11TTALeuL0.530.490.650.380.400.400.420.670.530.770.770.65TTG0.801.351.041.250.931.331.400.800.930.770.890.91CTT0.930.860.911.250.931.071.400.800.800.770.770.78CTC0.931.221.300.881.071.070.700.930.930.890.890.91CTA0.270.370.260.130.400.400.560.400.400.260.260.78CTG2.531.711.832.132.271.731.532.402.402.552.431.96ATGMetM1.001.001.001.001.001.001.001.001.001.001.001.00AATAsnN0.831.031.131.150.851.291.350.940.940.810.690.88AAC1.170.970.870.851.150.710.651.061.061.191.311.13CCTProP1.841.741.681.371.671.411.371.731.731.711.781.78CCC0.430.620.420.530.440.650.630.430.430.690.670.67CCA1.621.641.471.791.561.951.581.621.621.491.441.33CCG0.110.000.420.320.330.000.420.220.220.110.110.22CAAGlnQ0.550.861.000.910.570.820.80.550.550.730.730.61CAG1.451.141.001.091.431.181.201.451.451.271.271.39AGAArgR1.822.052.111.941.542.812.291.821.821.951.952.17AGG1.821.261.621.761.891.131.591.821.821.621.781.50CGT0.360.630.650.530.340.560.350.360.360.490.490.67CGC0.730.630.490.180.860.190.530.730.730.650.650.33CGA0.910.470.650.880.860.560.710.910.910.970.810.83CGG0.360.950.490.720.510.750.530.360.360.320.320.50AGTSerS0.491.380.750.620.420.831.420.460.470.570.861.08AGC1.140.771.351.231.401.170.471.381.261.431.141.08TCT2.111.541.801.691.951.331.112.001.891.431.141.69TCC1.141.381.201.231.121.501.891.081.261.291.571.38TCA1.140.920.750.771.121.170.951.081.111.141.000.77TCG0.000.000.150.460.000.000.160.000.000.140.290.00ACTThrT1.141.131.031.451.141.651.581.141.141.441.261.51ACC1.140.751.261.091.140.940.971.141.141.331.260.97ACA1.711.641.261.211.711.411.451.711.710.890.911.51ACG0.000.500.460.240.000.000.000.000.000.330.570.00GTTValV0.691.001.181.250.851.441.030.670.671.161.100.93GTC1.661.501.291.501.451.111.421.601.601.421.521.73GTA0.410.380.120.500.360.560.520.530.530.130.000.67GTG1.241.131.410.751.330.891.031.201.201.291.380.67TGGTrpW1.001.001.001.001.001.001.001.001.001.001.001.00TATTyrY0.781.161.001.050.861.001.000.560.670.630.530.84TAC1.220.841.000.951.141.001.001.441.331.371.471.16
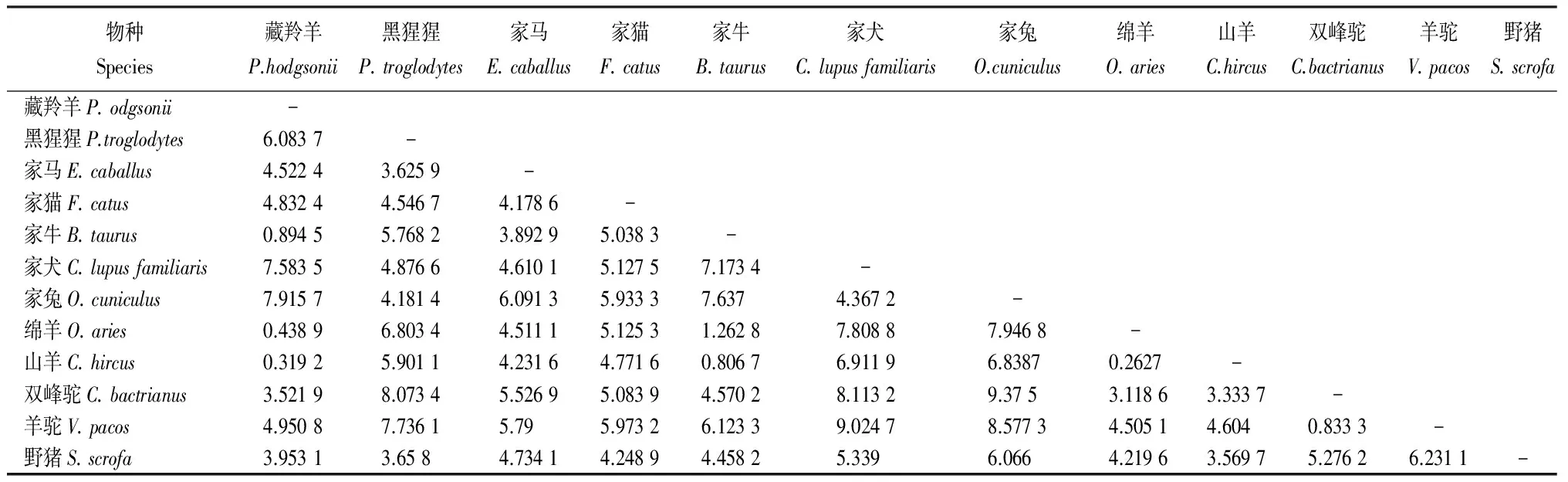
表5 不同物种TYRP1 基因间密码子使用偏性的欧式平方距离系数
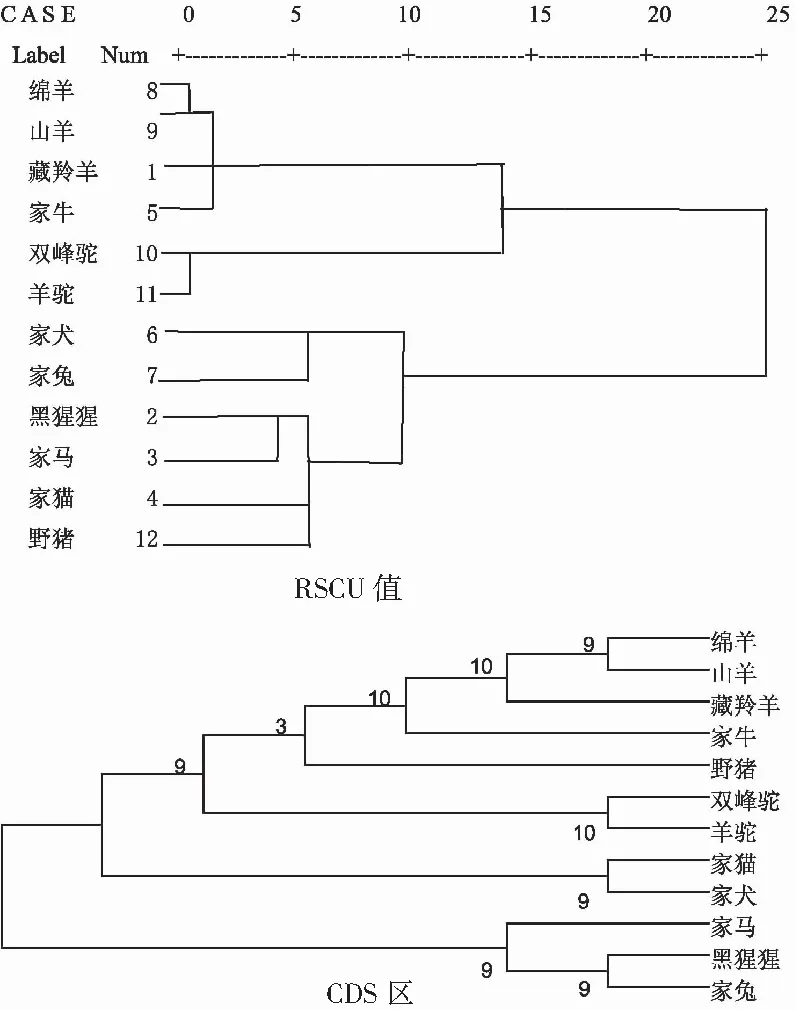
图示 基于TYRP1基因的RSCU值与CDS区的聚 类树状图
Fig. Cluster analysis ofTYRP1 gene coding region and RSCU
3 结论与讨论
通过分析山羊TYRP1基因的密码子使用特征发现,山羊偏好使用以A/T结尾的密码子,27个密码子存在使用偏性,其中使用频率最高的是TAA(3.00),也是该基因的终止密码子。关于终止密码子不用的终止密码子终止效率不同,其中以TAA的终止效率最高,其次是TAG,而TGA的终止效率最低[36]。Sun等[37]研究发现,终止密码子TAA主要出现于低复杂度的真核生物如酵母和非脊柱动物,TAG出现于真核生物的几率也很低,TGA主要出现于较复杂的真核生物中,尤其是脊柱动物。本研究发现,山羊TYRP1基因的终止密码子为TAA,这与Sun等研究结果不一致,这可能是由于终止密码子使用的偏好性因物种及基因的不同而有所差异。
本研究比较不同物种TYRP1基因密码子使用的偏好性时发现,山羊、藏羚羊、黑猩猩、家猫、家牛、家犬、家兔、绵羊、双峰驼、羊驼和野猪这11个物种的各密码子在编码氨基酸时出现的频率较为一致,且TYRP1基因表达水平一般,而家马的基因表达水平偏低。这可能是由于在基因类型及功能一定的条件下,物种间的差别对密码子使用偏性也存在差异。周童[13]等也发现,在进化过程中,亲缘关系较近的物种之间的同义密码子的使用具有较大的相似性。
将基于TYRP1基因同义密码子相对使用度的聚类结果和基于该基因编码区的系统发育结果比较发现,前者形成的聚类结果在亲缘关系相近的物种之间其密码子使用偏性较为接近,但在传统的动物性分类上相近的物种之间仍存在差异,如而家犬(食肉目)、家兔(兔形目)、黑猩猩(灵长目)、家马(奇蹄目)、家猫(食肉目)和野猪(偶蹄目)聚成一类。这与基于编码区序列形成的系统发育结果不一致,基于密码子偏好性的聚类并不能完全、真实地对应于系统发育关系[38],刘汉梅等[39],晁岳恩等[40],郭秀丽等[41]的研究中也出现过此种结果。这可能是由于在进化上,单基因可能存在较大的突变才造成其密码子使用偏性发生较大的变化,从而呈现出与真实的分类地位不同。TYRP1基因同义密码子使用偏性上的差异对该基因在生物体系中蛋白质的表达效率具有重要影响,提示今后在提高特定蛋白的表达效率时,可优先选择高表达的同义密码子,通过人工合成目的基因序列,这样能有效提高获得目标蛋白的水平。
对山羊及其他11个物种TYRP1基因对密码子使用的偏好性比较发现,山羊偏好使用以A/T结尾的密码子,在所有密码子中,RSCU值大于1.5的密码子共10个,以A、T结尾的有7个密码子,且终止密码子也使用了TAA。山羊与藏羚羊、黑猩猩、家猫、家牛、家犬、家兔、绵羊、双峰驼、羊驼和野猪在密码子使用上相似,与家马不同。基于同义密码子相对使用度的聚类与基于编码区构建的聚类不同,发现TYRP1基因编码区的差异大小更能反映物种间的亲缘关系。
[1] Krauss J,Geiger-Rudolph S,Koch I,et al.A dominant mutation in tyrp1A leads to melanophore death in zebrafish[J].Pigment Cell Melanoma Res,2014,27(5):827-830.
[2] Hearing V J. Mammalian monophenol monooxygenase (tyrosinase):purification,properties,and reactions catalyzed[J]. Methods Enzymol,1987,142:154-165.
[3] Prota G.Some new aspects of eumelanin chemistry[J].Prog Clin Biol Res,1988,256:101-124.
[4] Schmidt-K ntzel A,Eizirik E,O'Brien S J,et al.Tyrosinase and related protein 1 alleles specify domestic cat coat color phenotypes of the albino and brown loci[J].J Hered,2005,96(4):289-301.
[5] Hearing V J.Biochemical control of melanogenesis and melanosomal organization[J].The Journal of Investgative Dermatology Symposium Proceedings,1999,4(1):24-28.
[6] Jimenez-Cervantes C,Solano F,Kobayashi T,et al.A new enzymatic function in the melanogenic pathway.The 5,6-dihydroxyindole-2-carboxylic acid oxidase activity of tyrosinase-related protein-1(TRP1)[J].The Journal of Biological Chemistry,1994,269:17993-18000.
[7] Del Marmol V,Ito S,Jackson I,et al.TYRP-1 expression correlates with eumelanogenesis in human pigment cells in culture[J].Febs Letters,1993,327(3):307-310.
[8] 李春英.TYRP-1在正常黑素细胞及恶性黑素瘤细胞中的作用研究[D].西安:第四军医大学,2003.
[9] Ghosh T.Studies on codon usage in entamoeba histolytica[J].International Journal of Parasitology,2000,30:715-722.
[10] Karlin S,Mrazek J.What drives condon choices in human genes?[J].Journal of Molecular Biology,1996,262:459-472.
[11] 赵 翔,李 至,陆身枫,等.酵母Yarrowia lipolytica的密码子用法分析[J].复旦大学学报:自然科学版,1999(10):510-516.
[12] Chiapello H,Henaut A.Codon usage and gene function are related in sequences of arbidopsis thaliana[J].Gene,1998,209:GC1-GC38.
[13] 周 童,马建明,顾万君,等.哺乳动物MHC密码子使用概率的聚类分析[J].东南大学学报:自然科学版,2001,31(2):1-5.
[14] Duret L,Mouchiroud D.Expression pattern and, surprisingly, gene length shape codon usage in Caenorhabditis,Drosophila and Arabidopsis[J].Proc Natl Acad Sci USA,1999,96(8):4482-4487.
[15] Carlini D B,Chen Y,Stephan W.The relationship between third-codon position nucleotide content, codon bias,mRNA secondary structure and gene expression in the drosophild alcohol dehydrogenase genes Adh and Adhr[J]. Genetics, 2001, 159(2): 623-633.
[16] Sajau H,Washio T, Saito R,et al.Correlation between sequence conservation of the 5′untranslated region and codon usage bias in Mus musculus genes[J].Gene,2001,276(112):101-105.
[17] Stenstrm C M,Jin H N,Major L L,et al.Codon bias at the 3′-side of the initiation codon is correlated with translation initiation efficiency in Escherichia coli[J].Gene,2001,263(1/2):273-284.
[18] FedorovA,Saxonov S,Gilbert W.Regularities of context-dependent codon bias in eukaryotic genes[J].Nucleic Acids Res,2002,30(5):1192-1197.
[19] Ohno S.Universal rule for coding sequence construction:TA/GC deficiency-TG/CT excess[J].Proc Natl Acad Sci USA,1988,85(24):9630-9634.
[20] Karlin S,Campbell A M,Mrazek J.Comparative DNA analysis across diverse genomes[J]. Annu Rev Genetics,1998,32:185-225.
[21] 石秀凡,黄京飞,柳树群,等.人类基因同义密码子偏好的特征以及与基因GC含量的关系[J].生物化学与生物物理进展,2002,29(3):411-414.
[22] Rodriguez-Trelles F,Tarrio R,Ayala F J.Switch in codon bias and increased rates of Amino Acid substitution in the Drosophila saltans species group[J].Genetics,1999,153(1):339-350.
[23] Moriyama E N,Powell J R.Gene length and codon usage bias in Drosophila melanogaster, Saccharomyces cerevisiae and Escherichia coli[J].Nucleic Acids Res,1998,26(13):3188-3193.
[24] Moriyama E N,Powell J R.Codon usage bias and tRNA abundance in Drosophila[J].J Mol Evol,1997,45(5):514-523.
[25] Moszer I,Rocha E P C,Danchin A.Codon usage and lateral gene transfer in Bacillus subtilis[J]. Current Opinion in Microbiology,1999,2(5):524-528.
[26] 顾万君,马建民,周 童,等.不同结构的蛋白编码基因的密码子偏性研究[J].生物物理学报,2002,18(1):81-86.
[27] Romero H,Zavala A,Musto H.Codon Usage in Chlamydia Trachomatis is the Result of Strand-specific Mutational Biases and a Complex Pattern of Selective Forces[J].Nucleic Acids Research,2000,28:2084-2090.
[28] 石秀凡,黄京飞,梁宠荣,等.人类基因中同义密码子的偏好与密码子-反密码子间的结合强度密切相关吗?[J].科学通报,2000,45(23):2520-2525.
[29] 吴宪明,吴峰松,任大明,等.密码子偏性的分析方法及相关研究进展[J].遗传,2007,29(4):420-426.
[30] Sharp P,Mosurski K.Codon usage in yeast:cluster analysis clearly differentiates highly and lowly expressed genes[J].Nucleic Acids Research,1986,14:5125-5143.
[31] 刘庆坡,谭 军,薛庆中.籼稻品种93-11同义密码子的使用偏性[J].遗传学报,2003,30(4):335-340.
[32] Morton B R.Chloroplast DNA codon use: evidence for selection at the psb A locus based on tRNA availability[J]. J Mol Evol, 1993,37(3):273-280.
[33] Sau K,Gupta S K, Sau S,et al.Synonymous codon usage bias in 16 Staphylococcus aureus phages:Implication in phage therapy[J].Virus Res,2005,113(2):123-131.
[34] Tamura K,Dudley J,Nei M,et al.MEGA4:Molecular Evolutionary Genetics Analysis(MEGA)software version 4.0[J].Mol Biol Evol,2007,24:1596-1599.
[35] Satiou N,Nei M.The neighbor-joining method: a new method for reconstructing phylogenetic tree[J].Mol Biol Evol,1987,4:406-425.
[36] Tate W P,Mansell J B,Mannering S A,et al.UGA: a dual signal for ‘stop’ and for recoding in protein synthesis[J].Biochemistry (Mosc),1999,64(12):1342-1353.
[37] Sun J,Chen M,Xu J,et al.Relationships among stop codon usage bias, its context,isochores, and gene expression level in various eukaryotes[J].J Mol Evol,2005,61(4):437-444.
[38] Christianson M L.Codon usage patterns distort phylogenies from or of DNA sequences[J].Am J Bot,2005,92(8):1221-1233.
[39] 刘汉梅,赵 耀,顾 勇,等.几种植物waxy基因的密码子用法特性分析[J].核农学报,2010,24(3):476-481.
[40] 晁岳恩,吴政卿,杨会民,等.11种植物psbA基因的密码子偏好性及聚类分析[J].核农学报,2011,25(5):927-932.
[41] 郭秀丽,王 玉,杨路成,等.茶树CBF1基因密码子使用特性分析[J].遗传,2012,34(12):1614-1623.
(责任编辑: 刘忠丽)
Analysis of Codon Bias ofTYRP1 Gene inCaprahircus
LI Lisha1, LI Xianglong1.2*, ZHOU Rongyan1, LI Lanhui1
(1.AgricultureUniversityofHebei,Baoding,Hebei071000;2.HebeiNormalUniversityofScience&Technology,Qinhuangdao,Hebei066004,China)
As a link between the nucleic acid and protein, the usage of codon has important biological significance. The authors analyzed the codon usage bias ofTYRP1 gene inC.hircusby CodonW software and Codon Usage online program, compared the codon usage bias ofTYRP1 gene in different species, and compared the cluster based on the relative synonymous codon usage and the result of the phylogeny based on the coding region. The results showed that the preferences using codon ofC.hircusthad 27, and the value of the relative synonymous codon usage at the end of A and T were significantly higher. In codon usage,C.hircus,Pantholopshodgsonii,Pantroglodytes,Feliscatus,Bostaurus,Canislupusfamiliaris,Oryctolaguscuniculus,Ovisaries,Camelusbactrianus,VicugnapacosandSusscrofawere similar but different fromEquuscaballus. Clustering based on the relative synonymous codon usage was different from the results of phylogeny based on coding region, but the genetic relationship of theTYRP1 gene coding sequences was more reliable.
Caprahircus; tyrosinase-related protein 1; codon bias
2015-08-18; 2016-03-04修回
河北省高校创新团队领军人才培育计划(LJRC004);河北省应用基础研究计划重点基础研究项目“山羊彩色绒毛关键功能基因及调控元件克隆和功能研究”(15962901D);河北省科技计划项目(15226302D)
李丽莎(1989-),女,在读硕士,研究方向:动物遗传育种。E-mail:15131276262@163.com
*通讯作者:李祥龙(1963-),男,教授,博士,从事动物遗传育种研究。E-mail:lixianglongcn@yahoo.com
1001-3601(2016)03-0126-0113-07
S827; Q959.3
A

