CRISPR-Cas9技术发展史:25年的科学历程
郭晓强
深圳市第二人民医院,广东 深圳 518035
CRISPR-Cas9技术发展史:25年的科学历程
郭晓强†
深圳市第二人民医院,广东 深圳 518035
CRISPR-Cas是一种重要的原核生物获得性免疫系统。CRISPR序列可转录并加工为非编码RNA——crRNA,而Cas利用其DNA核酸外切酶完成RNA介导的靶DNA剪切,从而抵御噬菌体和质粒等DNA的入侵。在这一系统基础上改进并发明目前生命科学领域广泛应用的CRISPR-Cas9基因编辑技术。通过对CRISPR序列发现、结构命名、功能预测、实验证实、机制研究和系统改进等的描述,以期能对CRISPR-Cas9技术的诞生过程有一个全面的了解。
获得性免疫系统;原核生物;基因编辑;CRISPR-Cas9
1953年,DNA双螺旋模型的提出确立了DNA在分子生物学乃至整个生命科学的“中心”地位。由于DNA是遗传信息载体,因此对DNA进行精确操作(DNA编辑)进而调控生物学性状的研究,其重要性不言而喻。三位在DNA编辑技术——基因敲除方面做出奠定性贡献的科学家分享了2007年诺贝尔生理学或医学奖。然而,传统基因敲除技术尽管在阐明某些基因的功能方面发挥了重要作用,但操作过程较为繁琐,周期长,费时费力,亟需进一步完善。随后出现一系列DNA编辑技术,如ZFN(zinc fnger nuclease)和TALEN(transcription activator-like (TAL) effector nuclease)等,尤其是2012年出现的CRISPR-Cas9技术更是以操作简便、快速、高效而迅速成为实验室的必备技术之一,对推动生命科学的发展具有重要意义。此外,CRISPR-Cas9技术的发明也是一个具有传奇色彩的历程。
1 CRISPR序列的发现
这项技术发明在一定程度上可追溯到20世纪50年代开始的微生物遗传学、生物化学和基因组学,三位大师级科学家莱德伯格(Joshua Lederberg)、科恩伯格(Arthur Kornberg)和桑格(Frederick Sanger)为此做出了奠基性贡献。莱德伯格奠定细菌遗传学基础,从而使简单的细菌成为分子生物学模式生物[1];科恩伯格则奠定细菌分子生物学酶学基础,开创酶学研究科学范式[2];桑格则于1977年发明基因测序方法[3],从而使基因测序成为常规研究内容。CRISPR-Cas系统的发现则开始于细菌测序。
1987年,日本微生物学家石野良纯(Yoshizumi Ishino)在但中田(Atsuo Nakata)实验室对大肠杆菌的碱性磷酸酶同工酶(alkaline phosphatase isozyme, iap)进行测序,为更好地理解基因表达调节的机制,在对编码区进行检测的同时顺便对基因上游和下游序列完成了测序。在常规报道iap基因编码序列的同时,他们意外地发现终止密码子后的非编码区存在一些异常重复序列[4]。之所以异常源于两个原因:一方面原核生物如细菌的DNA利用率较高,因此它的重复序列较少(真核生物存在大量重复序列);另一方面传统的重复序列常为串联重复,而这次发现却是“重复-居间序列(spacer)-重复”这一排列特征(图1)。论文最后一句话是:“到目前为止,在其他原核生物未发现同源序列,并且这些序列的生物学意义尚不清晰。”[4]对这段重复序列的讨论说明他们也部分意识到这一现象,但就当时有限的知识而言,不可能对其有进一步理解。然而这一发现存在一个缺陷,就是尚不知这种现象是否具有普适性。如果仅仅是大肠杆菌iap基因特有,则重要性就大打折扣,因此科学界首先需要解决的是这种“诡异”的序列是否普遍存在。然而,后续研究进入一个缓慢发展期,很少有人对此现象进行研究(石野良纯等也转向翻译机制的研究)。
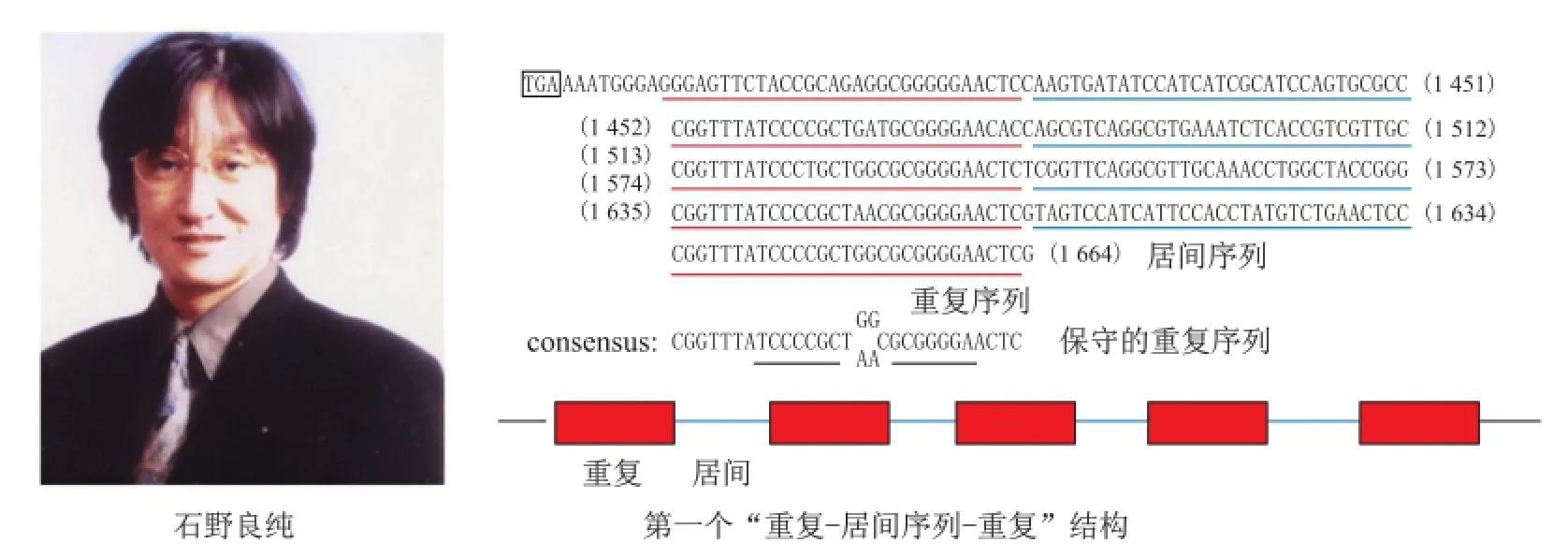
图1 CRISPR序列的发现
20世纪80年代末,西班牙阿利坎特大学(University of Alicante)的博士生莫伊察(Francisco Mojica)在一种嗜盐古菌(Haloferax mediterranei)中也发现一类“重复-居间序列-重复”特征序列[5]。莫伊察对这种现象非常感兴趣,因此进一步在其他微生物中寻找类似结构,基因组测序技术的突飞猛进为这项研究提供了极大便利。莫伊察通过对多种已完成基因组测序的原核生物进行序列比对分析发现这种现象非常普遍,到2000年,已在20多种不同微生物中发现这种特异序列。为便于进一步研究,将其命名为短规律间隔重复(short regularly spaced repeat, SRSR)[6]。
2002年,荷兰乌得勒支大学(Utrecht University)的詹森(Ruud Jansen)进一步发现多个微生物中存在这种特殊结构,并且不同物种的重复序列碱基数存在巨大差异,从21到37不等,如鼠伤寒沙门氏菌(Salmonella typhimurium)为21,而化脓性链球菌(Streptococcus pyogenes)为37。此外,他还发现这种序列只在原核生物存在,而病毒和真核生物均缺乏。为更好地规范相关研究,詹森在和莫伊察沟通后,将这种特殊结构重新定义为成簇规律性间隔短回文重复(clustered regularly interspaced short palindromic repeat, CRISPR)[7]。詹森还发现CRISPR序列附近还存在多个编码序列,推测它们参与了CRISPR的生理功能,因此将其命名为CRISPR相关基因(CRISPR-associated gene, Cas)(这种推测主要基于原核生物基因组多以操纵子形式存在,即功能相关基因串联分布在一起)。至此,在原核生物(包括细菌和古菌)中发现一个由特殊DNA序列(CRISPR)和多个编码基因(Cas)构成的独特系统,当然这个系统的作用尚一无所知。随后研究人员提出多种假说来解释这一系统,但大多基于一种想当然的推测,并未有太多的逻辑推理和数据支持。
2 CRISPR-Cas系统的生物学作用
2005年,CRISPR研究出现一个根本转折。来自西班牙和法国的三个研究小组几乎同时报道了一个重大发现:通过对CRISPR居间序列的系统分析,意外发现它们并非原核生物自身序列,而是来自病毒或质粒[8-10]。这一发现提出了一个重要问题:那就是原核生物获取这些序列的目的何在?
美国国立生物技术信息中心(National Center for Biotechnology Information, NCBI)进化生物学家库宁(Eugene Koonin)很早就对CRISPR-Cas系统拥有浓厚兴趣,但苦于无法理解它的生物学意义。当获悉CRISPR的居间序列来自病毒DNA后,库宁立刻意识到细菌可利用CRISPR作为一种防御病毒侵染的重要武器。在自然界,细菌时刻面临噬菌体(细菌病毒)等的攻击,但它们绝非被动受害者,而是在进化过程中形成多种防御措施,著名的如修饰-限制系统(对自身DNA碱基进行甲基化修饰,再利用限制性内切酶对入侵DNA进行剪切从而实现防御目的)。
早在2002年就发现原核生物中不编码蛋白质的CRISPR序列也可转录产生RNA[5],而1998年在真核生物中发现RNA干扰现象,即非编码的小RNA可影响mRNA稳定性或翻译效率。基于这些事实,库宁提出解释CRISPR-Cas作为获得性免疫系统的作用机制:细菌通过特定方式获取噬菌体DNA片段并将其整合到自身CRISPR重复序列之间形成居间序列,从而对外源入侵病毒产生“记忆”;这些序列可被转录出非编码RNA;当噬菌体再次感染时,这些RNA可依靠居间序列信息识别并破坏入侵者(图2)[11]。
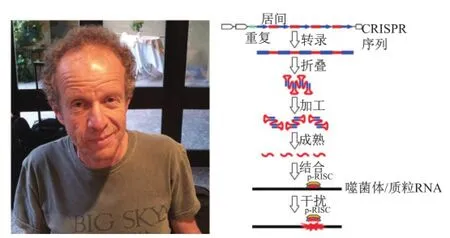
图2 CRISPR干扰假说的提出
在库宁提出这一假说时,科学界对CRISPR和Cas蛋白的作用还知之甚少,但这一思想激发了法国微生物学家巴兰古(Rodolphe Barrangou)的动力,他决定验证这一假说的可靠性。巴兰古之所以会验证这一假说,动力来源不仅仅在于假说的迷人魅力,更重要是出于工作需要。巴兰古在著名的酸奶公司丹尼斯克(Danisco)工作,时常面临的一大问题是产酸奶的嗜热链球菌(Streptococcus thermophilus)有时会爆发噬菌体感染而导致死亡,最终影响酸奶生产。库宁假说意味着可利用CRISPR-Cas系统来实现增强细菌抵抗噬菌体的目的。
巴兰古在霍瓦特(Philippe Horvath)等协助下首先利用两株噬菌体(P1和P2)侵染链球菌,结果杀死大部分细菌,但仍有部分“幸运”细菌保留下来,且当它们被进一步培养时获得噬菌体抗性。对这些抗性细菌的基因组分析表明,其CRISPR居间序列中出现噬菌体序列,并且与P1序列一致则对P1产生抗性,若与P2序列一致则对P2产生抗性,而如果为两株噬菌体公用序列,则对两株噬菌体均产生抗性(图3)。当将抗性细菌去除噬菌体序列则导致抗性消失,相反直接将噬菌体序列整合到未感染过噬菌体的细菌CRISPR中,细菌对首次噬菌体感染产生抗性[12]。这是首次在实验上证实CRISPR-Cas是一种细菌获得性免疫系统。
3 CRISPR-Cas系统的作用机制
巴兰古等的发现既是CRISPR-Cas系统研究的一个里程碑,也是一个转折点和分水岭。许多团队也开始意识到这一系统的重要性,从而促进这一领域的快速进展。

图3 CRISPR与细菌获得性免疫
2008年,荷兰瓦赫宁恩大学的范德欧斯特(John van der Oost)等通过研究大肠杆菌的CRISPR-Cas系统(Ⅰ型)发现CRISPR序列可转录并加工出非编码RNA——crRNA(CRISPR RNA),而crRNA介导了随后的干扰机制[13]。同一年,西北大学的松特海默尔(Erik Sontheimer)等则在表皮葡萄球菌(Staphylococcus epidermidis)的CRISPR-Cas系统(Ⅲ型)中发现,crRNA发挥干扰作用的靶点是DNA,而不像真核生物作用靶点为RNA[14]。这一发现不仅纠正了库宁假说,更重要的是为DNA编辑埋下伏笔。
2010年,对CRISPR-Cas系统的基本生物学作用和分子机制已有较清晰的理解,并将其应用于减少细菌噬菌体感染和细菌进化分析等。然而,CRISPR-Cas系统的应用范围极为有限,主要原因在于当时已研究的两种类型(Ⅰ型和Ⅲ型)都过于复杂,因此,寻找更为简单的体系成为一个重要方向。
卡彭蒂耶(Emmanuelle Marie Charpentier)是一位法国微生物学家,最初爱好为钢琴和舞蹈,但对医学的热爱使她最终投身于生命科学的研究。在巴黎皮埃尔和玛丽居里大学完成本科学业后,卡彭蒂耶来到附近的巴斯德研究所攻读博士学位。在这里她对基础科学产生了浓厚兴趣,特别是对细菌耐药机制尤为热爱。博士毕业后,卡彭蒂耶进入美国洛克菲勒大学开展博士后研究,重点关注肺炎链球菌的耐药性。后来她又在纽约大学医学院开展哺乳动物基因调控研究。在此过程中她一方面发现哺乳动物过于复杂,因此决定重回细菌研究;另一方面也意识到当时过于繁琐的哺乳动物基因编辑技术亟待改进。
2002年,卡彭蒂耶回到欧洲,首先在奥地利维也纳大学获得一份职位,并拥有独立的小实验室。尽管主要依赖短期基金项目支持,但仍孜孜不倦开展科学实验。随着哺乳动物RNA干扰现象的发现,卡彭蒂耶也开始关注细菌中非编码RNA的作用。在德国马普感染生物学研究所分子生物学家沃格尔(Jörg Vogel)协助下,卡彭蒂耶结合生物信息学方法在化脓链球菌(Streptococcus pyogenes)发现多种非编码RNA,特别是一类在CRISPR序列附近的新型小RNA,将其命名为反式激活CRISPR来源RNA(trans-activating CRISPR-derived RNA, tracrRNA),并推测它们与CRISPR系统有密切关系[15]。
由于当时已鉴定crRNA参与基因组DNA剪切,因此卡彭蒂耶推测tracrRNA可能通过与crRNA相互作用来引导酶发挥功能。卡彭蒂耶的这一假说非常激进,与传统观念相差甚远。一般认为特定序列DNA的识别由蛋白质如限制性内切酶、转录因子(ZFN和TELEN技术原理就是依据转录因子特异识别DNA序列实现剪切)等完成,尚未发现RNA介导。卡彭蒂耶这一“离经叛道”的想法吓坏了很多人,大部分研究生都不愿接手这一项目,最终德尔切瓦(Elitza Deltcheva)主动要求通过实验来验证卡彭蒂耶的假说。
2009年6月,卡彭蒂耶离开奥地利,加入新建的瑞典于默奥大学(Umeå University)微生物研究中心,但仍在维也纳大学保留实验室。德尔切瓦的实验进展比较顺利,不久就证实了卡彭蒂耶当初的推测。为避免文章被拒或延迟发表,他们花费一年多时间重复和完善实验以保证每一个结果的可靠性。2011年,卡彭蒂耶的发现在《自然》杂志发表,首次阐明tracrRNA在crRNA加工中的作用(图4):tracrRNA与转录出的CRISPR重复序列互补结合,这种结合在Cas9因子存在前提下被RNA酶Ⅲ识别和剪切,最终产生成熟crRNA[16]。这一发现具有十分重要的意义,相对于其他CRISPR系统往往需要一种crRNA但同时需多种Cas9蛋白参与,这一系统(Ⅱ型)则需两种RNA(crRNA和tracrRNA),虽增加RNA数量,却只需一种Cas9蛋白完成。这个系统如此简单,卡彭蒂耶意识到可将其改造为一种强有力的遗传操作工具用作基因编辑。
2011年,在波多黎各首都圣胡安(San Juan)举办的美国微生物会议上,卡彭蒂耶与美国加州大学伯克利分校结构生物学家杜德娜(Jennifer Anne Doudna)相遇。通过交流她们都对tracrRNA这一发现很感兴趣,随后决定合作开展进一步工作。
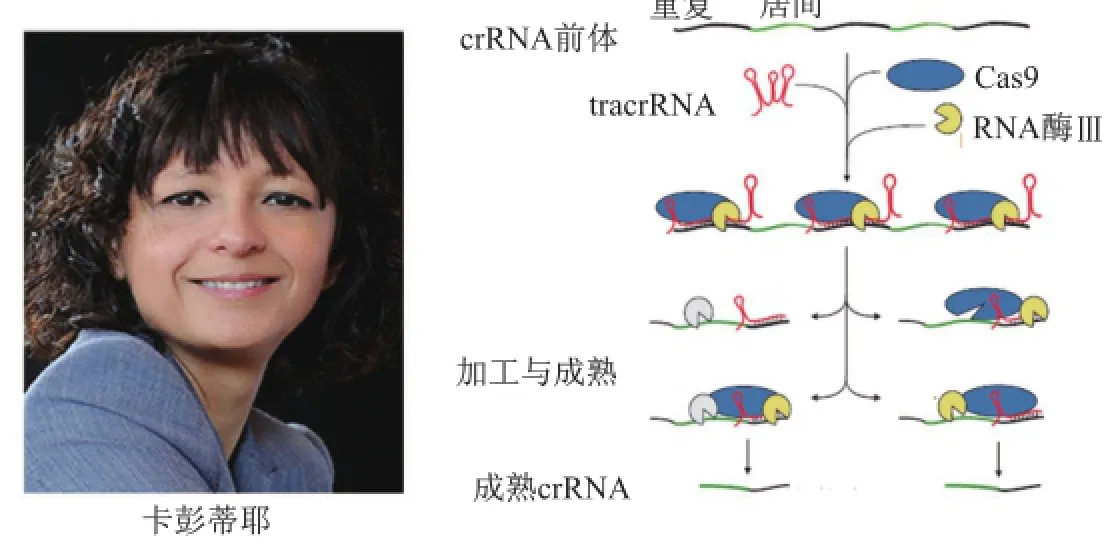
图4 tracrRNA生物作用的发现
4 CRISPR-Cas9基因编辑技术
杜德娜有着辉煌的学术背景,可称得上一位名副其实的“学术二代”。她从小就对科学发现充满巨大兴趣,1985年从波莫纳大学获得化学学士后进入哈佛大学跟随绍斯塔克(Jack Szostak,2009年由于端粒发现分享诺贝尔生理学或医学奖)进行博士学习。20世纪80年代,核酶(具有催化功能的RNA)的发现使科学界开始重新审视RNA的生物学功能,并越来越意识到RNA远比当初克里克中心法则中“遗传信息传递的中介”(三种RNA负责将DNA遗传信息传递给蛋白质)这一作用重要的多。因此,杜德娜开始关注RNA广泛的生物学作用,并在博士期间制备成功具有催化自我复制能力的RNA。
杜德娜在RNA方面的重要工作引起科罗拉多大学切赫(Tom Cech,1989年由于核酶发现而分享诺贝尔化学奖)的注意,因此邀请杜德娜加入实验室开展博士后研究——借助结构生物学工具研究核酶的作用机制。1991年,杜德娜成功制备出第一个核酶(四膜虫I类核酶)晶体,并采用X射线衍射技术获得这种RNA的三维结构,对理解核酶机制具有重要帮助。1994年,杜德娜加入耶鲁大学,继续与切赫合作开展核酶研究,同时她也逐渐成长为一位在RNA研究领域冉冉升起的科学新星。2002年,杜德娜从耶鲁大学转到加州大学伯克利分校,更为先进的技术平台使研究更加得心应手,她开始研究病毒RNA的作用。1998年,RNA干扰现象的发现进一步使科学家意识到RNA还是一类重要的基因表达调节分子,而杜德娜也将研究领域拓展到RNA干扰,研究参与这一过程的RNA酶如Argonaute和Dicer等的作用机制。杜德娜的学术贡献获得了科学界的高度认可,2004年当选为美国科学院院士[17]。
2005年,杜德娜也获悉原核生物CRISPRCas系统,并对此产生浓厚兴趣。由于当时推测采用RNA干扰方式杀死病毒,但详细机制可能不同于真核细胞。2007年,巴兰古等发现CRISPR-Cas系统的细菌免疫作用进一步坚定了杜德娜的决心,打算借助结构生物学方法探索这一新型“RNA干扰”中酶的作用机制。为此她专门成立一个细菌CRISPR-Cas系统的青年研究小组,主要包括博士后韦登赫福特(Blake Wiedenheft)和伊内克(Martin Jinek)等。该小组的优势在于拥有坚实的分子生物学、结构生物学和生物化学等基础,可将多种Cas蛋白完成纯化、结晶和结构测定,同时借助其他手段阐述酶的作用机制。他们先后确定Cas蛋白拥有DNA酶活性、RNA序列依赖的DNA核酸内切酶活性、crRNA加工活性[18]等。通过这些研究,杜德娜小组确定CRISPR-Cas是一种可对特定DNA序列进行定向剪切的细菌防御系统,但鉴于已研究的系统过于复杂,因此限制了这种系统的其他应用。
2011年之所以非常愉快地达成合作,原因在于杜德娜对这种简单的Ⅱ型系统特别是tracrRNA很着迷,而卡彭蒂耶则对Cas9作用机制更感兴趣。一位RNA专家(卡彭蒂耶)与一位酶学专家(杜德娜)的鼎力合作实现了最大程度的优势互补,而CRISPR-Cas9技术简单而言就是RNA(sgRNA)和酶(Cas9)。为尽快解决科学难题,合作沟通由杜德娜博士后伊内克和卡彭蒂耶博士后黑林斯基(Krzysztof Chylinski)具体负责,巧合的是他们都来自波兰,因此交流非常顺畅。他们合作纯化了Cas9蛋白,并证明Cas9具有依赖两种RNA的DNA核酸内切酶活性。Cas9拥有两个内切酶结构域,可分别对两条链进行切割。这个发现一方面拓展了卡彭蒂耶最初的发现,使tracrRNA拥有了双重作用(crRNA加工和靶DNA切割);另一方面还发现tracrRNA可与crRNA形成特殊二级结构来指导DNA剪切。这一现象立刻激发了研究激情,他们在保留二级结构基础上,将tracrRNA与crRNA两种RNA连接成一种RNA,称为单链引导RNA(single guide RNA,sgRNA),体外实验表明sgRNA也可指导Cas9蛋白完成对靶DNA的双链剪切(图5)。这一发现最终于2012年6月发表,它预示着可利用CRISPRCas9系统实现对目标DNA剪切,从而达到基因编辑的目的[19]。
卡彭蒂耶和杜德娜的发现既是细菌获得性免疫系统领域研究的里程碑,又是基因编辑领域的里程碑。CRISPR-Cas9 DNA精确剪切系统迅速成为多个实验室追逐的研究对象。
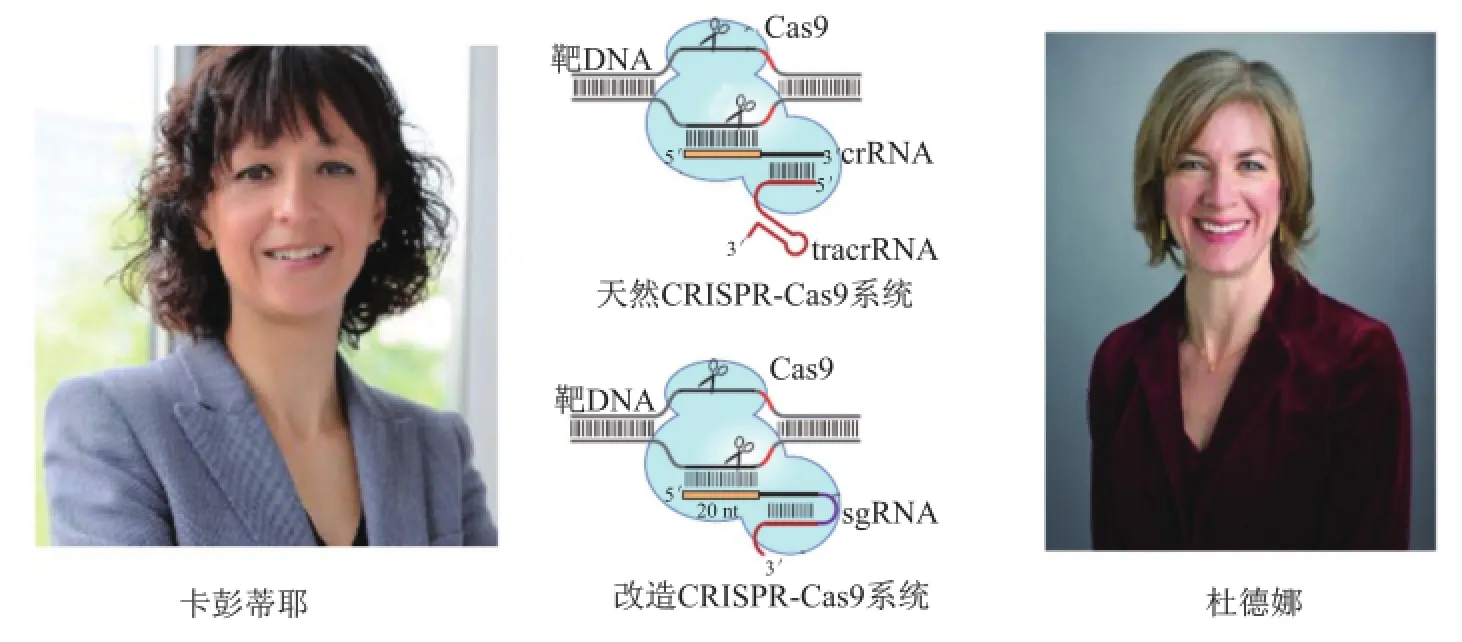
图5 CRISPR-Cas9技术的发明
5 CRISPR-Cas9技术的发展与应用
早在2011年,立陶宛维尔纽斯大学的斯克尼斯(Virginijus Siksnys)小组首次将嗜热链球菌的CRISPR-Cas9系统导入大肠杆菌,结果可使大肠杆菌获得噬菌体抵抗能力[20]。这一发现意味着CRISPR-Cas9系统可在不同物种内发挥相同功能,为将来应用于哺乳动物基因组编辑奠定坚实基础。此外,斯克尼斯小组也于2012年下半年实现体外DNA编辑[5]。2013年初,哈佛大学张锋和丘齐(George Church)则进一步在哺乳动物细胞内完成特定基因的编辑[21-22],特别是可利用多个sgRNA而实现多基因同时敲除,从而极大提升编辑效率和适用范围。2013年,先后有多家实验室成功利用CRISPR-Cas9完成基因编辑,引起了一场持续至今的研究热潮[23-24]。
CRISPR-Cas9技术已在全世界上千家实验室得到广泛应用。最主要的应用领域是基因组编辑,已在人细胞系和多种模式生物如酵母、果蝇、线虫、斑马鱼、小鼠、大鼠、猪和猴等完成感兴趣基因的编辑,并在此基础上建立多种疾病模型,为阐明疾病发生的分子机制和药物筛选提供重要平台。天然Cas9酶还被进行人工改造,一方面可减少基因编辑过程的脱靶效应,另一方面还被应用于转录调控(激活或抑制)等研究。随着CRISPR-Cas9技术的完善和安全性的改进,也可能在将来疾病治疗方面发挥重要作用[24-25]。
CRISPR-Cas9技术本身不仅取得巨大成功,还推动基因编辑新工具的层出不穷,出现了一系列改进版和更新版。Cpf1与Cas9类似,也是一种RNA依赖的DNA核酸内切酶,但在剪切靶DNA时只需crRNA参与,而无需tracrRNA辅助[26],用做基因编辑时可大大缩短引导RNA长度,因此CRISPR-Cpf1系统将更为简单。2009年,库宁等进一步提出假说,原核生物除CRISPR-Cas系统外,Argonaute蛋白(Ago)系统(不同于CRISPRCas系统只存在于原核生物,Argonaute蛋白从原核生物到真核生物都普遍存在,并且在真核生物RNA干扰过程中的作用机制已得到全面研究)也可发挥免疫作用以抵御质粒等外源DNA入侵,暗示着也可被改造为基因编辑工具。2014年,范德欧斯特等首次利用古菌——噬热栖热菌(Thermus thermophilus)的Ago蛋白(TtAgo)完成单链DNA(single stranded DNA,ssDNA)指导的靶DNA剪切[27],然而这种蛋白只能在65℃以上才发挥活性,从而限制了细胞及机体内的应用。这三种系统采用类似策略完成对目的基因的编辑(图6),随着技术的完善,将在基础研究和实际应用方面有着广阔的发展空间。
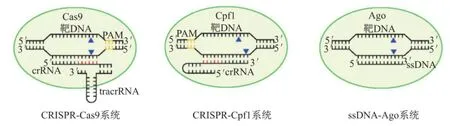
图6 三种编辑方法的比较
6 重大意义
CRISPR-Cas9技术的广泛应用证明了这项发明的重要意义,这一技术也于2013和2015年两次入选美国《科学》评选的十大科学突破,许多为此技术发明做出卓越贡献的科学家特别是卡彭蒂耶和杜德娜等已获得多项科学大奖[28]。科学界普遍认为,这项发明将来获得诺贝尔奖是毋容置疑的[15]。从获奖类别上讲,类似DNA重组、聚合酶链式反应等技术授予化学奖的可能性更高。但无论如何,这一突破对生命科学、医学等发展具有极大的推动作用。
CRISPR-Cas9技术的发明历程为我们提供了三个方面的思考。
首先,重视微生物研究。这是科恩伯格在酶学研究“十诫”中的名言[29]。微生物基础生命现象的生物化学机制研究为生物技术提供了思想源泉和操作工具。聚合酶链式反应(PCR)和桑格法测序来源于对微生物复制研究基础上DNA聚合酶纯化和机制研究,基因工程的突破来自微生物限制-修饰系统研究中发现的限制性内切酶,而CRISPR-Cas9技术则是对细菌获得性免疫系统的深入研究。因此,一系列重大生物技术的突破可称得上是微生物学与生物化学的完美结合。
其次,重视基础科学研究。这是科学发展的一般规律。科学与技术发展往往相辅相成,互相促进。科学理论突破为技术发明提供了思想源泉,而技术革新则为科学进步提供实验保证。科学的真谛原本就是为了探索自然界奥秘,在认识世界的基础上最终达到改造世界的目的,从而为人类生产能力提高和生活水平改善提供重要帮助。CRISPR-Cas9技术就是通过对细菌奇异序列的深入探索发现了新型的细菌获得性免疫系统,而进一步的机制研究触发了改造该系统应用于基因编辑的灵感。
最后,重视科学发展规律。科学研究一个重要特点就在于“前人植树,后人乘凉”。从最初不经意的发现(1987年),到沉默十几年的过渡(1987年到2005年),再到一朝突破(2007年和2011年)引发科学快速发展,到最终的技术发明(2012年),前后跨越25年(图7)。CRISPR-Cas9技术的发明是一种水到渠成的结果,而非刻意为之可达到的效果。科学研究充满了巨大的不确定性,因此应更多地鼓励和支持“看不到明显效益”的基础研究。目前科研领域过多关注研究的应用价值和市场潜力,然而欲速则不达,这种短视的眼光束缚了科学更好的发展。
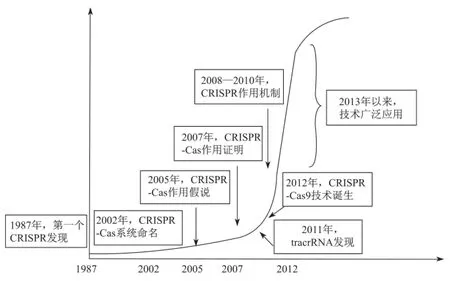
图7 CRISPR-Cas9技术发展的重大事件时间表
(2016年5月24日收稿)■
[1] 郭晓强. 细菌遗传学之父——乔舒亚·莱德伯格[J]. 自然杂志, 2012, 34(2): 119-124.
[2] 郭晓强. DNA酶学之父——科恩伯格[J]. 自然杂志, 2009, 31(4): 245-248.
[3] 郭晓强. 基因组学之父: 桑格[J]. 科学, 2014, 66(5): 59-62.
[4] SHINO Y, SHINAGAWA H, MAKINO K, et al. Nucleotide sequence of the iap gene, responsible for alkaline phosphatase isozyme conversion in Escherichia coli, and identifcation of the gene product [J]. J Bacteriol, 1987, 169(12): 5429-5433.
[5] LANDER E S. The heroes of CRISPR [J]. Cell, 2016, 164(1/2): 18-28.
[6] MOJICA F J, DÍEZ-VILLASEÑOR C, SORIA E, et al. Biological signifcance of a family of regularly spaced repeats in the genomes of Archaea, Bacteria and mitochondria [J]. Mol Microbiol, 2000, 36(1): 244-246.
[7] JANSEN R, EMBDEN J D, GAASTRA W, et al. Identification of genes that are associated with DNA repeats in prokaryotes [J]. Mol Microbiol, 2002, 43(6): 1565-1575.
[8] MOJICA F J, DÍEZ-VILLASEÑOR C, GARCÍA-MARTÍNEZ J, et al. Intervening sequences of regularly spaced prokaryotic repeats derive from foreign genetic elements [J]. J Mol Evol, 2005, 60(2): 174-182.
[9] POURCEL C, SALVIGNOL G, VERGNAUD G. CRISPR elements in Yersinia pestis acquire new repeats by preferential uptake of bacteriophage DNA, and provide additional tools for evolutionary studies [J]. Microbiology, 2005, 151(3): 653-663.
[10] BOLOTIN A, QUINQUIS B, SOROKIN A, et al. Clustered regularly interspaced short palindrome repeats (CRISPRs) have spacers of extrachromosomal origin [J]. Microbiology, 2005, 151(8): 2551-2561.
[11] MAKAROVA K S, GRISHIN N V, SHABALINA S A, et al. A putative RNA-interference-based immune system in prokaryotes: computational analysis of the predicted enzymatic machinery, functional analogies with eukaryotic RNAi, and hypothetical mechanisms of action [J]. Biol Direct, 2006, 1: 7.
[12] BARRANGOU R, FREMAUX C, DEVEAU H, et al. CRISPR provides acquired resistance against viruses in prokaryotes [J]. Science, 2007, 315(5819): 1709-1712.
[13] BROUNS S J, JORE M M, LUNDGREN M, et al. Small CRISPR RNAs guide antiviral defense in prokaryotes [J]. Science, 2008, 321(5891): 960-964.
[14] MARRAFFINI L A, SONTHEIMER E J. CRISPR interference limits horizontal gene transfer in staphylococci by targeting DNA [J]. Science, 2008, 322(5909): 1843-1845.
[15] ABBOTT A. The quiet revolutionary: How the co-discovery of CRISPR explosively changed Emmanuelle Charpentier’s life [J]. Nature, 2016, 532(7600): 432-434.
[16] DELTCHEVA E, CHYLINSKI K, SHARMA C M, et al. CRISPR RNA maturation by trans-encoded small RNA and host factor RNase III [J]. Nature, 2011, 471(7340): 602-607.
[17] MARINO M. Biography of Jennifer A. Doudna [J]. Proc Natl Acad Sci USA, 2004, 101(49): 16987-16989.
[18] HAURWITZ R E, JINEK M, WIEDENHEFT B, et al. Sequence- and structure-specific RNA processing by a CRISPR endonuclease [J]. Science, 2010, 329(5997): 1355-1358.
[19] JINEK M, CHYLINSKI K, FONFARA I, et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity [J]. Science, 2012, 337(6096): 816-821.
[20] SAPRANAUSKAS R, GASIUNAS G, FREMAUX C, et al. The Streptococcus thermophilus CRISPR/Cas system provides immunity in Escherichia coli [J]. Nucleic Acids Res, 2011, 39(21): 9275-9282.
[21] CONG L, RAN F A, COX D, et al. Multiplex genome engineering using CRISPR/Cas systems [J]. Science, 2013, 339(6121): 819-823.
[22] MALI P, YANG L, ESVELT K M, et al. RNA-guided human genome engineering via Cas9 [J]. Science, 2013, 339(6121): 823-826.
[23] PENNISI E. The CRISPR craze [J]. Science, 2013, 341(6148): 833-836.
[24] DOUDNA J A, CHARPENTIER E. Genome editing. The new frontier of genome engineering with CRISPR-Cas9 [J]. Science, 2014, 346(6213): 1258096.
[25] HSU P D, LANDER E S, ZHANG F. Development and applications of CRISPR-Cas9 for genome engineering [J]. Cell, 2014, 157(6): 1262-1278. [26] ZETSCHE B, GOOTENBERG J S, ABUDAYYEH O O, et al. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system [J]. Cell, 2015, 163(3): 759-771.
[27] SWARTS D C, JORE M M, WESTRA E R, et al. DNA-guided DNA interference by a prokaryotic Argonaute [J]. Nature, 2014, 507(7491): 258-261.
[28] 郭晓强, 黄卫人, 蔡志明. 一种全新DNA编辑工具——CRISPR-Cas9技术[J]. 科学通报, 2015, 60(30): 2833-2835.
[29] KORNBERG A. Ten commandments: lessons from the enzymology of DNA replication [J]. J Bacteriol, 2000, 182(13): 3613-3618.
(编辑:沈美芳)
The invention of CRISPR-Cas9 technology: 25-years scientific journey
GUO Xiaoqiang
Shenzhen Second People’s Hospital, Shenzhen 518035, Guangdong Province, China
CRISPR-Cas (clustered regularly interspaced short palindromic repeat-CRISPR-associated) is an important acquired immune system for prokaryotes. CRISPR sequences can be transcribed and processed into non-coding RNA, crRNA (CRISPRRNA). These Cas proteins can complete the RNA mediated target DNA cut using their DNA exonuclease, which is important for bacteriophage and plasmid DNA invader defense in prokaryotes. The CRISPR-Cas9 gene editing was invented on the system, which is widely used in life science. In the article, the following knowledge was described including discovery of CRISPR sequence, nomenclature of CRISPR, prediction of biological role, confrmation of experiment, research on mechanism and improvement of system. It is important for the comprehensive understanding of CRISPR-Cas9 technology invention.
acquired immune system, prokaryote, gene editing, CRISPR-Cas9
10.3969/j.issn.0253-9608.2016.04.008
†通信作者,E-mail: xiaoqiangguo123@163.com

