丙型肝炎病毒NS5A抑制剂的3D-QSAR研究
孟令鑫,刘蒙蒙,王远强,林治华,2(重庆理工大学药学与生物工程学院,重庆400054;2.重庆大学化学化工学院,重庆400044)
丙型肝炎病毒NS5A抑制剂的3D-QSAR研究
孟令鑫1,刘蒙蒙1,王远强1,林治华1,2
(1重庆理工大学药学与生物工程学院,重庆400054;2.重庆大学化学化工学院,重庆400044)
丙型病毒性肝炎感染是输血后肝炎的主要病因之一。NS5A蛋白的小分子抑制剂显示出很强的体外抑制病毒生长的活性,并且初步的临床评价也证实了NS5A抑制剂能很好地抑制体内丙型肝炎病毒的生长。因此,研发高效的NS5A小分子抑制剂为治疗丙型肝炎提供了新的策略。进行了daclatasvir丙型肝炎病毒NS5A复制抑制剂的三维定量构效关系(3D-QSAR)研究,通过SYBYL-X 2.1.1分子模拟软件系统搜寻方法搜寻出化合物的最低能量构象,然后在Triops力场中用共轭梯度最小化进行优化。应用比较分子力场分析(CoMFA)和比较分子相似性指数分析(CoMSIA)进行分子活性构象的选择、分子叠合、建立空间场范围以及数据统计。用22个衍生物作为训练集建立模型,用6个衍生物作为测试集来验证模型的优劣。结果表明:CoMFA模的交叉相互验证系数q2=0.578,回归系数r2=0.939,CoMSIA模型的q2=0.584,r2=0.968。这些结论为丙型肝炎病毒NS5A复合体抑制剂的药物设计和筛选提供了理论依据。
丙型肝炎病毒NS5A抑制剂;三维定量构效关系;比较分子场方法;比较分子相似性指数分析法
HCV是主要的人类病原体,全球感染人数约1.7亿,是人免疫缺陷病毒I型感染人数的5倍[1]。这些HCV感染个体中的相当一部分会发展成严重的进行性肝病,包括肝硬化和肝细胞癌[3]。对HCV生命周期[2]的探讨使我们得以研究针对不同病毒复制阶段直接作用的抗病毒药物。很多新型抗病毒药物,如NS3/4A蛋白酶抑制剂、核苷(酸)类似物和非核苷类抑制剂,以及NS5A抑制剂均已进入临床试验阶段[4]。丙型肝炎病毒NS5A复制抑制剂是可用于治疗HCV感染患者的化合物,可选择性地抑制HCV病毒的复制[5]。目前治疗HCV的标准方法为使用聚乙二醇化-干扰素和利巴韦林的联合疗法,目前该方法在达到持续病毒响应上没有理想的成功率,且会引起多种副作用[6]。因此,对开发解决当下这种医学需要的有效疗法是十分必要的。
比较分子场分析方法(CoMFA)原理:如果作用在一个受体上的是一组相似的化合物,并且作用方式相同,那么这组化合物的生物活性就由每个化合物周围分子场的差别来决定,这种分子场可以显示出受体与药物分子之间非键的相互作用特点。本文根据合理的重叠规则,将已经确立好的研究体系中各个化合物的药效构象重叠在一个空间网格上,这个空间网格将包容全部的化合物分子;使用一个探针原子,利用其在网格中移动的一定步长,计算在化合物构象式与每个点之间的静电势、疏水性和立体阻障作用;利用偏最小二乘法(partial least square,PLS)来确定区分被研究化合物活性的最少网格点(以障疏水性、静电势、立体阻相互之间的作用来表示),即得出3D-QASR;作出CoMFA系数图,根据系数图上的图像特征分析出哪些地方其场强弱对被研究化合物的生物活性影响最大,并以此为据,设计出具有更强生物活性的新化合物。而比较分子相似因子分析方法(COMSIA)与CoMFA方法最大的区别是分子场的能量函数采用的是与距离有关的高斯函数。在CoMSIA方法中,以化合物的立体场、疏水场、静电场和氢键场表示其分子特征,其中氢键场可分为氢键的给予体和氢键的受体两种。这种与距离有关的高斯函数弥补了CoMFA方法中只由静电场和立体场的函数表达的缺陷。
本实验中,通过运用sybyl 2.1软件将分子优化,获取了分子最优的叠合构象,并且通过比较分子场分析(CoMFA)建模,通过比较分子相似性指数分析(CoMSIA)考察模型的合理性。这些模型通过22个具有生物活性的化合物作为训练集来预测作为测试集的6个衍生物,从而验证模型的有效性,预测值和实验值能较好吻合会使模型具有强大的说服力。
1 研究方法
1.1数据来源
该化合物(见表1)的基本骨架结构见图1。根据随机数字表,从28个化合物中抽取6个作为预测集(含“*”号),余下22个作为训练集。以训练集建立3D-QSAR模型,并对预测集中化合物活性进行预测[7],进而检验所建模型的可靠性与预测能力。在CoMFA分析中这28个化合物以半最大效应制浓度(EC50)表示,实验值见表1。

表1 目标化合物(1-28)的结构与活性
1.2生物活性分子构象的优化
本文研究的丙型肝炎病毒NS5A复制抑制剂的结构通式如图1所示。建立分子定量构效关系(QSAR)的重要步骤之一就是化合物活性构象的确定[8]。本文选用Tripos力场MMFF94电荷将28个目标化合物进行优化,将最大重复次数增加到1 000,Gradient[21-23]减到0.005,从而得到各分子的优化构象,进而在3D定量构效关系中建立模型以进行研究分析。

图1 化合物的结构通式
1.3分子叠合
构象分子的叠合在3D-QSAR中是一个至关重要的步骤[9]。本文采用了将原子叠合的方式,即以生物活性最高的14号化合物作为模板分子,将各个分子的结构进行观察,得到了32个叠合位点,然后将分子中有相同原子特征的骨架结构进行叠合。28个化合物分子的叠合如图2所示。

图2 目标化合物的叠合图
1.4CoMFA模型的建立
采用SYBYL软件的QSAR模块进行分析。将上述叠加好的活性构象分子置于SYBYL/CoMFA自动生成的作用区域中。首先用PLS方法进行leave-one-out(LOO)交互验证,从而计算出所建立模型的最佳主成份数(n)和交叉验证的回归系数[10]。一般认为,交叉验证系数q2值大于0.5时,所建立的模型就有较可靠的预测能力[11]。然后再利用非交叉验证法对其进行回归计算,从而得到较好的CoMFA模型[12]。
1.5CoMSIA模型的建立
对于CoMSIA模型的建立,选取静电场(E)、立体场(S)、疏水场(H)、氢键受体场(A)和氢键供体场(D)来考虑受体与化合物之间的影响[13]。首先将全部5种场考虑用于建立CoMSIA模型,若所得到的计算结果中q2并不理想,即没有很好的预测能力,则选择通过对各种场组合的计算来获得具有较好交叉验证系数的分子场[14]。对于最佳的CoMSIA结果,交叉验证采用leave-one-out(LO0)得到最佳组分数和交叉验证的回归系数。然后进行非交叉验证,建立CoMSIA模型[15]。
2 结果与讨论
2.1统计结果
在CoMSIA模型的建立中,由于当把全部5种场考虑进去后所得到的q2大于0.5时才具有很好的预测能力,因此选择通过分别对各种场的组合进行计算来获得具有较好交叉验证系数的分子场。表2为其中一组活性的组合对CoMSIA和CoMFA结果的影响。最佳分子场组合为立体场(S)、静电场(E)、疏水场(H)、氢键受体场(A)和氢键供体场(D)[16]。
表3显示了CoMFA和CoMSIA的3D-QSAR模型的统计结果。在CoMSIA当中,只考虑了立体场(S)、静电场(E)、氢键受体场(A)和氢键供体场(D)的贡献,所得交叉验证系数较为满意[17](q2=0.584,R2=0.968,F=46.813,最佳组分数为2)。
从统计结果来看,CoMFA和CoMSIA方法都能对该系列化合物的结构活性关系给出较好相关模型[18]。CoMFA所得q2值比CoMSIA略低,CoMSIA的最佳组分数比相应CoMFA结果大,CoMFA模型考虑了静电场和立体场的贡献,而CoMSIA则考虑了立体场(S)、静电场(E)、氢键受体场(A)和氢键供体场的贡献[19]。从表4来看,除了个别化合物,两种模型对于训练集和测试集中化合物的活性预测还是比较准的。
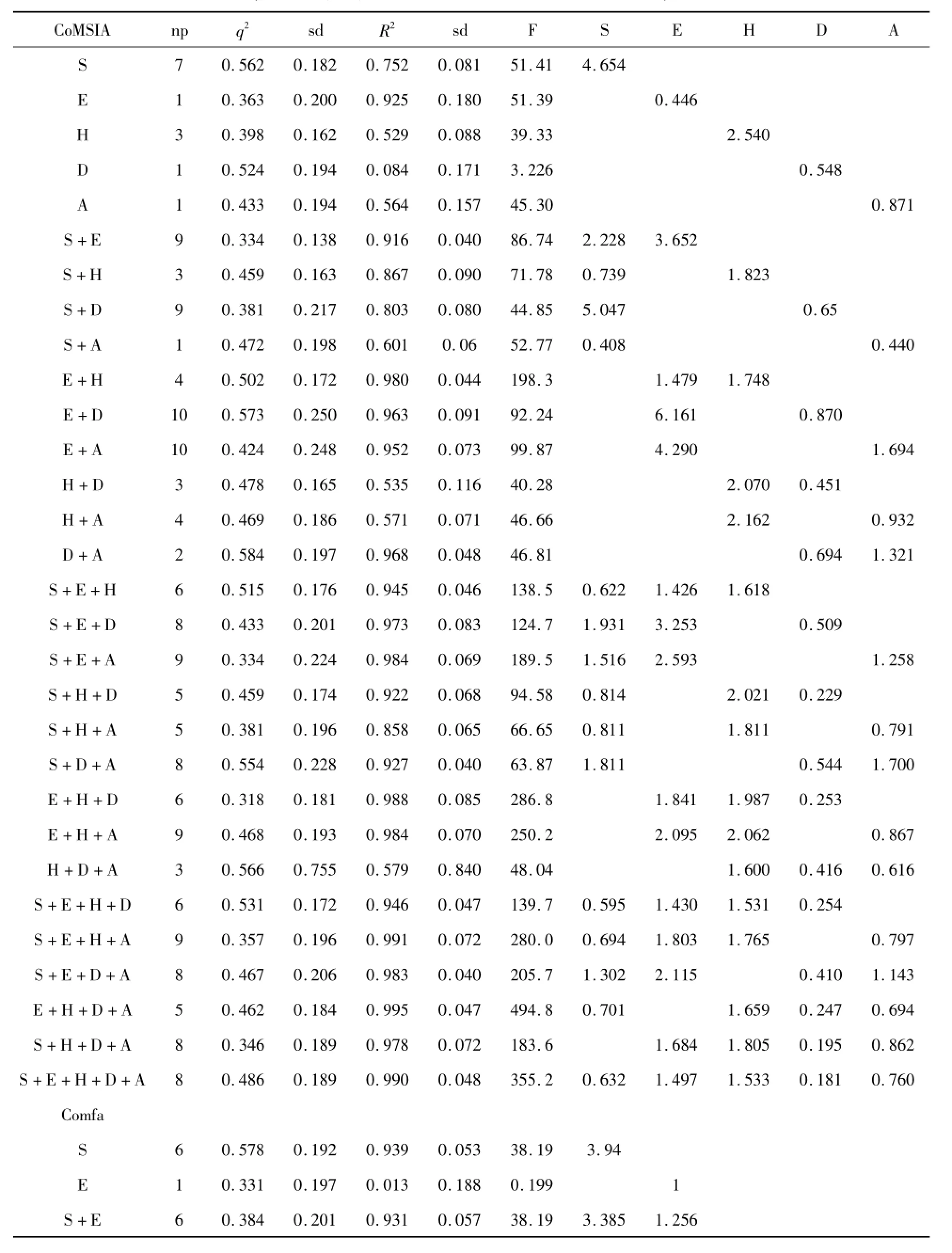
表2 各种分子场的组合对CoMSIA和CoMFA结果的影响

表3 CoMFA和CoMSIA的3D-QSAR模型的统计结果
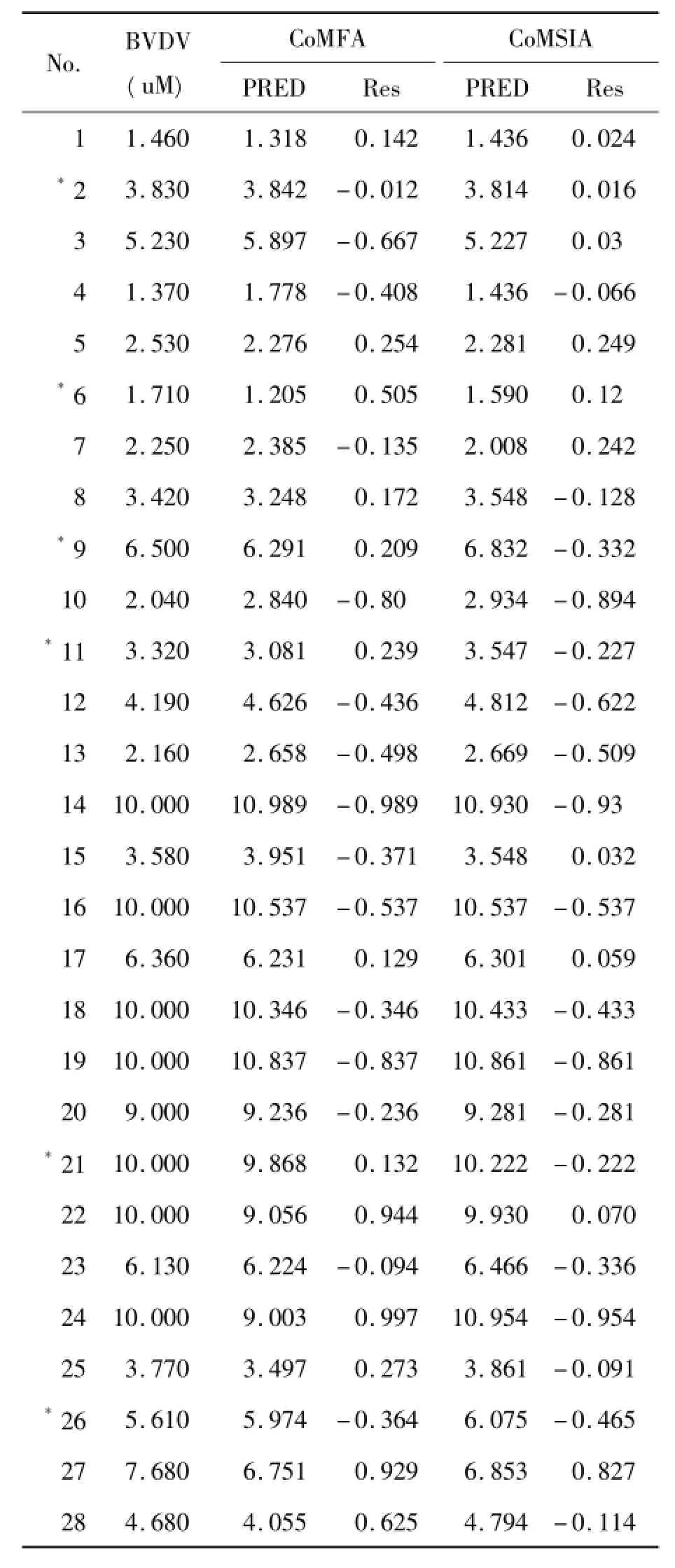
表4 目标化合物的活性
2.2QSAR模型的验证
结合CoMFA和CoMSIA进行3D-QSAR研究有助于得到更为可靠的预测模型[20]。在本研究中,最佳CoMFA模型和CoMSIA模型的R2值分别为0.939和0.968,CoMSIA的R2值要略高于CoMFA。用此CoMFA和CoMSIA模型来验证其稳定性和可预测性。先预测了22个训练集化合物,然后把没有进行预测的6个测试集化合物放入已经建好的模型中进行预测,从而对建立好的模型进行验证。表4中列出了28个化合物在CoMFA和CoMSIA模型中活性的预测值与实验值。在建立好的模型中,所有化合物的预测值与实验值的活性相关性表现在图3中。从实验值和预测值的相关图中可以看出,建立好的CoMFA和CoMSIA模型都对化合物有很好的预测性,其中CoMSIA模型有着更好的预测性。CoMFA模型的相关图显示,所有的训练集数据点并没有都集中在趋势线上,还是有些分散在趋势线两侧;CoMSIA模型的相关图显示,基本上所有的训练集数据点都集中于趋势线。由此可以看出,通过22个训练集化合物所建立的3D-QSAR模型有较强的相关性[23]。利用已经建立好的3D-QSAR模型对没有进行预测的6个测试集化合物进行活性预测,预测值和实测值也都能靠近趋势线且分布在其两侧,说明这个3D-QSAR模型的预测性是比较好的,可以用于预测与实验中的模板化合物骨架结构相似的分子的生物活性[24]。
2.3三维等值线图
通过CoMFA的一系列计算,可以很明显地看到分子在包括静电场、立体场在内的各种场性质条件下生物活性的影响。叠合分子不同性质的等值空间区域由不同颜色的等势面区域表示。其中静电效应以蓝色和红色的区域来代表,立体效应以黄色和绿色的区域来代表[25]。
图4(见本期封三)为以化合物3为参照分子的CoMFA模型的三维立体场等值线图,图4(a)为CoMFA模型的立体等值线图,图4(b)为CoMFA模型的静电场等值线图。图4(a)中绿色区域表示在该区域中增大取代基的体积,有利于增加化合物的生物活性,在黄色区域内增大立体位阻将使化合物生物活性递降。图4(b)中蓝色区域表示增加带正电荷基团有利于提高化合物的生物活性,而在红色区域增加带负电荷基团可使化合物的生物活性上升[26]。

图3 3D-QSAR模型中训练集和测试集EC50的实验值数据和预测值数据之间的相关性
图5(见本期封三)显示了以化合物3为参照分子的CoMSIA模型的三维等值线图。图5(a)为CoMSIA模型的立体场分布图;图5(b)为CoMSIA模型的静电场分布图;图5(c)为CoMSIA模型的氢键受体场分布图;图5(d)为CoMSIA模型的氢键供体场分布图;图5(e)为CoMSIA模型的疏水场分布图。从CoMSIA模型的立体场分布图可以看出:它与CoMFA的立体场分布图大致相同,说明此模型的立体场等值线分布图还是比较可靠的,其对化合物改造的指导作用也是大致相同。例如,从图5(b)反映的静电场分布图上可以看出:在蓝色的区域加强化合物的正电性,在红色区域引入电负性较大的基团有助于提高化合物的生物活性。图5(c)提供了关于氢键供体场的有关信息,在蓝绿色空间给化合物引人氢键供体的基团或原子有利于提高化合物与受体的亲和力从而增加其生物活性,而在黄色区域氢键受体场的增加对提高化合物的活性不利。从图5(d)显示的氢键受体场分布图可以看出,在紫色给化合物引入氢键受体的基团或原子有利于增加化合物与受体的亲和力而提高活性,红色区域增加氢键受体场不利于提高化合物的活性。
3 结论和展望
本文对丙型肝炎病毒NS5A复制抑制剂进行了系统的3D-QSAR研究。通过分子力学的结构优化,选取分子的最低能量构象并进行了分子叠合。基于LOO法建立了在三维水平上的定量关系模型,其CoMFA的交互验证系数q2=0.578,传统的相关系数R2=0.939,CoMSIA的交互验证系数q2=0.584,传统的相关系数R2=0.968。利用此模型对6个化合物进行预测,预测值与实验值非常吻合,表明所建模型具有良好的稳定性及预测能力。根据CoMFA和CoMSIA的三维等值线图,解释了化合物中各位置上取代基对化合物活性的影响,为进一步设计、合成高活性的标题化合物提供一定的理论依据。
查阅相关文献与数据库Reaxys后,以活性最高的模板骨架分子14号为例,笔者设计了其合成路线,见图6和7。
实验过程中也有一些问题需要进一步研究和探讨:QSAR研究需要以大量高质量的活性数据为基础,而由于实验条件和方法的差异,不同来源的数据可比性差,往往不能综合起来建模。活性数据样本量直接影响着模型的好坏,一般认为模型训练集的样本量应高于建模参数个数的5倍以上,即至少需要5个以上样本的活性数据才能建立相对稳健的单参数模型。此次试验可能会出现不稳定因素。由于化合物的活性通常受到多个因素的制约,因而建模时又应考察尽量多的相关结构参数以获得高拟合精度和强预测能力的模型。虽然数据库有许多用于QSAR研究的结构参数,但远不能满足QSAR研究的需要。QSAR方法的研究不会只是停留在采用一些经验参数定量描述有机化合物的活性,而是会向着更加注重模型的理论性、智能化和程序化方向发展。如何提取出表达物质结构特征的结构描述符才是QSAR研究的关键。
本文对丙型肝炎病毒NS5A复制抑制剂进行了定量构效关系的研究,虽然得到了较好的结果,但还有待进一步深入研究和不断完善。总的来说,丙型肝炎病毒NS5A复制抑制剂药物的研究已经取得了重大的成果,其构效关系研究还有较广阔的空间和发展前景,有效的丙型肝炎病毒NS5A复制抑制剂进入临床并走进市场指日可待。

图6 14号模板分子
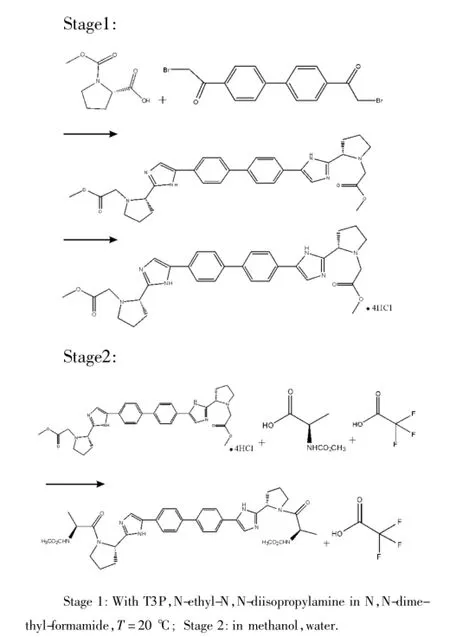
图7 合成路线
[1]龚巧玲,王建,贺福初.丙型肝炎病毒非结构蛋白NS2的研究进展[J].生命科学,2009,32(1):12-13.
[2]龚国忠,Siddiqui A,苏先狮.丙型肝炎病毒非结构蛋白5a对细胞周期的影响[J].中华肝脏病杂志,2004,15(1):51-53.
[3]党晓燕,成军,邓红.多嘧啶序列结合蛋白与丙型肝炎病毒的关系[J].世界华人消化杂志,2003,23(14):22 -22.
[4]代志琰,李刚.丙型肝炎病毒NS5A分子及其干扰素敏感决定区的研究进展[J].研究生学刊:自然科学医学版,2002,13(4):15-17.
[5]熊育林,张长江,王小红.丙型肝炎病毒基因结构及功能[J].中国生物化学与分子生物学报,2008,18(7):31-32.
[6]许小亮,李君武.丙型肝炎病毒非结构蛋白3蛋白酶抑制剂的研究进展[J].医学研究生学报,2005,16(10):11-12.
[7]Lohmann V,Korner F,Koch J O,et al.Replication of subgenomic hepatitis C virus RNAs in a hepatoma cell line[J].Science,1999,28(5):110-113.
[8]Elazar M,Cheong K H,Liu P,et al.Amphipathic helixdependent localization of NS5A mediateshepatitis C virus RNA replication[J].JVirol,2003,7(7):6055-6061.
[9]Shirota Y,Luo H,Qin W P,et al.Hepatitis C virus(HCV)NS5A binds RNA-dependent RNA polymerase(RdRP)NS5B and modulates RNA-dependent RNA polymerase activity[J].J Biol Chem,2002,27(7):11149 -11155.
[10]Qin W,Yamashita T,Shirota Y,et al.Mutational analysis of the structure and functions of hepatitis C virus RNA-dependent RNA polymerase[J].Hepatolog,2001,3(3):728-737.
[11]Shimakami T,Hijikata M,Luo H,et al.Effect of interaction between hepatitis C virus NS5A and NS5B on hepatitis C virus RNA replication with the hepatitis C virus replicon[J].JVirol,2004,7(8):2738-2748.
[12]Iusida M I,Nagano FujiiM,Nidom CA,etal.Correlation between mutations in the interferon sensitivity-determining region of NS5A protein and viral load of hepatitis C virus subtypes 1b,1c,and 2a[J].JClin Microbiol,2001,39(11):3858-3864.
[13]He Y,YanW,Coito C,et al.The regulation of hepatitis C virus(HCV)internal ribosome-entry site-mediated translation by HCV replicons and nonstructural proteins[J].J Gen Virol,2003,84(3):535-543.
[14]Garcia2Saez I,DeBonis S,Lopez R,et al.J.Biol.Chem.,2007,28(2):9740-9747.
[15]Mayer T U.Small molecule inhibitor of mitotic spindle bipolarity identified in a phenotype-based screen[J].Science,2008,28(6):971-974.
[16]Kozielski F,DeBonis S,Skoufias D A.Screening for inhibitors of microtubule-associated motor proteins[J]. Methods Mol Med,2007,13(7):189-207.
[17]Orr G A,Verdier-Pinard P,McDaid H,et al.Mechanisms of Taxol resistance related to microtubules[J].Oncogene,2008,2(2):7280-7295.
[18]Kavallaris M.Microtubules and resistance to tubulinbinding agents[J].Nat Rev Cancer,2010,10(8):194 -204.
[19]Hickson I,Zhao Y,Richardson C J,et al.Identification and Characterization of a Novel and Specific Inhibitor of the Ataxia-TelangiectasiaMutated Kinase ATM[J].Cancer Res,2004,6(4):9152-9159.
[20]Podevin P,Sabile A,Gajardo R,etal.Expression of hepatitis C virus NS5A naturalmutants in a hepatocytic cell line inhibits the antiviral effectof interferon in a PKR-independent manner[J].Hepatology,2001,3(3):1503 -1511.
[21]Aizaki H,Saito S,Ogino T,et al.Suppression of interferon-induced antiviral activity in cells expressing hepatitis C virus proteins[J].J Interferon Cytokine Res,2000,20(3):1111-1120.
[22]Paterson M,Laxton C D,Thomas H C,et al.Hepatitis C virus NS5A protein inhibits inteferon antiviral activity,but the effectsdo notcorrelatewith clinical response[J]. Gastroenterology,1999,1(17):1187-1197.
[23]Williams B R.Signal integration via PKR[J].SciSTKE,2001,8(9):2-22.
[24]Nousbaum J B,Polyak S J,Ray S C,et al.Prospective characterization of full-length hepatitis C virus NS5A quasispecies during induction and combination antiviral therapy[J].JVirol,2000,7(4):9028-9038.
[25]Noguchi T,Satoh S,Noshi T,et al.Effects of mutation in hepatitis C virus nonstructural protein 5A on interferon resistancemediated by inhibition of PKR kinase activity in mammalian cells[J].Microbiollmmunol,2001,4(5):829-840.
[26]Enomoto N,Sakuma L,Asahina Y,et al.Mutations in the nonstructural protein 5A gene and response to interferon in patients with chronic hepatitis C virus 1b infection[J].N Engl JMed,1996,3(34):77-81.
(责任编辑何杰玲)
3D-QSAR Studies of Hepatitis C Virus NS5A Inhibitors
MENG Ling-xin1,LIU Meng-meng1,WANG Yuan-qiang1,LIN Zhi-hua1,2
(1.College of Pharmacy and Biological Engineering,Chongqing University of Technology,Chongqing 400054,China;2.Chemistry and Chemical Engineering,Chongqing University,Chongqing 400044,China)
Viral hepatitis C infection is one of themain causes of the hepatitis after blood transfusion. NS5A protein of smallmolecule inhibitors shows strong activity in inhibiting the growth of the vitro virus,and the preliminary clinical evaluation also confirmed that NS5A inhibitors can inhibit the growth of hepatitis c virus in the body.Therefore,the research and developmentof efficientNS5A smallmolecule inhibitors provides a new strategy for the treatment of hepatitis C.In this study,we investigatedthe daclatasvir hepatitis C virus NS5A inhibitor complex 3D-QSAR,and searched out the lowest energy conformations of compounds through SYBLE-X 2.1.1 molecularmodeling software system search method,and then Triops force field conjugate gradientminimization optimization.Comparative Molecular Field Analysis(CoMFA)and Comparative Molecular Similarity Indices Analysis(CoMSIA)were used to havemolecular active conformation selection,molecular alignment,as well as the establishmentof spatial statistics field range.In this experiment,taking 22 derivatives as the training set to build themodel,themerits of the model was validated with 6 derivatives as a test set.Results show that Cross CoMFA model's mutual authentication factor q2=0.578,and the regression coefficient r2=0.939,while CoMSIA model q2=0.584,and the r2=0.968.These conclusions laid a reliable theoretical basis for drug design and screening of hepatitis C virus NS5A complex inhibitors.
hepatitis C virus NS5A;3D-QSAR;CoMFA;CoMSIA
R914.2
A
1674-8425(2015)05-0052-09
10.3969/j.issn.1674-8425(z).2015.05.010
2015-03-06
国家自然科学基金资助项目(81171508);重庆市自然科学基金重点项目(CSTC2013JJB10004);重庆理工大学研究生创新基金资助项目(YCX2013222)
孟令鑫(1987—),女,青海西宁人,硕士研究生,主要从事药物设计与合成研究。
孟令鑫,刘蒙蒙,王远强,等.丙型肝炎病毒NS5A抑制剂的3D-QSAR研究[J].重庆理工大学学报:自然科学版,2015(5):52-60.
format:MENG Ling-xin,LIUMeng-meng,WANG Yuan-qiang,etal.3D-QSAR Studies of Hepatitis CVirus NS5A Inhibitors[J].Journal of Chongqing University of Technology:Natural Science,2015(5):52-60.

