侧枝萌发诱导基因(LIF)的原核表达与农杆菌介导转化麻风树幼
叶宗桦等


摘要:麻风树(Jatropha curcas L.)的有限分枝是限制麻风树油产量的最主要因素之一,为了奠定基因调控麻风树分枝的基础,研究并优化了侧枝萌发诱导基因LIF(lateral shoot-inducing factor )的原核表达条件,并成功获得了重组蛋白。通过研究农杆菌浓度、抗生素浓度建立了稳固高效的麻风树农杆菌((LBA4404)高效转化体系;将LIF基因转入麻风树基因组中,通过GUS染色、PCR、Southern blot检测,确定了LIF基因已经成功转化入麻风树基因组中,获得了高达(23.91±5.78)%的转化率。
关键词:麻风树;侧枝萌发诱导基因(LIF);幼叶;农杆菌
中图分类号:Q943.2 文献标志码: A文章编号:1002-1302(2015)09-0014-06
收稿日期:2014-08-28
基金项目:国家自然科学基金(编号:51308464);西南交通大学科技创新项目(编号:A0920502051405-62)。
作者简介:宗桦(1981—),女,江苏宜兴人,博士,讲师,主要从事植物学研究。E-mail:14744632@qq.com。属于大戟科的麻风树(Jatropha curcas L.)是一类具有极强抗旱性的大灌木或小乔木,其种子中含油丰富,并能较容易地转化成纯度能满足美国和欧洲标准的生物柴油[1]。此外,麻风树油还可应用于制造肥皂、化妆品、染料等产品[2-4]。然而,由于麻风树的种子产量较低,极大地限制了麻风树油的推广应用。迄今为止,提高麻风树种子的产量仍是一件十分困难的事情,这是由于麻风树的种子产量受到多种因素影响,如自然环境[5]、麻风树的分枝形态[6]、基因型[7]、经营管理方法[8]等。在这些因素当中,麻风树结果母枝的分枝形态被认为是最重要的制约因素之一,因此许多学者认为增加麻风树结果母枝的数量就能有效地提高麻风树种子的产量[8-9]。
近年来,世界各国都开始关注植物分枝发育控制机理的研究,通过采用包括突变体分离、定位、克隆相关基因和相关基因分析等分子生物技术等方法,不断加深对植物分枝发育的认识。目前在许多物种,如矮牵牛[10-11]、烟草[12]、番茄、豌豆、玉米、水稻和拟南芥[13]中都开展了突变体作为分枝的变化模式研究。2005年,Nakagawa等从矮牵牛中克隆出了侧枝萌发诱导基因LIF,是目前唯一一个被报道的分枝基因[14]。
Nakagawa等研究发现,LIF基因在矮牵牛、烟草、拟南芥中过量表达均可以导致植株分枝明显增多,高度明显降低,叶片小化[14],由此Nakagawa等推断,LIF基因在双子叶植物中能保守地表达,可以调控双子叶植物的分枝[14]。进一步研究发现,LIF编码的蛋白属于一种TFⅢA型锌指蛋白,具有这种类型结构域的蛋白一般是结合DNA作为转录因子参于多种植物的调控过程。然而由于LIF被发现的时间短,因此针对LIF诱导的蛋白的功能研究还未开展。再加上植物分枝的形成是一个受多种因素影响的复杂的生物学现象,因此,针对单个基因开展蛋白纯化研究对于了解其在植物形态构成上的功能有一定的难度。而通过重组的方式在大肠杆菌中进行原核表达为LIF基因的功能研究提供了一个良好的途径。众所周知,大肠杆菌是现代分子生物学研究中最常用的材料之一,目前除了己经掌握了它的分子生物学和分子遗传学方面的大量资料外,在基因表达方面也有深入研究。大肠杆菌遗传背景清楚,容易培养,能大规模发酵,并具有大量可供选择利用的克隆和高效表达载体[15]。
本研究构建了LIF原核表达载体,转入大肠杆菌BL21中,将转化子进行诱导表达,获得重组蛋白,摸索表达的最优条件,为进一步探讨LIF的功能奠定基础。同时,将LIF基因转入麻风树基因组,了解LIF基因在麻风树中的表达方式,为基因调控麻风树的分枝做好准备工作。
1材料与方法
1.1植物材料和菌株
试验所用矮牵牛(Petunia hybrida Vilm)种苗种植在温室(14 h光照/10 h黑暗,室温22 ℃)中。成熟麻风树种子来源于攀枝花市。种苗种植于盛满沙和营养土的塑料盆钵中,于温室中生长2个月(16 h光照/8 h黑暗,室温28 ℃)后,取第1~3节上的叶片用作试验。灭菌后的麻风树幼叶切成为 1 cm2 的小块,接入愈伤组织诱导培养基中预培养2 d。
Escherichia coli菌株BL21、农杆菌菌株LBA4404均为笔者所在实验室保存。
1.2矮牵牛LIF基因开放阅读框获得和原核表达载体构建
1.2.1LIF基因ORF的扩增提取矮牵牛的RNA,用Oligo (dT)18为引物进行反转录,反转录产物作为模板,设计引物lof33、lof52进行PCR扩增ORF序列。酶切位点(下划线部分)分别为BamHⅠ、HindⅢ。引物序列如下:lof33:5′-CCCAAGCTTTCATAAGAACTTTCTTGTGCCTAAACC-3′;lof52:5 ′-CGCGGATCCATGGAAACTAGTAAAAATCAGCCGTC-3′;反转录引物:Oligo (dT)18:5′-GCTGTCAACGATACGCTACGTAACGGCATGACAGTG (T)18-3′。反应条件为:95 ℃预变性5 min;94 ℃ 40 s,62 ℃ 40 s,72 ℃ 1 min,34个循环;72 ℃延伸10 min。将PCR产物电泳后进行回收,回收按照DNA胶回收试剂盒的说明步骤进行。
1.2.2原核表达载体的构建原核表达所用质粒载体选用pET32[16],含有T7启动子、6个His标签,具有Amp抗性。将胶回收的片段连接到pMD19-T载体上,连接产物转化大肠杆菌DH5α感受态获得转化子pMD19-T-LIF,摇菌1 h后涂LB平板,37 ℃过夜培养。于第2天挑菌,摇菌后送样测序。经测序检测获得LIF的ORF片段后,摇菌提质粒。用限制性内切酶BamHⅠ、HindⅢ双酶切载体pET32,胶回收线性载体。用T4连接酶连接目的片段与线性载体(均带有BamHⅠ、HindⅢ黏性末端)。16 ℃连接过夜,获得重组子pET-LIF (简称PL)。随后将重组子转入大肠杆菌BL21感受态细胞获得转化子PLB,摇菌后提取质粒,采用酶切、菌落PCR和测序等手段检验目的片段是否正确。同时也将空载pET32用同样的方法转入大肠杆菌BL21以获得空白对照转化子pET32-Empty (简称PE)。引物均由上海英骏生物技术有限公司合成。
1.3PLB的诱导表达
挑取重组菌PLB、PE分别接种到15 mL的AMP-LB液体培养基中,放置于37 ℃下振荡培养12 h。再从过夜培养物种各取500 μL菌液接种于新鲜的10 mL AMP-LB液体培养基中,37 ℃下振荡培养直到菌液的D600 nm= 0.6~0.7。随后将PLB菌液均分后放置于不同的诱导温度、诱导时间和添加不同浓度的IPTG中进行诱导(表1)。
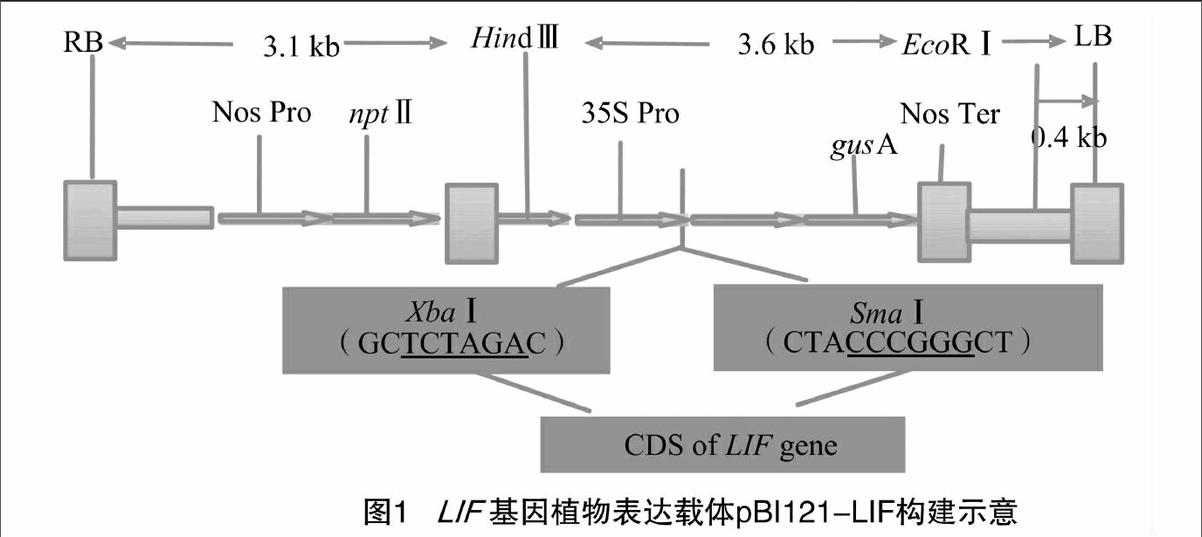
1.3矮牵牛LIF基因蛋白编码序列 (coding sequence,CDS)和植物表达载体的构建
1.3.1CDS的扩增首先在GenBank搜索出矮牵牛LIF基因的编码蛋白(accession number:AB035093),根据其蛋白序列设计出C1、C2引物序列用于扩增矮牵牛CDS,酶切位点分别为XbaⅠ、SmaⅠ酶切位点(酶切位点见下划线)。引物序
1.3.2植物表达载体的构建将LIF基因的CDS插入表达载体pBI121[17]中,LIF基因的插入位置见图1,获得重组载体pBI121-LIF。pBI121载体含有Npt Ⅱ基因,抗卡拉霉素(Kan);含GUS基因作为报告基因;含CaMV35S启动子(笔者所在实验室保存)。提取重组子pBI121-LIF质粒后,用反复冻融法[18]将其转化入农杆菌LBA4404[19]感受态细胞中,同时也将空载pBI121以同样的方法转入2种农杆菌感受态中以获得空白对照,获得转化子pBI121-Empty (简称PBE)。检验后的含有LIF基因的农杆菌单菌落接种到1 mL YEB液体培养基中,培养基中加入50 μg/mL链霉素(Str )、100 μg/mL 利福平(Rif)、100 μg/mL Kan,于恒温摇床上 27 ℃、200 r/min摇培过夜。取出500 μL培养过夜的菌液转入新配制的有同样抗生素的YEB液体培养基中,培养直到D600 nm=1.0时,用新鲜的MS液体培养基(pH值5.4~5.8)分别稀释为D600 nm=0.1~0.5用于转化。
1.4培养基成分和培养条件
试验中外植体培养均选用MS培养基[20]为基础培养基,其中蔗糖质量浓度为3%,琼脂质量浓度为0.8%,pH值为58。外植体的培养均在28 ℃的温室中进行,并保持16 h光照、8 h黑暗交替。具体培养基配方如下[21]:愈伤组织诱导培养基(CI):MS+2.0 mg/L 6-BA+0.05 mg/L 2,4-D;愈伤组织再生培养基(PR):MS+0.5 mg/L 6-BA+0.5 mg/L KT+0.1 mg/L IBA+0.1 mg/L GA3;生根培养基(RM):MS+02 mg/L IBA;大肠杆菌LB培养基:10 g/L胰蛋白胨,5 g/L酵母提取物,10 g/L NaCl,pH值7.0。固体LB培养基在以上成分中添加15 g/L琼脂粉。
1.5外植体的转化和选择培养
预培养之后的幼叶外植体浸入30 mL含LIF基因的农杆菌菌液体中,在28 ℃、200 r/min的摇床上侵染10 min。用干燥的无菌滤纸吸去多余菌液后将外植体转移到CI培养基中,在全暗的温室(28 ℃)中共培养2~7 d。随后,将外植体转入加入了300 mg/L头孢霉素(Cef)的新鲜CI培养基中,继续暗培养3周。待愈伤组织产生后,马上转入加入了Kan(0、10、20、30、40 mg/L)、150 mg/L Cef的新鲜PR培养基中诱导抗性丛生芽。经过4周的筛选后,将抗Kan(KanR)的麻风树再生单芽切下并转入加入Kan的RM生根培养基中。经3~4周诱导后,将生根的植株移入放置于温室中的培养钵中,培养钵中加入珍珠岩 ∶沙 ∶土=1 ∶1 ∶1(体积比)的混合基质中练苗2周后移入大田。
1.6转基因植株的鉴别
GUS染色:将共培养后的叶盘、愈伤组织和抗Kan的丛生苗切成小块后放在1.5 mL的离心管中,加入GUS染液,37 ℃ 水浴16 h[22]。植株材料用70%的乙醇脱色2~3次,在解剖镜和显微镜下进行观察和拍照。
PCR检测:采用CTAB法[23]提取转基因麻风树的DNA,设计引物扩增检验是否获得转基因植株。首先设计了2条引物:qc1:5′-ACCGTCGGGCAAAGACAGATT-3′;qc2:5′-GACCGCATCGAAACGCAGCAC-3′,预期扩增的长度为 538 bp。扩增序列中包含LIF基因中246 bp的序列和GUS基因中长度为292 bp的序列,PCR反应参数为:95 ℃预变性5 min;94 ℃30 s,62 ℃40 s,72 ℃ 1 min,34个循环;72 ℃延伸10 min。
Southern blot检测:为了进一步确定LIF基因已经插入麻风树基因组,先从PCR检测呈阳性 (PCR+)的植株中提出DNA(20 μg),随后用EcoRⅠ、HindⅢ于37 ℃酶切过夜。EcoRⅠ、HindⅢ在质粒pBI121上各仅有1个酶切位点。以重组质粒pBI121-LIF为模板用引物qc1、qc2扩增LIF基因的ORF,按照PCR Digoxigenin (DIG) Probe Synthesis Kit(Roche Diagnostics)说明制作探针。杂交的具体操作按照Raffeiner的报道[24]进行,3 d后进行拍照(Kodak 8-10 X-Omat AR,Rochester)。
1.7数据分析
研究中用到的相关计算公式如下:
存活率=有分化能力的幼叶外植体数量/外植体总数×100%;
GUS的瞬时表达率=GUS显阳性(GUS+)的存活外植体数量/外植体总数×100%。
所有的数据均使用SPSS统计软件(ver.11.0)进行Duncans多重分析比较。
2结果与分析
2.1原核表达载体的获得
以反转录的cDNA为模板,用引物lof33、lof52进行PCR扩增LIF基因的ORF序列,获得了1条650 bp的片段(图2-A),胶回收后与载体pMD19-T相连接,并转入DH5α感受态获得转化子pMD19-T-LIF;提取pMD19-T-LIF转化子质粒双酶切后的片段(650 bp),与BamHⅠ、HindⅢ双酶切后的pET32载体连接,获得重组子pET-LIF;将连接后的产物转入BL21感受态,经过菌落PCR、双酶切(图2-B)和测序验证,证明获得了转化子PLB,原核表达载体PLB构建成功。
2.2PLB诱导表达条件的优化研究
通过图3的SDS-PAGE分析,比较得出了在不同的诱导条件下重组菌的最佳表达条件,并成功诱导重组蛋白的表达。在不同的诱导温度梯度下,PLB均比对照多出1条约为43 ku的目的条带;当诱导温度为设置为37 ℃时,目的条带十分明显;当诱导温度为30 ℃时,也能看到目的条带,但比37 ℃稍弱;20 ℃下诱导的PLB表达最弱。在不同的诱导时间梯度下,PLB都比对照多出1条约为43 ku的目的条带,说明在2、4、6 h 3个处理时间内,PLB均有表达。但3个处理时间下的诱导表达量有所不同,诱导2 h的条带最弱;而诱导4、6 h的条带都很亮,说明这2个时间内的融合蛋白诱导表达最强;随着时间从4 h延长到6 h,表达量没有随之增加,表明诱导4 h为最佳收获时间。如果菌体过度培养,一方面由于Amp逐渐失活,失去选择压后质粒快速丢失,不能增加表达产量; 另一
方面由于菌体老化和代谢产物的影响,菌体不但不继续生长,反而会死亡、分解,使目的蛋白丢失[25]。由图3-C可以看出,只要加入了IPTG,PLB就能产生大小约为43 ku的重组蛋白。不同浓度的IPTG对PLB的诱导效果有差异,其中IPTG为0.120 g/L时诱导的重组蛋白量最大,其次是0.240 g/L,0.024 g/L诱导PLB表达的量最少,无IPTG时无重组蛋白产生。根据表达载体pET32的分子量、LIF基因ORF成熟肽分子量的估算,融合蛋白的总分子量应为43 ku。
2.3农杆菌侵染最佳浓度
本研究发现,菌液浓度是影响转化效率的关键因素。由表2可见,当用D600 nm=0.1的LBA4404菌液侵染后,愈伤组织产生了数量最多的蓝斑(GUS+);随着浓度的升高(D600 nm>0.1),多余的菌体改变了培养基的pH值,并抑制了愈伤组织的产生,严重制约转化效率。这一现象与Yong等的研究结论相一致,他们认为高浓度的农杆菌液反而会产生较低的转化效率[26-27]。因此,本研究结果表明,D600 nm=0.1的农杆菌LBA4404最适于麻风树幼叶转化。
2.4Kan的筛选效果
在许多木本植物的转化过程中,Kan都被用于筛选转基因植株[28-31],Kan的敏感性取决于植物种类、外植体类型[32]。本研究表明,不同的浓度梯度(20、30、40 mg/L)的Kan均能筛选出PCR检验呈阳性(PCR+)的植株(表3)。虽然40 mg/L Kan对愈伤组织再生有一定抑制作用,但只有高浓度的Kan才能完全抑制未转化丛生芽的再生。相比之下,在加入20、30 mg/L Kan的培养基中会有部分未转化的丛生芽产生,称之为“逃离”。因此,本研究表明40 mg/L是Kan用于筛选的最佳浓度。Kan能有效地筛选出麻风树转基因植株,与Li等提出的Kan不适于用于麻风树的筛选的结论相悖[33]。培养4周后,待抗Kan的丛生芽(图4-C)长至2~3 cm 高时,转入加入40 mg/L Kan的RM培养基中诱导生根(图4-D)。用Kan持续筛选能有效抑制未转化植株的逃离,持续筛选非常必要[34]。培养1个月后,生根的植株移入培养钵中进行2周的炼苗(图4-E)。
2.5GUS 染色分析
为了确认转化子,诱导出的愈伤组织和KanR再生植株都需要进行GUS染色。经GUS染色后,发现有68%的愈伤组织出现蓝斑,表明呈现GUS+(表2)。愈伤培养4周后, 有35%的GUS+愈伤组织分化出KanR丛生芽。用40 mg/L Kan筛选出的所有植株都呈现GUS+(表3)。此外,笔者发现KanR植株的蓝斑都沿着茎节和叶柄分布(图5-e至图5-h),在愈伤组织上则是随机分布(图5-b),在根系中无活性。这一现象与Nakagawa等在LIF基因超表达的矮牵牛中GUS蓝斑的分布趋势一致[14]。Nakagawa等推断,LIF基因可能参与了矮牵牛叶腋处的细胞分裂素的产生过程。此外,本研究在对照植株中未观察到GUS蓝斑(图5-c、图5-d)。
2.6PCR和Southern检测
GUS+再生苗首先采用PCR法一步验证基因转入的真实性。PCR检测提取了转基因植株的DNA为模板,所以消除了残留菌株的干扰,比GUS染色更可靠。由图6-A可以看出,随机抽取的GUS+再生苗均扩增出了预期0.54 kb大小片段,未转基因的对照植株无任何条带出现。通过PCR检测证明,201个叶盘在经过3个月的转化后,有48个产生了转基因植株,所占比例达到(23.91±5.78)%(表3),转化效率明显高于Li等报道的13%的转化率[34]。随后,根据PCR的检测结果,进一步使用Southern Blot最终检测转基因植株。本研究随机抽取了12个PCR+植株以GUS基因的部分片段为探针进行Southern杂交,预期的杂交片段约为3.6 kb, 图6-B显
示了3株PCR+植株的杂交结果,均获得了预期杂交带。未转基因的对照无任何条带产生,说明载体c重组载体pBI121-LIF已经成功转化入麻风树基因组DNA中。植株1产生了7条杂交带,说明基因组中插入了多个拷贝,其中有2个条带的长度明显>3.6 kb,推测可能有完整的T-DNA拷贝插入;同时观察到有4条杂交带<3.6 kb,说明T-DNA有部分插入的拷贝存在,T-DNA的部分插入是转基因过程中的常见现象。植株2、植株3均只有1条3.6 kb的杂交带,说明转化为单拷贝,T-DNA也是部分插入。不同数目的拷贝证明植株为独立个体。
3结论与讨论
植物分枝发育在植物形态建成中具有非常重要的地位,植物的分枝是控制作物结实并进而影响最终产量的重要农艺性状之一,分枝多的植株结实部位多,但影响群体通风透光状况,分枝与主茎的夹角直接影响群体透光性和对光能的利用。然而,由于分枝的基因调控影响因素众多,且机理复杂,因此目前仅处于探索阶段。LIF基因由于发现时间较晚,其具体功能和作用机理仍未明确。目前只有1例进行LIF类锌指蛋白的功能研究,即通过洋葱表皮细胞的瞬时表达GFP-LIF-GUS蛋白的研究表明LIF基因被转运到细胞核中,为证实LIF是1种转录因子提供了有力的证据[14]。本试验采用pET32为表达载体,构建了LIF基因原核表达载体,并将转化子进行诱导表达获得了重组蛋白,分析得出了LIF所对应蛋白表达的最佳条件,为进一步探讨LIF的功能奠定了基础。
麻风树作为一种能源植物,已经引起了世界各国的高度关注。但由于其为木本植物,目前大部分的研究成果都集中在麻风树再生体系的研究上。本研究以麻风树为幼叶研究对象,通过摸索Kan浓度成功建立了高效稳固的农杆菌转化体系。与前人的报道(针对麻风树子叶的转化体系)相比,本体系不仅转化效率高、稳定性强,而且在植物材料的选择上更经济、节约,本转化体系的成功建立为研究者大规模的开展麻风树各类基因的研究创造便利。在此基础上,本研究将LIF基因顺利转入麻风树基因组中,由于LIF基因能在双子叶植物中保守表达,因此具有很强的应用范围。将LIF转入麻风树中不仅有助于进一步了解LIF基因的作用原理和表达方式,还能为今后开展麻风树分枝的基因调控工作奠定坚实的基础。在今后的研究中,将在田间持续观察转基因苗的分枝能力并计算种子产量。
参考文献:
[1]Alok-Kumar T,Akhilesh K,Hiflur R[J]. Biodiesel production from Jatropha curcas with high free fatty acids:an optimized process[J]. Biomass and Bioenergy,2007,31(8):569-575.
[2]Kandpal J B,Madan M. Jatropha curcus:a renewable source of energy for meeting future energy needs[J]. Renewable Energy,1995,6(2):159-160.
[3]Adebowale K O,Adedire C O. Chemical composition and insecticidal properties of the underutilized Jatropha curcas seed oil[J]. African Journal of Biotechnology,2006,5(10):901-906.
[4]Mazumdar P,Basu A,Paul A,et al. Age and orientation of the cotyledonary leaf explants determine the efficiency of de novo plant regeneration and Agrobacterium tumefaciens-mediated transformation in Jatropha curcas L.[J]. South African Journal of Botany,2010,76(2):337-344.
[5]Openshaw K. A review of Jatropha curcas:an oil plant of unfulfilled promise[J]. Biomass and Bioenergy,2000,19(1):1-15.
[6]Achten W M J,Verchot L,Franken Y J,et al. Jatropha bio-diesel production and use[J]. Biomass and Bioenergy,2008,32(12):1063-1084.
[7]Ginwal H S,Rawat P S,Srivastava R L. Seed source variation in growth performance and oil yield of Jatropha curcas Linn. in central India[J]. Silvae Genetica,2004,53(4):186-192.
[8]Gour V K. Production practices including post-harvest management of J. curcas[C]//Singh B,Swaminathan R,Ponraj V,et al. Rashtrapati Bhawan,New Delhi,India:Biodiesel conference toward energy independence-focus of Jatropha,2006:223-251.
[9]Abdelgadir H A,Johnson S D,van Staden J. Promoting branching of a potential biofuel crop Jatropha curcas L.by foliar application of plant growth regulators[J]. Plant Growth Regulation,2009,58(3):287-295.
[10]Bennett T,Leyser O. Something on the side:axillary meristems and plant development[J]. Plant Molecular Biology,2006,60(6):843-854.
[11]Lewis J M,Mackintosh C A,Shin S A,et al. Overexpression of the maize Teosinte Branched 1 gene in wheat suppresses tiller development[J]. Plant Cell Reports,2008,27(7):1217-1225.
[12]Bereterbide A,Hernould M,Castera S,et al. Inhibition of cell proliferation,cell expansion and differentiation by the Arabidopsis SUPERMAN gene in transgenic tobacco plants[J]. Planta,2001,214(1):22-29.
[13]Mcsteen P,Leyser O. Shoot branching[J]. Annual Review of Plant Biology,2005(56):353-374.
[14]Nakagawa H,Jiang C J,Sakakibara H,et al. Overexpression of a petunia zinc-finger gene alters cytokinin metabolism and plant forms[J]. The Plant Journal,2005,41(4):512-523.
[15]Fischer B,Sumner I,Goodenough P. Isolation and renaturation of bioactive proteins expressed in Eschrichia coil as inclusion bodies[J]. Arzneimittelforschung,1992,42(12):1512-1515.
[16]LaVallie E R,DiBlasio E A,Kovacic S,et al. A thioredoxin gene fusion expression system that circumvents inclusion body formation in the E. coli cytoplasm[J]. Biotechnology,1993,11(2):187-193.
[17]Hatanaka T,Choi Y E,Kusano T,et al. Transgenic plants of coffee Coffea canephora from embryogenic callus via Agrobacterium tumefaciens-mediated transformation[J]. Plant Cell Reports,1999,19(2):106-110.
[18]Holsters M,De Waele D,Depicker A,et al. Transfection and transformation of Agrobacterium tumefaciens[J]. Molecular & General Genetics,1978,163(2):181-187.
[19]Hoekema A,Hirsch P R,Hooykaas P J J,et al. A binary plant vector strategy based on separation of vir-and T-region of the Agrobacterium tumefaciens Ti-plasmid[J]. Nature,1983,303(5913):179-180.
[20]Murashige T,Skoog F. A revised medium for rapid growth and bioassays with tobacco tissue cultures[J]. Physiologia Plantarum,1962,15(3):473-479.
[21]宗桦,王盛华,邓新林,等. 麻疯树叶片高效再生体系与不定芽起源[J]. 应用与环境生物学报,2010,16(6):789-793.
[22]Dutta I,Majumder P,Saha P,et al. Constitutive and phloem specific expression of Allium sativum leaf agglutinin (ASAL) to engineer aphid (Lipaphis erysimi) resistance in transgenic Indian mustard (Brassica juncea)[J]. Plant Sci,2005(169):996-1007.
[23]Murray M G,Thompson W F. Rapid isolation of high molecular weight plant DNA[J]. Nucleic Acids Research,1980,8(19):4321-4325.
[24]Raffeiner B,Serek M,Winkelmann T.Agrobacterium tumefaciens-mediated transformation of Oncidium and Odontoglossum orchid species with the ethylene receptor mutant gene etr1-1[J]. Plant Cell,Tiss Organ Cult,2009,98(2):125-134.
[25]刘永庆,陈溥言,杜念兴,等. 生长抑素(SS)基因在大肠杆菌pT7ZZ表达系统中的克隆与表达[J]. 中国生物制品学杂志,2002,15(1):17-20.
[26]Yong W T L,Abdullah J O,Mahmood M. Optimization of Agrobacterium-mediated transformation parameters for Melastomataceae spp. using green fluorescent protein(GFP)as a reporter[J]. Scientia Horticulturae,2006,109(1):78-85.
[27]Xu,L,Najeeb U,et al. Establishment of Agrobacterium-mediated BT gene transformation system mat rush (Juncus effusus L.)[J]. Pakistan J. Bot,2009,41(5):2615-2624.
[28]Prakash M G,Gurumurthi K. Genetic transformation and regeneration of transgenic plants from precultured cotyledon and hypocotyl explants of Eucalyptus tereticornis Sm. using Agrobacterium tumefaciens[J]. In Vitro Cellular & Developmental Biology-Plant,2009,45(4):429-434.
[29]James D J,Passey A J,Webster A D,et al. Transgenic apples and strawberries:advances in transformation,introduction of genes for insect resistance and field studies of tissue cultured plants[J]. Acta Horticulturae,1993(336):179-184.
[30]Fillatti J A,Sellmer J,Mccown B,et al. Agrobacterium-mediated transformation and regeneration of Populus[J]. Molecular & General Genetics:MGG,1987,206(2):192-199.
[31]McGranahan G H,Leslie C A,Uratsu S L,et al. Agrobacterium-mediated transformation of walnut somatic embryos and regeneration of transgenic plants[J]. Nature Biotechnology,1988(6):800-804.
[32]Klimaszewska K,Lachance D,Pelletier G,et al. Regeneration of transgenic Picea glauca,P. Mariana,and P.Abies after cocultivation of embryogenic tissue with Agrobacterium tumefaciens[J]. In Vitro & Cell Developmental Biology-Plant,2001,37(6):748-755.
[33]Li M R,Li H Q,Jiang H W,et al. Establishment of an Agrobacteriuim-mediated cotyledon disc transformation method for jatropha curcas[J]. Plant Cell Tissue and Organ Culture,2008,92(2):173-181.
[34]Athmaram T N,Bail G,Devaiah K M. Integration and expression of Bluetongue VP2 gene in somatic embryos of peanut through particle bombardment method[J]. Vaccine,2006,24(15):2994-3000.

