污水处理系统中硝化菌的菌群结构和动态变化
曾 薇,张丽敏,王安其,张 洁,彭永臻,段俊岭(.北京工业大学环境与能源工程学院,北京 004;.北京城市排水集团有限责任公司,北京 00044)
污水处理系统中硝化菌的菌群结构和动态变化
曾 薇1*,张丽敏1,王安其1,张 洁1,彭永臻1,段俊岭2(1.北京工业大学环境与能源工程学院,北京 100124;2.北京城市排水集团有限责任公司,北京 100044)
研究分析了4种不同工艺类型的城市污水处理厂中氨氧化细菌(AOB)和亚硝酸盐氧化细菌(NOB)的丰度及菌群结构.实时定量PCR结果表明4种工艺中AOB菌群的丰度范围为8.56×106~4.46×107cells/gMLSS;NOB菌群的丰度为3.37×108~1.53×109cells/gMLSS.每个工艺中Nitrospira都是优势NOB,占NOB菌群的88% 以上. A2O工艺冬季AOB和Nitrospira丰度比夏季均有所降低,这是导致冬季生物脱氮效果变差的主要原因.基于 amoA基因的系统发育分析结果显示所有的序列属于Nitrosomonas,其中Nitrosomonas oligotropha cluster 占克隆文库的60.1%,是 AOB 种群中的优势菌属,Nitrosomonas-like cluster和 Nitrosomonas europaea cluster次之,分别占克隆文库的29.6%和9.1%.N. europaea cluster只在A2O工艺中出现,且在A2O工艺夏季污泥样品克隆文库中达到44.7%.低DO运行使N. europaea cluster成为优势 AOB是 A2O工艺夏季出现较高亚硝酸盐积累率的主要原因.研究结果证实了城市污水处理厂中优势 AOB和 NOB分别为Nitrosomonas和Nitrospira,硝化菌群占总菌群的1%~7%,其丰度、相对含量和菌群结构是影响硝化效果的主要因素.
氨氧化细菌(AOB);亚硝酸盐氧化细菌(NOB);城市污水处理厂(WWTPs);实时定量PCR (QPCR);amoA基因
生物脱氮已广泛应用于城市污水处理厂,硝化作用是生物脱氮的首要环节,其功能微生物包括将氧化为的氨氧化细菌(AOB)和将氧化为的亚硝酸盐氧化细菌(NOB)[1].AOB菌群主要属于变形菌纲的β-Proteobacteria和γ-Proteobacteria两个亚纲[2],其中β亚纲分为亚硝化单胞菌群(Nitrosomonas)和亚硝化螺菌群(Nitrosospira).NOB主要有硝化杆菌属(Nitrobacter)、硝化螺菌属(Nitrospira)、硝化刺菌属(Nitrospina)及硝化球菌属(Nitrococus)[3].硝化菌群的丰度和菌群结构直接影响污水处理厂的硝化效果.
近年来,不依赖于纯培养的分子生物学分析方法成为研究污水处理系统硝化菌群的主要技术[4-6].已有研究证实绝大多数的生物反应器里Nitrosomonas是AOB中的优势菌属,而Nitrosospira只出现在个别反应器里[2,7-8].对于NOB菌群,以前一直认为Nitrobacter是污水生物处理系统中亚硝酸盐氧化的主导者[9].也有研究者认为自然环境中的 NOB以 Nitrobacter和Nitrospira类型为主,其中 Nitrobacter是土壤中NOB的主导类型,而Nitrospira在污水生物处理系统中分布较为广泛[10].后来的一些研究表明Nitrospira和Nitrobacter均存在于城市污水处理厂[11],但是发现 Nitrospira 在污水生物处理系统中更为常见且数量高于Nitrobacter[12].由此可见,城市污水处理系统中 NOB 的优势菌群因工艺类型和运行参数的不同而具有各自的特征.很多实际污水处理系统中的硝化细菌对环境因素以及工艺参数非常敏感,例如温度、DO、水力停留时间(HRT)以及抑制剂等因素均可以影响硝化细菌的组成[13-15].以往对硝化菌群的定量分析主要集中AOB菌群的研究上[16],关于NOB的定量分析,尤其是 NOB的两个亚属 Nitrospira和Nitrobacter的定量研究非常有限.本研究通过对不同工艺的城市污水处理厂中AOB、NOB的定量分析,考察工艺类型及运行条件对 AOB和NOB(Nitrospira和Nitrobacter)丰度的影响.
基于16S rRNA和amoA基因的现代分子生物学方法为复杂环境中AOB的多样性以及菌群结构分析提供了有力的手段[17-19].氨单加氧酶(Ammonia monooxygenase, AMO)是氨氧化菌所特有的一种胞内酶,由amoA、amoB和amoC 3个亚基组成,其中 amoA的基因产物含有该酶的活性位点.很多学者对不同AOB菌株的amoA基因和16S rRNA基因进行测序和系统发育树分析后,发现大部分AOB在分别基于amoA基因和16S rRNA基因的系统发育树上的分类有着高度的相似性[19-21],但 amoA基因类引物的扩增特异性更强,对 AOB菌群遗传差异的分辨能力更高[18,22].本研究基于amoA基因建立系统发育树,能够将AOB菌群进行更为细致精确的分类.
本研究选择具有代表性的大型市政污水处理厂,分析不同工艺的城市污水处理厂活性污泥中AOB和NOB菌群结构和代谢活性的差异,揭示污水处理厂AOB和NOB菌群结构、丰度与工艺运行的相关性.
1 材料与方法
1.1城市污水处理厂的活性污泥样品
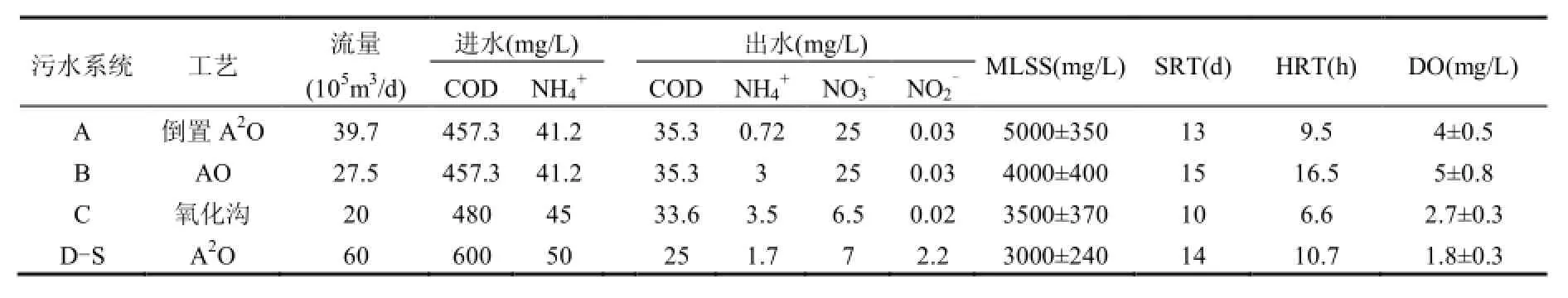
表1 4个污水处理系统的进出水水质指标及运行参数Table 1 Influent and effluent characteristics and operation parameters of four WWTPs
实验所用的 5个污泥样品均取自二沉池回流污泥,命名为: A、B、C、D-S(夏季样品)、D-W(冬季样品).A、B、C、D-S取样时间为夏季7月,D-W为冬季2月份.A和B取自同一水厂不同工艺:A是倒置A2O工艺,缺氧段位于前端,优先考虑脱氮效果,即:缺氧-厌氧-好氧方式运行;B是AO工艺,采用缺氧-好氧技术;C取自卡鲁赛尔氧化沟工艺;D-S和D-W取自同一工艺的夏季和冬季,工艺为传统的A2O工艺,即厌氧-缺氧-好氧.各个工艺进出水水质指标及运行参数见表1.
1.2DNA提取、PCR、克隆测序
用1×PBS清洗污泥样品3次,14000×g离心2min(离心机:MIKRO 22 R,德国Hettich),去除上清液,置于-20℃保存.采用试剂盒(Fast DNA Spin kit for soil, MP, USA)对DNA进行提取,提取后的DNA 通过 Nanodrop Spectrophotometer ND-1000 (Thermo Fisher Scientific, USA)测量核酸浓度及纯度.
PCR 反应采用试剂盒(Promega GoTaq Green Master Mix, USA),反应体系为 25μL:12.5μL GoTaq Green Master Mix,1μL (10mmol/L)正向引物,1μL (10mmol/L)反向引物,0.5~2μL DNA模板,一定量的ddH2O.PCR程序见表2.

表2 PCR扩增程序及特异性引物Table 2 Specific primers and PCR programs
amoA的 PCR产物经琼脂糖凝胶电泳(Agarose MS-6,TaKaRa,Japan)检测,为单一的目的条带,切胶,用纯化试剂盒(Agarose Gel DNA Purification Kit Ver. 2.0TaKaRa, Japan )进行纯化.纯化后的 DNA用试剂盒(Zero Background TA Topoisomerase Cloning Kit, Clonesmarter, USA)进行连接转化,连接体系为 10μL,包括 1μL pCloneEZ-TOPO 载体,1μL 10×Enhancer, 0.5~8μLDNA,一定量的用 ddH2O.连接反应完成后将产物加入到感受态细胞DH5a(中美泰和,国产)中进行转化.每个样品随机挑出50个amoA基因的阳性克隆子进行测序,构建克隆文库.
1.3克隆文库建立和系统发育分析
构建文库的序列通过Mothur软件按照97%相似度进行OTU划分,将每个OTU的代表序列与NCBI数据库中利用BLAST下载的相似性最高最具代表性的菌株序列一起进行比对.采用MEGA5.0利用邻接法(Neighbor joining method)进行系统发育分析,通过自举分析方法(Bootstrap)检验系统发育树各分支置信度,重复1000次.
1.4实时定量PCR (QPCR)
采用特异性引物对AOB amoA功能基因,隶属于NOB菌群的Nitrospira和Nitrobacter以及全菌的 16S rRNA进行 QPCR扩增.反应在Mx3005P实时定量PCR扩增仪(Agilent Technologies,American)上进行,采用试剂盒( SYBR Premix Ex Taq kit,TaKaRa,Japan)进行反应,体系为25μL包括12.5μL的SYBR缓冲液,正反向引物各 1μL(10mmol/L),0.5μL ROX,DNA模板 2μL,一定量的 ddH2O.采用试剂盒(MiniBEST Plasmid Purification Kit Ver.4.0,TaKaRa,Japan)回收质粒.
标准曲线的建立:用 NanoDrop ND-1000(Thermo,American)分光光度计测定回收质粒的浓度(MiniBEST Plasmid Purification Kit Ver.4.0,TaKaRa,Japan).将纯化后的质粒以10倍的浓度梯度稀释用作QPCR标准品.反应在Mx3005P实时定量PCR扩增仪(Agilent Technologies,American)上进行,采用试剂盒(SYBR Premix Ex Taq kit,TaKaRa,Japan)进行反应,体系为25μL.
1.5登录号
本研究所测得的AOB序列上传至GenBank数据库,AOB的序列登录号为:KR018127-KR018368.
2 结果与讨论
2.1AOB与NOB的实时荧光定量PCR分析
AOB、NOB (Nitrobacter和Nitrospira) 和总菌的实时荧光定量PCR的标准曲线的效率都在90%~110%之间,相关系数均大于0.998,特异性和扩增效率都符合精确定量的要求.标准品分别如下:AOB 为 8.55×101~8.55×108拷贝,Nitrobacter为1.05×102~1.05×109拷贝, Nitrospira 为 1.31×102~1.31×109拷贝,总菌为 4.98×101~4.98×108拷贝.
表3为AOB和NOB绝对定量和相对定量的结果.5个污泥样品中总菌丰度在一个数量级上,定量范围为 1.10×1010(B)~9.27×1010(D-S)cells/gMLSS.硝化菌群在总菌中的百分含量大约为1%-7%.AOB种群数量AO工艺(B)最低,为8.56×106cells/gMLSS,其它 4个污泥样品在1.20×107~4.46×107cells/gMLSS之间.倒置A2O工艺 (A样品)AOB占总菌比例最高为0.3%,其他3个工艺在0.02%~0.08%之间.结合表1的进出水水质指标可以看出倒置A2O工艺的出水氨氮浓度最低(0.72mg/L),硝化效果最好.因此,AOB在总菌中的相对含量决定了水厂的硝化效果. Nitrospira在 D-S中丰度最高,达到了 1.53× 109cells/gMLSS,占总菌比例为1.64%,在其他3个工艺中数量级均为 108cells/gMLSS,占总菌比例为 1.36%~6.12%之间.Nitrobacter除了在样品D-S中丰度为零外,其余在 1.69×107~6.78× 107cells/gMLSS之间,占总菌比例为 0.15%~0.39%之间.D-W中的总菌、AOB、Nitrospira丰度较D-S均有所降低,但Nitrobacter却由0增加到 1.45×107cells/gMLSS.冬季样品(D-W)中硝化菌群丰度和百分含量的降低可能是很多城市污水处理厂冬季脱氮效果变差的主要原因.

表3 总菌、AOB和NOB的实时定量结果Table 3 QPCR results of bacteria, AOB and NOB
本研究AOB amoA基因定量分析的数量级大致在107~108cells/gMLSS之间,与已有的多数研究结果基本一致[7,24-25],但略低于个别研究[26-27].对 NOB定量结果显示本研究中的城市污水处理系统中Nitrospira是NOB菌群中的优势菌属,丰度要比Nitrobacter高出一个数量级.关于AOB和NOB丰度,有的研究认为AOB和NOB在同一个数量级[16],也有研究发现AOB丰度比NOB低一个数量级[28].从表3可以看出,本研究的水厂中 AOB种群丰度要比 NOB (Nitrobacter和 Nitrospira)低一个数量级.除了样品 D-S(A2O工艺),其他工艺的 Nitrospira和Nitrobacter均存在,且相差一个数量级,只有样品D-S中Nitrobacter含量为0.结合表1中D-S出水的和,计算出亚硝积累率为24%,由此推断采用A2O工艺的D水厂在夏季可能出现了短程硝化现象,并且在向短程硝化转化的过程中,Nitrobacter先于 Nitrospira被淘汰.结合表 1的运行参数发现D-S的溶解氧要明显低于其他3个工艺,低溶解氧可能是造成短程硝化的原因之一.进入冬季,由于水温降低,短程硝化向全程硝化转化,Nitrobacter又出现在D-W样品中.
2.2基于OTU的AOB系统发育分析
5个amoA基因克隆文库中共得到242条amoA序列,按照 97%的相似度划分为 21个OTUs.5个污泥样品构建的 amoA基因克隆文库的Good覆盖率、Chao1丰富度估计、OTU值与估计的 Chao1值之比、香农指数见表4. 5个污泥样品的amoA基因的OTU分布如图1所示.
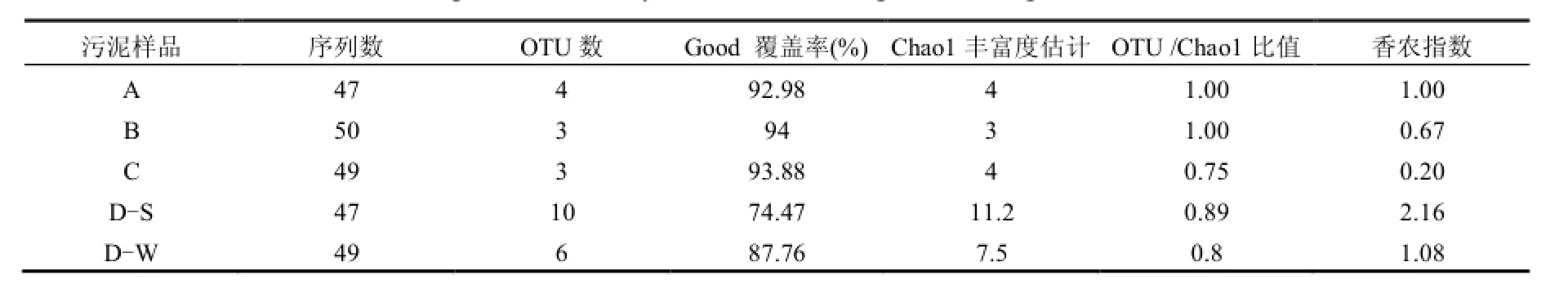
表4 amoA基因克隆文库的序列多样性及覆盖率Table 4 Sequences diversity and Good coverage of amoA gene clone libraries

图1 5个污泥样品中AOB amoA基因的OTU分布Fig.1 Relative distribution of OTUs based on amoA genes in 5samples
如表 4所示,Good覆盖率最低的是 DS74.47%,但其OTU值与估计的Chao1值之比达到89%.OTU值与估计的Chao1值之比最低的是样品C75%,其Good覆盖率达到了93.88%.这两组数据说明这2个amoA克隆文库可以代表2个污水处理厂AOB的群落组成.样品A和B的 Good覆盖率均在 90%以上,OTU值与估计的Chao1值之比均为 100%.与D-S比较,D-W的OTU值与估计的Chao1值为87.76%,明显高于D-S(74.47%),数据可信度更高.高景峰等[29]对 10个污水处理系统中的 AOB菌群进行研究,Good覆盖率在 65.4%~100%之间,说明本研究建立的amoA克隆文库可以代表每个污水处理厂中AOB群落组成.表4计算了5个amoA基因克隆文库中的香农指数,根据表中结果发现C样品中amoA基因多样性要低于其他样品,香农指数只有0.2.另外4个样品的香农指数在0.67~2.16之间,对比其他研究可说明本研究的 5个样品的AOB 群落多样性在正常范围之内[24-25,29],其中D-S多样性最丰富.从图1和表4可以看出,样品C仅有3个OTU,且有2个OTU各仅含有1条序列,意味着样品C中的AOB多样性最不丰富.虽然样品A和B所含OTUs数分别为4和3,但从图1可以看出,每个样品所含序列较均匀的分布在各个 OTU,因此具有一定的生物多样性水平;样品D-S含有10个OTUs,多样性水平最高,与表4的香农指数分析一致. D-W共有6个OTU,其中有4个OTU与D-S是重复覆盖的,说明虽然污水处理系统温度发生了变化,OTU分布会有一定变化,但其优势OTU得以保留.
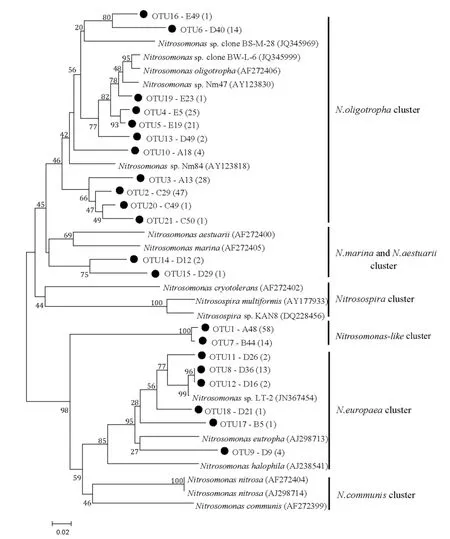
图2 基于amoA基因的AOB系统发育树Fig.2 NJ phylogenetic tree of AOB group based on amoA genes
利用21个OTUs中的代表序列建立的NJ系统发育树如图2所示.从图2可以看出有11个OTU属于N. oligotropha,有 6个OTU 属于N. europaea cluster,有2个OTU属于Nitrosomonaslike cluster,有2个OTU (仅包括3条序列)属于N. marina 和N. aestuarii.其中OTU1、OTU2、OTU3分别含有58、47、28条序列,是AOB菌群的优势OTU,属于N. oligotropha和Nitrosomonas-like cluster.
2.3AOB的系统发育树分析
5个污泥样品中AOB种群分布以及相对含量如图3所示.基于 amoA基因的系统发育分析结果(图2)显示所有的序列属于Nitrosomonas,没有发现Nitrosospira cluster.AOB的分布从多到少分 别 为 :N. oligotropha、 Nitrosomonas-like cluster、N. europaea cluster、N.matina and N.aestuarii cluster.其中Nitrosomonas oligotropha cluster和Nitrosomonas-like cluster是AOB种群中的两大优势菌群,分别占克隆文库的60.08%和29.63%,与已有研究结果一致[2,30].从图 3可以看出,除了样品B中N.oligotropha cluster仅为2%,其余4个样品都含有N.oligotropha cluster且含量丰富.其中样品C所有的 AOB amoA序列以及样品D-W中97.96%的AOB amoA序列都属于N.oligotropha cluster.其 他2个 样 品 中N.oligotropha cluster占克隆文库的比例在40.43%~60.42%之间.Nitrosomonas-like cluster分布在3个样品中,并且在样品B中所占比例达到98%,在样品A和D-S中所占比例分别为39.58% 和 8.51%,在样品 C和 D-W 中未检测到.N. europaea cluster占克隆文库的 9.05%,只在采用A2O工艺的样品D-S和D-W中发现,这可能与该A2O工艺进水氨氮浓度相对较高有关.而且N. europaea cluster在夏冬两季样品中的百分含量发生明显的变化,D-S中 N. europaea cluster占AOB总量的 44.68%,是优势 AOB菌属,但在D-W样品中降到了2.04%.在其他3个工艺中并未检测到N. europaea cluster.另外,在D-S样品中发现了少量的N.matina和N.aestuarii cluster.
比较样品D-S和D-W的AOB群落结构,一个明显的变化特征是进入冬季后,原本处于优势的N. europaea cluster明显减少,而N. oligotropha cluster显著增多,达到AOB总量的98%.因此,冬季水厂硝化效果变差的原因除了与硝化菌群丰度降低有关,还可能与上述AOB群落结构的变化有关.如前所述,样品D-S所在的污水处理厂夏季出现较高的亚硝酸盐积累率,这可能与夏季样品中N. europaea cluster的优势地位有关;冬季随着短程现象的消失,N. europaea cluster含量减少.有研究认为N. europaea 有优先利用亚硝酸盐作为电子受体的能力[30-31],经过低 DO驯化的短程硝化活性污泥中以N. europaea为主[32].从表1可以看出,4种工艺中,D-S样品所在的A2O工艺 DO值最低,仅为 1.8mg/L,远低于其他 3种工艺(3~6mg/L).而且只有A2O工艺(D-S和D-W)中出现了N. europaea,并在样品D-S克隆文库中所占比例达到44.69%.冬季短程硝化逐渐变为全程硝化,隶属于NOB的Nitrobacter重新出现,同时系统内N. europaea cluster大量消失,在样品D-W中仅占AOB总量的2%.由此可以推测低DO运行使N. europaea成为优势AOB可能是D-S出现短程硝化的原因.

图3 5个污泥样品中AOB种群分布以及种群相对含量Fig.3 Phylogenetic distribution and relative abundance of AOB groups in 5samples
3 结论
3.1本研究的城市污水处理厂活性污泥样品中优势 AOB和 NOB分别为 Nitrosomonas和Nitrospira.硝化菌群达到总菌群的 1%~7%,其丰度,尤其是 AOB在总菌中的相对含量决定了系统的硝化效果.AOB丰度比NOB丰度低一个数量级.NOB菌群中的Nitrospira丰度比Nitrobacter丰度高一个数量级,是明显的优势NOB.
3.2本研究的活性污泥样品中的 AOB全部隶属于Nitrosomonas,其中N. oligotropha cluster是AOB 中的优势菌属,占克隆文库的 60%. Nitrosomonas-like cluster次之,占克隆文库的30%. N. europaea cluster只出现在A2O工艺中. 3.3 A2O工艺冬季AOB和Nitrospira丰度的降低是导致冬季生物脱氮效果变差的原因.A2O工艺夏季污泥样品中N. europaea cluster占该克隆文库的 44.7%,是优势 AOB.低 DO运行使 N. europaea cluster成为优势AOB是A2O工艺夏季出现较高亚硝酸盐积累率的主要原因.
[1] Yin J, Xu W F. Ammonia biofiltration and community analysis of ammonia-oxidizing bacteria in biofilters [J]. Bioresource Technology, 2009,100(17):3869-3876.
[2] Purkhold U, Pommerening-röser A, Juretschko S. Phylogeny of all recognized species of ammonia oxidizers based on comparative 16S rRNA and amoA sequence analysis:implications for molecular diversity surveys [J]. Applied Environmental Microbiology, 2000,66(12):5368-5382.
[3] Orso S, Gouy M, Navarro E, et al. Molecular phylogenetic analysis of Nitrobacter spp. International Journal of Systematic Bacteriology, 1994,44:83-86.
[4] Ye L, Zhang T. Ammonia-oxidizing bacteria dominates over ammonia-oxidizing archaea in a saline nitrification reactor under low DO and high nitrogen loading [J]. Biotechnology and Bioengineering, 2011,108(11):2544-2552.
[5] Limpiyakorn T, Sonthiphand P, Rongsayamanont C. Abundance of amoA genes of ammonia-oxidizing archaea and bacteria in activated sludge of full-scale wastewater treatment plants [J]. Bioresource Technology, 2011,102(4):3694-3701.
[6] Mußmann M, Brito I, Pitcher A. Thaumarchaeotes abundant in refinery nitrifying sludges express amoA but are not obligate autotrophic ammonia oxidizers [J]. Proceedings of the National Academy of Sciences of the United States of America, 2011,108(40):16771-16776.
[7] Wells G F, Park H D, Yeung C H, et al. Ammonia-oxidizing communities in a highly aerated full-scaleactivated sludge bioreactor: betaproteobacterial dynamics and low relative abundance of Crenarchaea [J]. Environmental Microbiology, 2009,11(9):2310-2328.
[8] Zhang T, Ye L, Tong A, et al. Ammonia-oxidizing archaea and ammonia-oxidizing bacteria in six full-scale wastewater treatmentbioreactors[J].AppliedMicrobiologyand Biotechnology, 2011,91(4):1215-1225.
[9] Gieseke A, Bjerrum L, Wagner M. Structure and activity of multiple nitrifying bacterial populations co-existing in a biofilm [J]. Environmental Microbiology, 2003,5(5):355-369.
[10] Cébron A, Garnier J. Nitrobacter and Nitrospira genera as representativesofnitrite-oxidizingbacteria:Detection,quantification and growth along the lower Seine River (France)[J]. Water Research, 2005,39(20):4979-4992.
[11] Siripong S, Rittmann B E. Diversity study of nitrifying bacteria in full-scale municipal wastewater treatment plants [J]. Water Research, 2007,41(5):1110-1120.
[12] Robinson K G, Dionisi H M, Harms G. Molecular assessment of ammonia and nitrite-oxidizing bacteria in full -scale activated sludge wastewater treatment plants [J]. Water Science and Technology, 2003,48(8):119-126.
[13] Bollmann A, French E, Laanbroek H J. Isolation, cultivation, and characterization of ammonia-oxidizing bacteria and archaea adapted to low ammonium concentrations [J]. Methods Enzymol,2011,486:55-88.
[14] Geets J, Boon N, Verstraete W. Strategies of aerobic ammonia-oxidizing bacteria for coping with nutrient and oxygen fluctuations [J]. FEMS Microbiol Ecol, 2006,58:1-13.
[15] Blackburne R, Yuan Z, Keller J. Partial nitrification to nitrite using low dissolved oxygen concentration as the main selection factor [J]. Biodegradation, 2008,19(2):303-312.
[16] Wang F, Liu Y, Wang J H, et al. Influence of growth manner on nitrifying bacterial communities and nitrification kinetics in three lab-scale bioreactors [J]. J Ind Microbiol Biotechnol, 2012,39:595-604.
[17] Kowalchuk G A, Stienstra A W, Heilig G H. Changes in the community structure of ammonia-oxidizing bacteria during secondary succession of calcareous grasslands [J]. Environmental Microbiology, 2000,2(1):99-110.
[18] Rotthauwe J, Witzel K, Liesack W. The ammonia monooxygenase structural gene amoA as a functional marker: molecular fine-scale analysis of natural ammonia-oxidizing populations [J]. Appl. Environ. Microbiol., 1997,63(12):4704-4712.
[19] Nicolaisen M H, Ramsing N B. Denaturing gradient gel electrophoresis (DGGE) approaches to study the diversity of ammonia-oxidizing bacteria [J]. J Microbiol Methods, 2002,50:189-203.
[20] Purkhold U, Wagner M, Timmermann G. 16S rRNA and amoA based phylogeny of 12 novel betaproteobacterial ammoniaoxidizing isolates: Extension of the dataset and proposal of a new lineage within the nitrosomonads [J]. International Journal of Systematic and Evolutionary Microbiology, 2003,53(5):1485-1494.
[21] Aakra A, Utaker J, Nes I. Comparative phylogeny of the ammoniamonooxygenase subunitand 16S rRNA genes of ammoniaoxidizing bacteria [J]. FEMS Microbiology Letters, 2001,205:237-242.
[22] Hoshino T, Noda N, Tsuneda S. Direct detection by in situ pcr of the amoa gene in biofilm resulting from a nitrogen removal processss [J]. Applied Environmental Microbiology, 2001,67(11):5261-5266.
[23] Ferris M J, Muyzer G, Ward D M. Denaturing gradient gel electrophoresis profiles of 16S rRNA-defined populations inhabiting a hot spring microbial mat community [J]. Appl. Environ. Microbiol., 1996,62(2):340-346.
[24] Gao J, Luo X, Wu G, et al. Quantitative analyses of the composition and abundance of ammonia-oxidizing archaea and ammonia-oxidizing bacteria in eight full-scale biological wastewater treatment plants [J]. Bioresource Technology, 2013,138:285-296.
[25] Bai Y, Sun Q, Wen D, et al. Abundance of ammonia-oxidizing bacteria and archaea in industrial and domestic wastewater treatment systems [J]. FEMS Microbiology Ecology, 2012,80(2):323-330.
[26] Limpiyakorn T, Sonthiphand P, Rongsayamanont C, et al. Abundance of amoA genes of ammonia-oxidizing archaea and bacteria inactivated sludge of full-scale wastewater treatment plants [J]. Bioresource Technology, 2011,102(4):3694-3701.
[27] Kayee P, Sonthiphand P, Rongsayamanont C, et al. Archaeal amoA genes outnumber bacterial amoA genes in municipal wastewater treatment plants in Bangkok [J]. Microbial Ecology,2011,62(4):776-788.
[28] Zeng W, Bai X, Zhang L, et al. Population dynamics of nitrifying bacteria for nitritation achieved in Johannesburg (JHB) process treating municipal wastewater [J]. Bioresource Technology, 2014,162:30-37.
[29] Gao J, Luo X, Wu GX, et al. Abundance and diversity based on amoA genes of ammonia-oxidizing archaea and bacteria in ten wastewater treatment systems. Appl. Microbiol. Biotechnol., 2014,98:3339-3354.
[30] Bock E, Schmidt I, Stüven R, et al. Nitrogen loss caused by denitrifying Nitrosomonas cells using ammonium or hydrogen as electron donors and nitrite as electron acceptor [J]. Arch. Microbiol, 1995,163:16-20.
[31] Kuai L P, Verstraete W. Ammonium removal by the oxygen-limited autotrophic nitrification-denitrification system [J]. Appl. Environ. Microbiol, 1998,64(11):4500-4506.
[32] Park H D, Noguera D R. Evaluating the effect of dissolved oxygen on ammonia-oxidizing bacterial communities in activated sludge [J]. Water Res., 2004,38:3275-3286.
Community structures and population dynamics of nitrifying bacteria in activated sludges of wastewater treatmentplants.
ZENG Wei1*, ZHANG Li-min1, WANG An-qi1, ZHANG Jie1, PENG Yong-zhen1, DUAN Jun-ling2(1.College of Environmental and Energy Engineering, Beijing University of Technology, Beijing 100124, China;2.Beijing Drainage Group Limited Liability Company, Beijing 100044, China).
China Environment Science, 2015,35(11):3257~3265
Community structures and population dynamics of nitrifying bacteria determine biological nitrogen removal from municipal wastewater. The population structures and dynamics of ammonia-oxidizing bacteria (AOB) and nitrite-oxidizing bacteria (NOB) in four full-scale wastewater treatment plants (WWTPs) were investigated in this study. Quantitative real-time PCR results showed that the abundance of AOB was in a range of 8.56×106~4.46×107cells/gMLSS,while NOB was varying in 3.37×108~1.53×109cells/gMLSS. In each process Nitrospira was the dominant species of NOB. Nitrospira abundance was obviously higher than Nitrobacter, accounting for 88% of total NOB. In the A2O process the abundances of AOB and Nitrospira in winter were less than those in summer, leading to decline of biological nitrogen removal. The phylogenetic analysis of AOB amoA genes indicated that all the sequences were affiliated with genera Nitrosomonas, among which Nitrosomonas oligotropha cluster was the dominant species, accounting for 60% of the clone libraries. The pre-dominant AOB were Nitrosomonas-like cluster and Nitrosomonas europaea cluster, accounting for 29.6% and 9.1% of the clone libraries, respectively. N. europaea cluster was only found in A2O process, and reached 44.7% of total AOB in summer sample, which was a main reason causing high nitrite accumulation during summer operation of A2O process. The outcomes verified that the dominant AOB and NOB in WWTPs was Nitrosomonas and Nitrospira, respectively. Nitrifying bacteria accounted for 1%~7% of total bacteria. The abundances, relative distributions and community structures of nitrifying bacteria significantly influence the performance of biological nitrogen removal.
ammonium oxidizing bacteria (AOB);nitrite oxidizing bacteria (NOB);wastewater treatment systems (WWTPs);real-time quantitative PCR (QPCR);amoA genes
X703
A
1000-6923(2015)11-3257-09
2015-04-03
国家自然科学基金项目(51278007,51578016);教育部新世纪优秀人才支持计划项目(NCET-11-0891)
* 责任作者, 教授, zengwei_1@263.net
曾 薇(1974-),女,黑龙江省哈尔滨人,教授,博士,主要从事污水生物处理研究.发表论文70余篇.

