One tube nested-PCR 在SNP 基因型分型中的应用
张羽,张晓娟,王胜宝,宋晓利,李小刚
(1.陕西理工学院生物科学与工程学院,陕西 汉中 723000;2.陕西省农业科学研究所,陕西 汉中 723000)
单核苷酸多态性(single nucleotide polymer- phism,SNP)主要是指由于基因组核苷酸水平上的单碱基的转换、颠换以及单碱基的插入或缺失变异引起的DNA 序列多态性,是许多物种基因组中最常见的变异形式,在基因组中的分布频率很高。SNP在人类[1]、老鼠[2]、拟南芥[3]、大麦[4–5]、大豆[6]、甜瓜[7]、玉米[8]和水稻[9–12]等多个物种中的高密度分布已有报道。作为第3 代分子标记的SNP,在基因组中数量多,分布密度高,特别是由于其不需要根据片段大小将DNA 分型,因此,可以用高通量基因芯片技术自动化完成基因分型。目前,检测SNP 的方法有很多种,但有的需要昂贵的设备及试剂;有的检测过程繁琐,适合于普通实验室检测的不多。传统的nested–PCR(巢式PCR)是一种PCR 改良模式,它由2 轮PCR 扩增和2 套引物对组成。首先利用外引物对DNA 进行第1 轮扩增,然后以第1次PCR 反应的产物作为模板进行第2 轮扩增,第2次PCR 引物与第1次反应产物的序列互补,第2次PCR 产物即为目的基因[13–16]。其优点在于:如果第一次扩增产生了错误片断,那么,第二次能在错误片段上进行引物配对并扩增的几率是很低的,因此,巢式PCR 的扩增特异性强,且传统的巢式PCR 需要进行2 轮PCR。本研究中利用one tube nested–PCR 方法,把nested–PCR 的4 条引物同时加在1 管PCR 反应中进行扩增,通过引物设计、引物浓度梯度优化及反应程序改良,达到一步快速检测SNP 基因型的目的。本研究中以1个水稻香味基因Fgr 和3个稻瘟病基因Pi–ta、Pi9、Pigm为例,探索了one tube nested–PCR 技术的引物设计原理和反应条件,旨在为生物的SNP 基因分型研究提供参考。
1 材料和方法
1.1 材料
以宜香A 为香稻品种对照材料,黄花占为非香稻品种对照材料。Pi–ta 的抗病对照材料为Tetep,感病对照材料为Nipponbare。Pi9 基因抗病对照材料为WHD75–1–127,感病对照材料为93–11。Pigm基因抗病对照材料为谷梅4,感病对照材料为Nipponbare[17]。以上材料均由陕西省水稻研究所和国家水稻种质资源中期库提供。
1.2 方法
1.2.1 引物设计
通过 NCBI 网站(http:www.ncbi.nlm.nih.gov) GenBank 登录号找出所研究基因的核苷酸序列及Nipponbare 和93–11 中对应的染色体序列,编号为AP004463。BAC 克隆得到水稻非香型基因的核苷酸序列,香型基因核苷酸序列来自文献[18]的报道。GenBank 登录号为AF207842 和AY196754 的BAC克隆得到编码稻瘟病基因 Pi–ta 的基因序列;GenBank 登录号为DQ285630.1 的BAC 克隆得到Pi9 基因序列;GenBank 登录号为AP005930.3 的BAC 克隆得到Nipponbare 的6 号染色体序列;GenBank 登录号为AAAA02018909 的BAC 克隆得到93–11 的6 号染色体序列。
同一基因的不同基因型个体进行同源序列比对,根据序列差异设计引物。在设计引物时,3′端落在发生SNP 的位置,由于Taq 酶缺乏3′→5′外切酶的活性,因此,如果3′末端的碱基和模板不互补,特异性扩增条带就会因为延伸速度低于正常末端碱基配对引物的延伸速度而不出现,表明模板DNA与引物3′末端没有相应的突变;反之,则表明模板DNA 上存在与引物3′末端相应的突变碱基。如图1所示,将特异引物(P2 和P3)的3′端落在发生SNP的位点上,如果设计P2 的3′端和野生型互补,那么P3 的3′端就设计成和突变型互补。如图2 所示,2 条特异性条带(P1P3 和P2P4)和对照带(P1P4)都出现的个体为杂合体,只出现1 条特异性条带和对照带的为纯合体。

图1 PCR 4 条引物的位置 Fig.1 Relative positions of four primers for PCR

图2 PCR 扩增电泳结果示意图 Fig. 2 Schematic of PCR products on electrophoresis
1.2.2 基因组DNA 提取及PCR
用SDS 法提取水稻基因组DNA。用0.8%的琼脂糖凝胶检测水稻基因组DNA。
PCR 反应体系为:2×Taq MasterMix 15 uL,水稻基因组DNA 1 μL,内外引物比5∶1,反应总体积30 μL。
反应程序为:94.0℃预变性3.5min,94.0℃变性30 s,4 条引物中Tm最高值增加5℃开始降温,每个循环降低1℃,退火1min,72.0℃延伸45 s,15个循环。94℃变性30 s,Tm最低值降低5℃,退火30 s,72.0℃延伸30 s,20个循环,最后72.0℃延伸7min。
用2% ~3%的琼脂糖凝胶电泳分离PCR 产物,用BioRad 凝胶成像仪拍照,或用8%的非变性聚丙烯酰胺凝胶电泳分离PCR 产物,用银染法显色拍照。
2 结果与分析
2.1 引物信息
用来扩增4个结构基因的引物序列如表1 所示。
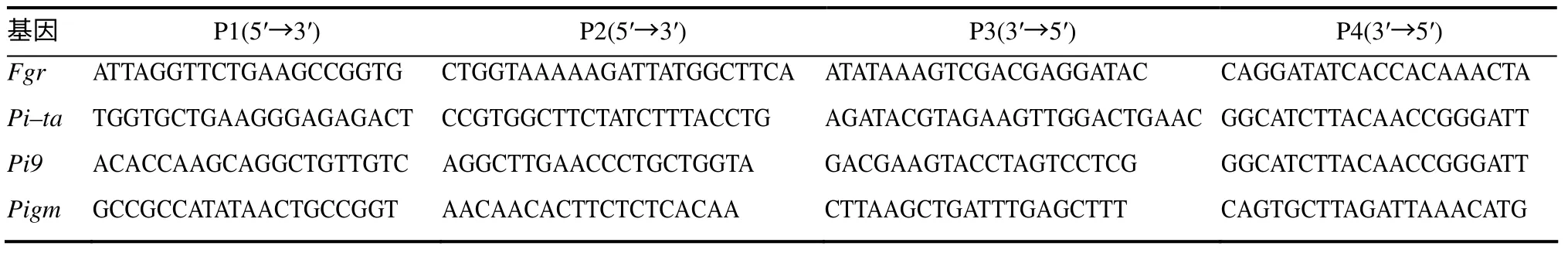
表1 引物信息 Table 1 Primers information
2.2 发生SNP 的位置及内外引物的位置
图3、图4、图5、图6 中方框所示为发生SNP的位置,下划线处为4 条引物所在位置。从图3 可以看出,非香稻和香稻基因核苷酸序列同源性比对,发生了3个SNP(A→T,A→T,C→T)和8个Indel。如果设计的特异引物序列包含了这11个突变,那么扩增特异性将非常强。

图3 非香稻和香稻Fgr 基因结构部分核苷酸序列的同源性比对结果 Fig. 3 Results of homology alignment on partial Fgr sequences of fragrant and non-fragrant strains
图4 为抗病材料和感病材料Pi–ta 基因核苷酸序列的比对结果。本研究中发生了1个SNP(G→T)。依照Bryan 等[19]的研究结果,由于发生了1个SNP(G→T),导致抗感基因的编码产物由抗病产物的丙氨酸(GCT)变异为感病产物的丝氨酸(TCT)。

图4 抗病材料和感病材料Pi–ta 基因结构部分核苷酸序列的同源性比对结果 Fig. 4 Results of homology alignment on partial Pi–ta sequences of the resistant and susceptible strains
图5 为Pi9 基因、粳稻Nipponbare 的6 号染色体与籼稻93–11 的6 号染色体基因序列的同源性比对 结果。从理论上讲,只要含Pi9 基因和不含Pi9基因的材料发生了SNP 的变化,都可以在发生SNP处设计引物加以区别。

图5 抗病材料和感病材料Pi9 基因结构部分核苷酸序列的同源性比对结果 Fig. 5 Results of homology alignment on partial Pi9 sequences of disease-resistant and disease-susceptible strains
Deng Yiwen 等[20]将谷梅4 号的Pigm 定位于水稻第6 染色体上标记C5483 和C0428 之间的70 kb区间内,包含5个NBS–LRR 抗性基因,与Pi2、Pi9 紧密连锁或等位,并在此区间开发了检测Pigm基因的CAPS(酶切扩增多态性序列)标记。笔者将Pigm 基因的CAPS 标记转换成SNP 标记。图6 所示为Pigm 基因包含CAPS 标记的部分核苷酸序列。

图6 Pigm 基因标记的相对位置及序列 Fig. 6 Relative marker positions and sequence of Pigm
2.3 检测结果
如图7 所示,在水稻的香味基因和非香基因DNA 序列比对中不仅出现了3个SNPs,还有8 bp的InDels,这使得包含此多态性序列的特异性引物的特异性结合进一步增强,用one tube nested– PCR方法,在野生型和突变型基因型上很容易区分。香稻材料扩增出188 bp 的特异性条带,非香稻材料扩增出279 bp 的特异性条带,400 bp 左右的条带为外引物扩增的共有带。

图7 one tube nested–PCR 检测Fgr 基因结果 Fig. 7 The detection results of Fgrgene with one tube nested–PCR
稻瘟病抗性基因Pi–ta 的抗病材料扩增出286 bp 的特异条带,感病材料扩增出161 bp 的特异条带(图8)。

图8 One tube nested–PCR 检测Pi–ta 基因结果 Fig. 8 The detection results of Pi–tagene with one tube nested–PCR
Pi9 基因的分子标记有PB8[21–22]和PB9–1[23]。PB8 标记是显性标记,不能区分个体的基因型是杂合体还是纯合体,只能用在育种的早期世代选择。PB9–1 标记虽然是共显性标记,但含有抗性Pi9 基因的材料和粳稻材料只相差3 bp,和籼稻材料只相差5 bp,用琼脂糖凝胶区分不了,而聚丙烯酰胺凝胶只有在胶浓度大、胶薄、电泳时间长的实验条件下才能区别开来[24]。本研究中根据Pi9 基因序列和感病对照材料Nipponbare、93–11 进行同源性比较的结果和不同水稻基因型的SNP 开发出了与Pi9 基因紧密连锁的共显性标记,抗病材料扩增出341 bp的特异条带,感病材料扩增出219 bp 的特异条带,外引物扩增出521 bp 左右的条带(图9 所示)。

图9 one tube nested–PCR 检测Pi9 和Pigm 的结果 Fig.9 Detection results of Pi9 and Pigmgene with one tube nested–PCR
基于目前公共数据库中还没有Pigm 基因序列,而CAPS 标记就是因为在酶切识别序列内发生了SNP,根据PCR 扩增后酶切判断基因型,如果酶切 不完全会造成基因型误读,另外有的酶价格十分昂贵,故本研究中将Deng Yiwen 等[20]报道的检测Pigm 基因的CAPS 标记转换成SNP 标记。经研究,Pigm 基因感病纯合体的一段序列为GAATTC,这正好是EcoRI 的酶切识别位点,而其抗病纯合体的此段序列突变为AAATTC(G→A)。本研究中根据发生的这个SNP,设计了4 条引物进行Pigm 基因的检测。抗病材料扩增出150 bp 的特异带,感病材料扩增出200 bp 的特异带,外引物扩增出310 bp 左右的带(图9)。
3 利用开发的基因标记鉴定资源的结果
在构建引物时,发生SNP 的位置决定了2 条内引物(特异引物)的位置,1 条内引物的3′末端设计成和野生型基因的序列完全互补,另1 条内引物的3′末端设计成和突变型匹配。外引物的位置调整遵循以下的原则:首先,保证2 条特异性条带大小容易在琼脂糖凝胶上区分(至少把2 条特异性条带大小差异控制在50 bp 以上);其次,外引物扩增的条带不能太大,虽然外引物条带不影响基因型的判读,由于和传统PCR 技术及nested–PCR 技术不同,在本研究中,当目的片段大小在400 bp 以上时难以扩增出产物,从Pi–ta 和Pi9 基因的目的片段大小及扩增结果得到验证(图8、图9、图10),借鉴前面的经验,在设计Pigm 的4 条引物的时候,把Pigm 的目的片段控制在400 bp 以内,所有的目的片段都得到了很好的扩增(图11)。图10 和图11 为检测陕西省2013年水稻区域试验结果的随机抽取,由于试验材料大部分为F1,因此基因型大多数为杂合体。

图10 利用开发的Pi9 基因标记鉴定资源结果 Fig.10 Detection resource results via developed Pi9genetic markers

图11 利用开发的Pigm 基因标记鉴定资源结果 Fig.11 Detection resource results via developed Pigmgenetic markers
4 讨论
传统的三引物法检测SNP 需要对同一个样品进行2次PCR,而one tube nested–PCR 技术,通过一管PCR 扩增,即可判读材料的基因型。one tube nested–PCR 技术由于是在1个PCR 反应中同时加入4 条引物,反应中不仅涉及引物与模板间的竞争,同时还涉及引物之间的竞争,因此它比常规的nested–PCR 技术要难,关键是多个靶点扩增条件往往不兼容,特别是退火温度,克服4 条引物的退火温度不一致的情况,可以采用以下方式增加PCR 扩增的特异性:1) 复性温度采用变温的方式,先高温复性后低温复性以及先低温后高温复性的方式进行PCR 扩增;2) 采用传统的touch–down PCR 的方法,从4 条引物中的最高Tm值每个循环降低1℃至最低Tm值;3) 改良touch–down PCR 的方法,在4条引物中最高温度的1 条Tm值增加5℃开始降温,每个循环降低1℃,运行15个循环,然后再在4条引物中最低1 条的Tm值降低5℃,运行20个循环。另外,不同引物的浓度也会影响反应结果,首先,4 条引物按相同浓度添加,然后通过增加“弱”条带的引物浓度和减少“强”条带的引物浓度来使3条带扩增强度一致。
[1] Sachidanandam R,Weissman D,Schmidt S C,et al.The International SNP map workinggroup:A map of humangenome sequence variation containing 1.42 million single nucleotide polymorphisms[J].Nature,2001,409:928–933.
[2] Lindblad-Toh K,Winchester E,Daly M J,et al. Large-scale discovery andgenotyping of single- nucleotide polymorphisms in the mouse[J].Nature Genet,2000,24(4):381–386.
[3] Cho R J,Mindrinos M,Richards D R,et al. Genome- wide mapping with bi-allelic markers in Arabidopsis thaniana[J].Nature Genet,1999,23(2):203–207.
[4] Kato R,Varshney R K,Thiel T,et al.Generation and comparision of EST-derived SSRs and SNPs in barley[J].Hereditas,2001,135(2/3):145–151.
[5] Kanazin V,Talbert H,See D,et al.Discovery and assay of single-nucleotide polymorphisms in barley[J].Plant Mol Biol,2002,48(5/6):529–537.
[6] Zhu Y L,Song Q J,Hyten D L,et al.Single-nucleotide polymorphisms in soybean[J].Genetics,2003,163(3):1123–1134.
[7] Morales M,Roig E,Monforte A J,et al.Single-nucleotide polymorphisms detected in expressed sequence tags of melon[J].Genome,2004,47(2):352–360.
[8] Balley J,Barker G,Edwards K J,et al.Mining for single nucleotide polymorphisms and insertion/deletion in maize expressed sequence tag data[J].Plant Physiol,2003,132(1):84–91.
[9] Goff S A,Ricke D,Lan T H,et al.A draft sequence of the ricegenome[J].Science,2002,79(5565):92–100.
[10] International Rice Genome Sequencing Project.The map-based sequence of the ricegenome[J].Nature,2005,436(7052):793–800.
[11] Nasu S,Suzuki J,Ohta R,et al.Search for an analysis of single nucleotide polymorphism(SNP) in rice and establishment of SNP markers[J].DNA Res,2002,9(5):163–171.
[12] Feltus F A,Wan J,Schulze S R,et al.An SNP resource for ricegenetics and breeding based on subspecies incica and japonicagenome alignments[J].Genome Res,2004,14(9):1812–1819.
[13] 王婧,毕阳,朱艳,等.巢式PCR快速检测西瓜细菌性果斑病菌[J].中国农业科学,2014,47(2):284–291.
[14] Jiang J Z,Liang Y Y,Luo L J,et al.Nested PCR detection of abalone shriveling syndrome-associated virus in China[J].Journal of Virological Methods,2012,184:21–26.
[15] Shimano S,Sambe M,Kasahara Y.Application of nested PCR–DGGE(denaturinggradientgel electrophoresis) for the analysis of ciliate communities in soils[J]. Microbes and Environments,2012,27(2):136–141.
[16] Shen Wankuan,Xi Pinggen,Li Minhui,et al. Development of a sensitive nested-polymerase chain reaction (PCR) assay for the detection of Ustilago scitaminea [J].African Journal of Biotechnology,2012,11(46):10541–10547.
[17] 国家水稻数据中心.基因数据库.http://www.Ricedata. cn/gene/,2014–05.
[18] Louis M,Bradbury T,Timothy L,et al.Thegene for fragrance in rice[J].Plant Biotechnology Journal,2005,3:363–370.
[19] Bryan G T,Wu K S,Farrall L,et al.A single amino acid difference distinguishes resistant and susceptible alleles of the rice blast resistancegene Pi-ta[J].Plant Cell,2000,12(11):2033–2046.
[20] Deng Y,Zhu X,Shen Y,et al.Genetic characterization and fine mapping of the blast resistance locus Pigm (t) tightly linked to Pi2 and Pi9 in a broad-spectrum resistant Chinese variety[J].Theor Appl Genet,2006,113:705–713.
[21] Liu G,Lu G,Zeng L,et al.Two broad–spectrum blast resistancegenes,Pi9 (t) and Pi2 (t) are physically linked on rice chromosome 6[J].Mol Genet Genomics,2002,267:472– 480.
[22] Qu S H,Liu G F,Zhou B,et al.The broad-spectrum blast resistancegene Pi9 encodes a nucleotide-binding site- leucine-rich repeat protein and is a member of a multigene family in rice[J].Genetics,2006,172:1901– 1914.
[23] 殷得所,夏明元,李进波,等.抗稻瘟病基因Pi9 的STS 连锁标记开发及在分子标记辅助育种中的应用[J].中国水稻科学,2011,25(1):25–30.
[24] 张羽,冯志峰,张晗,等.陕西省水稻种质资源中Pi9基因的分布状况[J].四川农业大学学报,2013,31(2):115–121.

