4 种蔷薇珊瑚的核糖体基因ITS 的序列分析和分子鉴定
张 颖,姚雪梅,王尔栋,杜江涛,李洪武
(1.海南大学海洋学院,海南 海口570228;2.三亚理工职业学院,海南 三亚572002)
蔷薇珊瑚属(Montipora)属刺胞动物门(Cnidaria),珊瑚虫纲(Anthozoa),六放珊瑚亚纲(Hexacorallia),石珊瑚目(Scleractinnia),鹿角珊瑚科(Acroporidae).该属从始新世至今分布在印度-太平洋区,是该区系第二大的造礁石珊瑚属,其物种也是海南珊瑚资源的优势种之一.因此,明确蔷薇珊瑚属内各种间的遗传关系和分类地位对于研究整个珊瑚礁生态有着重要的意义.
蔷薇珊瑚体小,生长型多,变异大,鉴定难度较大. 珊瑚骼亚块状、叶片状、分枝或皮壳状,无轴珊瑚体,珊瑚体壁多孔,无轴柱或微弱发育,其共骨网状结构主要分为平滑型/浅窝型和乳突型/瘤突型两大类[1].共骨特征是蔷薇珊瑚传统形态学分类的主要依据之一.然而,由于蔷薇珊瑚极易受生长环境塑造,具有很强的表型可塑性,因此,同种珊瑚于不同生长环境的生态种差别极大;而不同种的石珊瑚在相同的生长环境中又会产生趋同进化,其生态型也可能较为接近[2-6],这使得蔷薇珊瑚在种水平上的鉴定相当困难,常存在很多争议.该属的有效种至今仍是未知数,此时若以分子生物学的方法作为工具来加以辨认和区分,相对于形态学性状而言,分子性状不仅是前者的补充,而且它还具有前者无法比拟的许多优点,能够较准确地反映生物类群之间的系统发生关系[7-8].
目前,用于分子系统发育学研究的主要方法有核糖体DNA(Ribosomal DNA,rDNA)和线粒体DNA 序列分析.其中,rDNA 的内转录间隔区(Internal Transcribed Spacer,ITS)可作为非编码区,它能被5.8S rDNA 分成ITS1、ITS2 两段,受到的选择压力较小,进化速度快,有比较丰富的变异位点和信息位点,且便于设计引物和进行PCR 扩增,一般认为,它适合用于属间、种间,甚至种内的系统发育研究[9],也适合石珊瑚目的系统发生关系的研究[10].Takabayashi 等用石珊瑚特有的ITS 引物对7 个属的珊瑚进行了多样性研究,发现不仅可以利用ITS 序列进行种间基因的多样性研究,而且还可以利用它进行种内基因的多样性研究[11],如Lam 等曾利用ITS 序列来鉴定2 种香港地区的扁脑珊瑚[12],其后,ITS 序列被用于分析鹿角珊瑚属和沙珊瑚属的进化关系[13-15].
本文通过对采自海南西沙的疑惑蔷薇珊瑚、壁垒蔷薇珊瑚、单星蔷薇珊瑚和叶状蔷薇珊瑚的rDNA ITS 区(ITS1 + 5.8S + ITS2)序列的分析,构建了蔷薇珊瑚属的系统发育信息,探讨了ITS 序列作为分子标尺对该属分类研究的适用性,旨在为该属珊瑚的快速和准确的鉴种提供有力的帮助.
1 材料和方法
1.1 材料 本次所研究的4 种珊瑚均采自海南西沙,采样活体均被带回实验室暂养,先拍摄每个样品的整体照片,然后用镊子镊取鲜活珊瑚样品的小块(2 cm3),用酒精固定后,放置于体式显微镜下,观察珊瑚骨骼的微细结构.为了确定其形态学鉴定的可靠性,根据《中国动物志·造礁石珊瑚》所提供的形态学信息,对每个物种的群体特征、骨骼的微细结构进行认真对照,并进行种的鉴定.构建蔷薇珊瑚属ITS 进化树所用的珊瑚资料见表1.
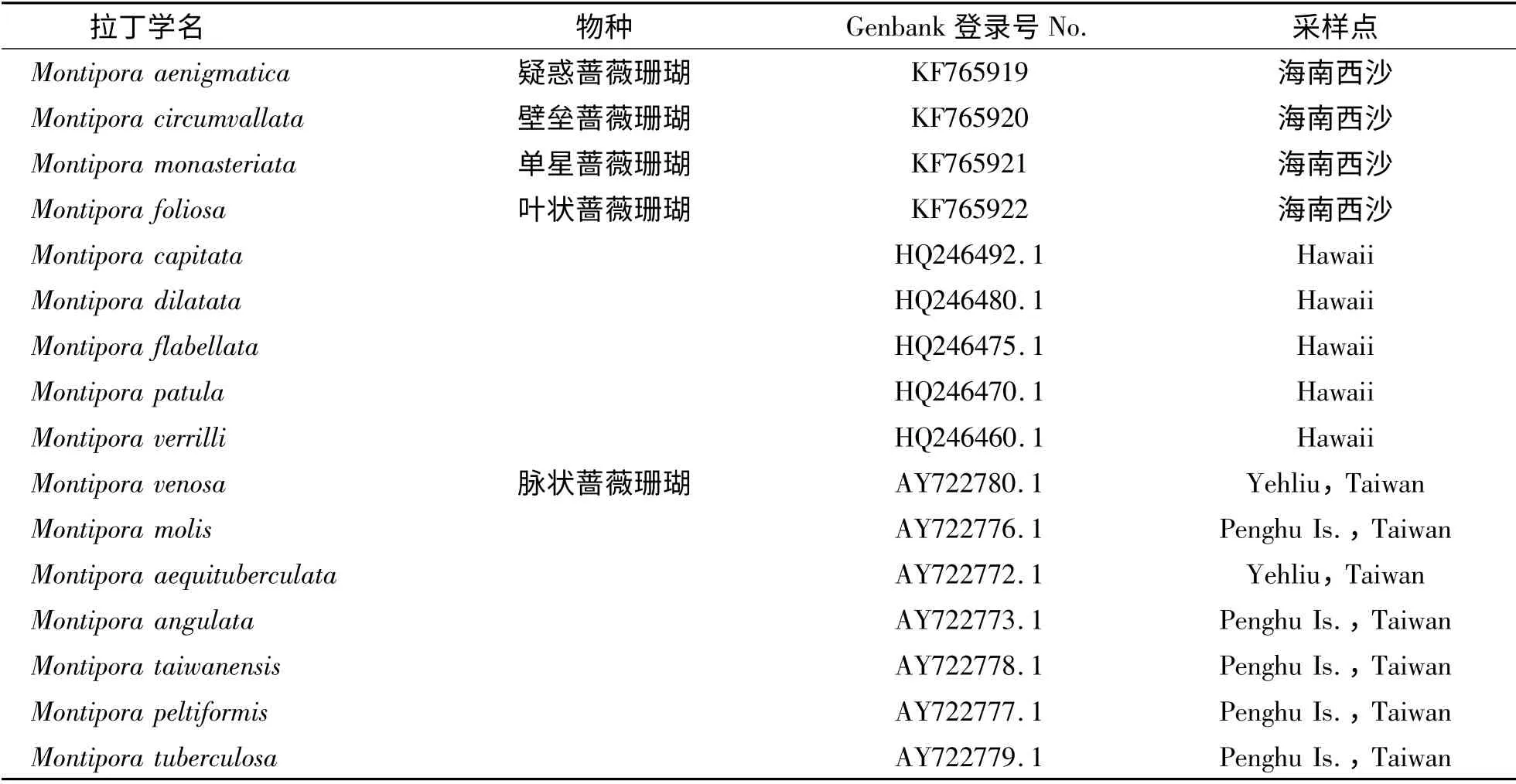
表1 构建珊瑚ITS 进化树所用的GenBank 序列信息
1.2 实验方法
1.2.1 DNA 的制备 采用宝生物工程(大连)有限公司的TaKaRa 基因组DNA 的广谱型提取试剂盒提取珊瑚基因组DNA.
1.2.2 目的片段的PCR 扩增及测序 根据GenBank 的蔷薇珊瑚rDNA 部分序列,设计通用引物5' -AAGGTTTCCG TAGGTGAACCTGC-3'和5' -CGTYGG ATTCCAACCGT-3'.反应体系为25 μL,包括10 ×Buffer 2.5 μL,MgCl2 2 μL,dNTP 2 μL,Taq 酶0.2 μL,引物各1 μL,DNA 模板1 μL,H2O 12.3 μL.PCR 反应程序:94 ℃预变性4 min;94 ℃变性35 s,58 ℃退火30 s,72 ℃延伸1 min,34 个cycle;72 ℃延伸10 min.所获得的片断需切胶回收、纯化及测序.
1.2.3 数据分析 对已经除去引物的序列进行Clustal X 排序,然后用DNA assist 软件两两比对,再结合人工校正后,用MEGA 5.1 进行碱基组成分析和遗传距离计算. 增加蔷薇珊瑚属的其他12 个种,使用MEGA 5.1 软件,以UPGMA 方法分析获得系统发育树.
2 结 果
2.1 克隆获得序列分析和两两比对 通过克隆获得的疑惑蔷薇珊瑚、壁垒蔷薇珊瑚、单星蔷薇珊瑚和叶状蔷薇珊瑚的片段长度为756 ~766 bp,均包括部分18S rDNA 区、ITS 区(ITS1 + 5.8S + ITS2)和部分28S rDNA 区.其中,ITS 区的总长度为535 ~545 bp,各区段的长度见表2. 4 种蔷薇珊瑚的部分18S rDNA区序列完全一致,ITS1 区有11 个位点变异,5.8S 区有1 个位点变异,ITS2 区有11 个位点变异,部分28S rDNA 区有2 个位点变异.从碱基缺失位点、数量和位置看,ITS 区的同源性很高.ITS1 序列的(G+C)含量为45.5% ~46.9%,ITS2 序列的(G+C)碱基含量为48.6% ~50.3%(见表2).
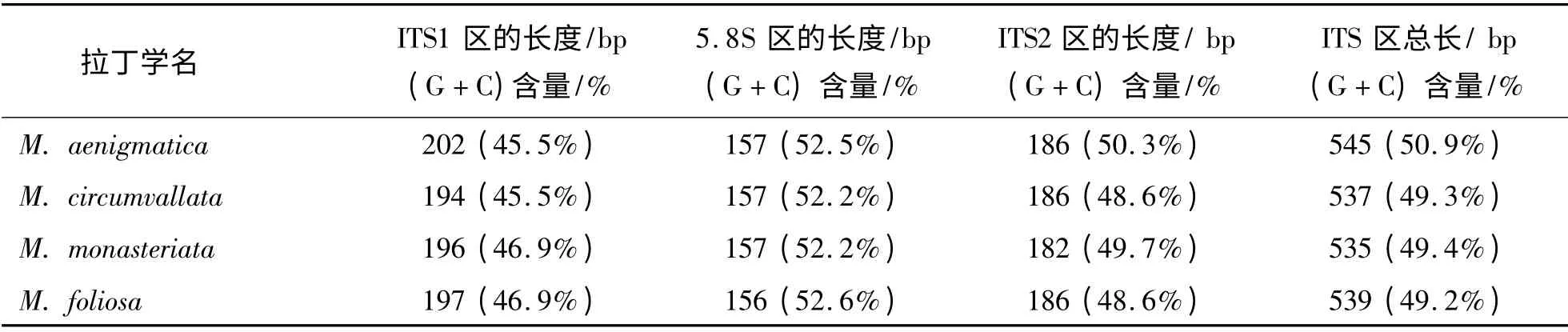
表2 4 种珊瑚核糖体基因ITS 区的长度和(G+C)含量
4 种蔷薇珊瑚的ITS 序列的两两比对结果见表3,由表3 可见,ITS 的同源性在96.9% ~99.1%之间,其中,ITS1 的同源性在95% 以上,ITS2 的同源性在94.1% 以上,5.8S rDNA 区的同源性在99.4% ~100%之间.对不同物种而言,ITS1 和ITS2 不会同时达到100% 的同源性,例如单星和叶状蔷薇珊瑚的ITS1 的同源性为100%,但是ITS2 的同源性则为94.1%.
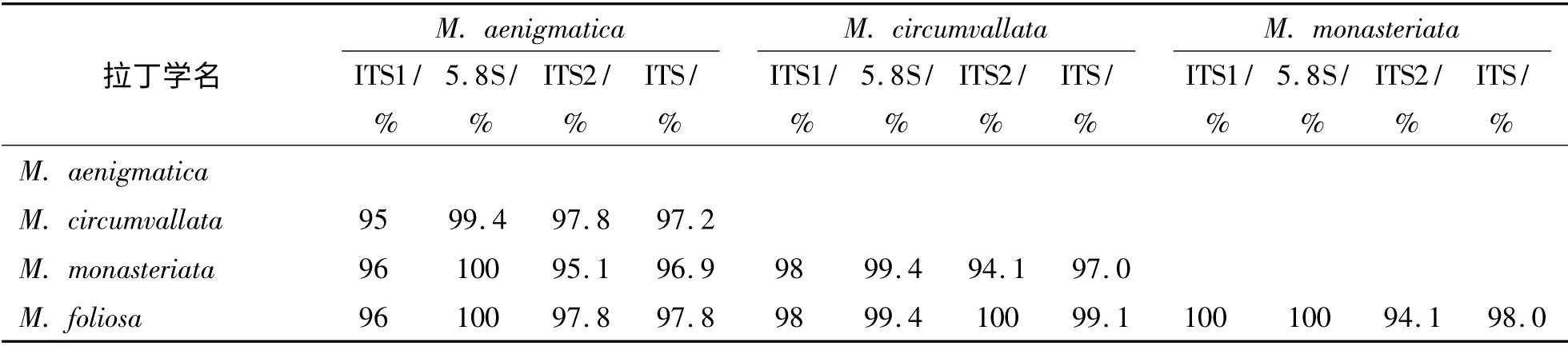
表3 ITS 序列的两两比对

表4 基于ITS 基因的16 种蔷薇珊瑚种间遗传距离
2.2 各类群序列遗传距离分析 具乳突型共骨特征的海南4 种蔷薇珊瑚的遗传距离在0.002 ~0.013之间,其他地区的乳突型共骨珊瑚,如M. peltiformis 和M. aenigmatic,其遗传距离平为0.007;具平滑型/浅窝型共骨特征的珊瑚,如M.venosa 和M.angulata,其遗传距离在0.02 左右;而M.capitata 和其他珊瑚的遗传距离在0.016 ~0.145 之间,明显大于其他珊瑚间的遗传距离(表4).
2.3 亲缘关系分析 4 种珊瑚序列的系统发育树结果如图5,由系统发育树可知,具乳突型/瘤突型共骨特征的蔷薇珊瑚集中在一起而形成一大支;具平滑型/浅窝型共骨特征的珊瑚则集中为另外一大支;M.capitata 单独分出一支.本研究的海南4 种蔷薇珊瑚位于具乳突型/瘤突型共骨特征的珊瑚进化树分支上,且很集中地位于某一个小分支上.
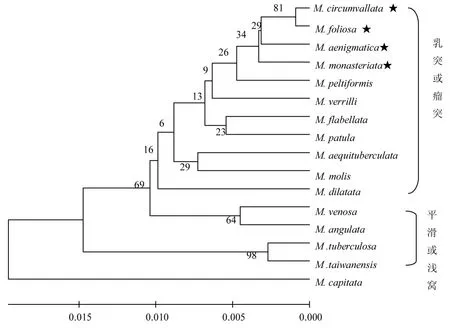
图5 蔷薇珊瑚属ITS 序列构建UPGMA 系统发育树
3 讨 论
对壁垒蔷薇珊瑚、疑惑蔷薇珊瑚、单星蔷薇珊瑚和叶状蔷薇珊瑚的ITS 序列进行两两比对,分析显示,ITS1 和ITS2 各段序列的同源性在94.1% 以上,而单星蔷薇珊瑚和叶状蔷薇珊瑚的ITS1 区的同源性高达100%.Chen 等发现鹿角珊瑚属的ITS 序列的变异性和多态性非常高[13],但本文却发现蔷薇珊瑚属的ITS序列的同源性明显很高,其变异性下降,可能的解释如同Van Oppen 等和Vollmer 等所分析的一样,即由于部分蔷薇珊瑚种通过杂交发生了基因交换并构成了网状进化模式[16-17].
4 种珊瑚和其他12 种蔷薇珊瑚的两两遗传距离显示,乳突型/瘤突型共骨珊瑚的种间遗传距离小,该类珊瑚与平滑型/浅窝型共骨珊瑚的种间遗传距离大,其分析结果与形态学分类结果较一致.和其他珊瑚属的种间遗传距离相比,蔷薇珊瑚属的种间遗传距离明显要低很多. 李文娟等用ITS 序列研究了滨珊瑚属,其种间的平均遗传距离为0.097[18],而蔷薇珊瑚属的种间遗传距离为0.025,这个也符合Van Oppen等和Vollmer 等所提出的部分蔷薇珊瑚种通过杂交发生了基因交换并构成了网状进化模式的推测[16-17].
进化树把蔷薇珊瑚分成乳突型/瘤突型和平滑型/浅窝型两大分支,而M. capitata 为单独一支.本文研究的海南4 种蔷薇珊瑚位于乳突型共骨分支内.进化树显示的系统发育关系与形态学分类结果是一致的.Forsman 等研究的6 种蔷薇珊瑚均为乳突型共骨[19],其以ITS 序列构建的系统发育关系也与本文的研究结果基本一致.
不同的分子标记方法在研究石珊瑚进化关系的结果方面存在一定的差异,和传统形态学分类的结果也常存在矛盾[20],故需根据实际研究对象和研究内容选择合适的分子标记[7].尽管在该属内的4 个物种间ITS1 和ITS2 的同源性比较高,但二者不会同时都高达100%,总是在其中1 个ITS 序列(ITS1/ ITS2)间存在差异性,这说明ITS 序列在该属内的不同种之间仍然表现出多态性.在对蔷薇珊瑚属中的某些相似物种进行细化时,以ITS 作为分子标尺,并应用整个ITS 区序列来进行鉴定,这比单纯依靠ITS1 或ITS2 序列更能准确地区分2 个相似物种.
Van Oppen 等认为,以线粒体DNA 控制区域和Pax-C 基因作为分子标记所构建的进化关系与形态学的分类基本上不存在一致,而且这2 个分子标记的分析结果也存在矛盾[16].Forsman 等的研究表明,以线粒体DNA 和核内ITS 序列作为标尺所构建的进化关系与形态学构建的进化关系存在一定的一致性[19].本研究以ITS 序列作为分子标尺所构建的进化关系与形态学构建的发育关系基本相符,可作为蔷薇珊瑚属珊瑚鉴定的依据,具有一定的适用性和可靠性.
[1]邹仁林.中国动物志·造礁石珊瑚[M].北京:科学出版杜,2001.
[2]Bruno J F,Edmunds P J. Clonal variation for phenotypic plasticity in the coral Madracis mirabilis[J]. Ecol,1997,78(7):2 177 -2 190.
[3]Fukami H,Budd A F,Paulay G,et al. Conventional taxonomy obscures deep divergence between Pacific and Atlantic corals[J]. Nature,2004,427(6 977):832 -835.
[4]Fukami H,Chen C A,Budd A F,et al. Mitochondrial and nuclear genes suggest that stony corals are monophyletic but most families of stony corals are not Order Scleractinia,Class Anthozoa,Phylum Cnidaria[J]. PloS one,2008,3(9):e3 222 .
[5]Todd P A. Morphological plasticity in scleractinian corals[J]. Biol Rev Camb Philis Soc,2008,83(3):315 -337.
[6]Forsman Z H,Barshis D J,Hunter C L,et al. Shape-shifting corals:Molecular markers show morphology is evolutionarily plastic in Porites[J]. BMC Evol Biol,2009,9(1):45 -49 .
[7]尤丰,黄晖,董志军,等.造礁石珊瑚的分子系统学研究进展[J].海洋通报,2010,29(4):459 -465.
[8]Flot J F,Magalon H,Cruaud C,et al. Patterns of genetic structure among Hawaiian corals of the genus Pocillopora yield clusters of individuals that are compatible with morphology[J]. Comptes Rendus Biologies,2008,331(3):239 -247.
[9]牛宪立,姬可平,吴群,等.rDNA ITS 区序列分子标记技术在植物学研究中的应用[J].生物信息学,2009,7(4):268-271.
[10]刘丽,李文娟,刘楚吾,等.徐闻角孔珊瑚系统发育关系[J].海洋湖沼通报,2011(4):52 -56.
[11]Takabayashi M,Carter D,Ward S,et a1. Inter-and intra-specific variability in ribosomal DNA sequence in the internal transcribed spacer region of corals. // GREENWOOD J G,HALL N J. Proceedings of the Australian Coral Reef Society 75th Anniversary Conference[C]. Brisbane:School of Marine Science,University of Queensland,1998:241 -248.
[12]Lam K,Morton B. Morphological and ITS1,5.8S,and partial ITS2 ribosomal DNA sequence distinctions between two species Platygyra (Cnidaria:Scleractinia)from Hong Kong[J]. Mar Biotechnol,2003,5(6):555 -567.
[13]Chen C A,Chang C C,Wei N V,et al. Secondary structure and phylogenetic utility of the ribosomal internal transcribed spacer 2 (ITS2 )in scleractinian corals[J]. Zool Stud,2004,43:759 -771.
[14]Wei N V,Wallace C C,et al. Analyses of the ribosomal internal transcribed spacers and the 5.8S gene indicate that extremely high rDNA heterogeneity is a unique feature in scleractinian coral genus Acropora (Scleractinian Acroporidae)[J]. Zool Stud,2006,45(3):404 -418.
[15]Stefani F,Benzoni F,Pichon M,et a1. Genetic and morphometric evidence for unresolved species boundaries in the coral genus Psammocora (Cnidaria;Scleractinia)[J]. Hydrobiologia,2008,596:l53 -172.
[16]Van Oppen M J H,Koolmees E M,Veron J E N. Patterns of evolution in the scleractinian coral genus Montipora (Acroporidae)[J]. Mar Biol,2004,144:9 -18.
[17]Vollmer S V,Palumbi S R. Hybridization and the evolution of reef coral diversity[J]. Science,2002,296:2 023 -2 025.
[18]李文娟,刘楚吾,刘丽,等.利用ITS 基因研究徐闻滨珊瑚的系统发生关系[J].海洋科学,2012(7):56 -60.
[19]Forsman Z H,Concepcion G T,Haverkort R D,et al. Ecomorph or endangered coral?DNA and microstructure reveal Hawaiian species complexes:Montipora dilatata/flabellata/turgescens & M. patula/verrilli[J]. PloS One,2010,5(12):e15 021.
[20]Huang D,Meier R,Todd P A,et al. More evidence for pervasive paraphyly in scleractinian corals:systematic study of Southeast Asian Faviidae (Cnidaria;Scleractinia)based on molecular and morphological data[J]. Mol Phylogenet Evol,2009,50(1):102 -116.

