利用高通量测序分析白花泡桐盐胁迫相关microRNAs
王园龙, 曹 林, 邓敏捷, 马一平,赵振利, 牛苏燕, 王晓丹, 范国强
(1.河南农业大学泡桐研究所,河南 郑州 450002; 2.北京师范大学地理与遥感科学学院,北京 100875)
利用高通量测序分析白花泡桐盐胁迫相关microRNAs
王园龙1, 曹 林1, 邓敏捷1, 马一平2,赵振利1, 牛苏燕1, 王晓丹1, 范国强1
(1.河南农业大学泡桐研究所,河南 郑州 450002; 2.北京师范大学地理与遥感科学学院,北京 100875)
为了鉴定和研究白花泡桐中与盐胁迫相关的microRNAs(miRNAs),构建了对照组PF2和处理组PF2-S4 2个小RNA文库。采用高通量测序技术总共鉴定出来了33个已知miRNA和80个新miRNA,在这些miRNAs中分别有27个(3个已知miRNA和24个新miRNA)差异显著表达和有45个(13个已知miRNA和32个新miRNA)差异极显著表达. 最后采用qRT-PCR技术验证了5个miRNA。
白花泡桐;microRNAs;盐胁迫;高通量测序
木本植物不仅在水土保持和维持自然界碳循环中起关键作用,而且为工业、造纸业等民生行业提供重要的原材料[1]。但是它们经常遭受到环境中包括盐胁迫在内的多种生物和非生物胁迫而导致减产甚至死亡。当今土壤盐渍化不单是干旱半干旱地区的问题,已演变成为世界范围内影响农林业发展的重要因素。全球范围内超过10%的陆地正在遭受到不同程度的盐渍化侵害而且还呈现不断加重的趋势[2]。所以对植物的抗盐胁迫研究及育种已经成为摆在植物新品种培育科研工作者面前的一道急需解决的难题。泡桐因其生长迅速,材性优良,适合农桐间作而被广泛种植在世界范围内250万hm2的农田中[3,4]。泡桐产业的发展可以为带来良好的经济效益和生态效益,但是土壤盐渍化约束着泡桐产业的发展。所以在泡桐的抗盐胁迫基础研究中首先鉴定出泡桐抗盐胁迫相关基因,将有助于弄清楚泡桐的抗盐机制,并且有针对性地进行泡桐的新品种培育。近年来,有关泡桐抗丛枝病[5-7],抗干旱[3, 6,8,9]的转录组研究取得了一定的进展,找到了与泡桐丛枝病发病及干旱相关的基因,但是至今为止还没有关于泡桐抗盐胁迫等相关分子领域的研究报道。microRNAs(miRNAs)是一类内源性非编码sRNAs,在转录后水平上起到负调控基因表达的作用[10]。植物中,miRNAs参与包括生长、发育、新陈代谢和抗胁迫等多种生物学过程[11]。目前,大量响应植物生长发育以及与胁迫相关的miRNAs被鉴定出来[10,12-14]。近年来, 范国强等[4,15, 16]采用高通量测序技术已鉴定了二、四倍体泡桐种的差异表达miRNAs并且预测分析了它们的功能。然而,至今还未有关于盐胁迫下泡桐的miRNAs的研究报道。本研究构建了处理组和对照组2个sRNA文库来鉴定分析盐胁迫下白花泡桐的miRNAs,并采用qRT-PCR方法来验证盐胁迫下白花泡桐差异表达miRNAs的准确性。以期为揭示miRNAs调控盐胁迫条件下白花泡桐体内基因表达的分子机制,阐明盐胁迫的分子机理奠定理论基础。
1 材料与方法
1.1 试验材料
植物材料为河南农业大学林木生物技术实验室培养的二倍体白花泡桐组织培养苗。在温度(25±2)℃,光强130 μmol·m-2·s-1,光照时间16 h·d-1的条件下进行愈伤组织培养30 d。而后移植到室外培养30 d后选择生长一致植株单株栽植于装有等量普通园土的营养钵中,正常生长50 d后进行盐胁迫处理。
以土壤含盐量(NaCl质量/干土质量×100%)分别为0,0.4%设置处理试验且命名为PF2和PF2-S4。每个处理组设置3个生物学重复。具体试验方法如下:(1)分别称取每钵中所需NaCl的质量,平均分成3份;(2)取1份完全溶于水后施入园土中,钵底垫有托盘,将渗水重新倒入钵中,避免盐分损失,每3 d进行1次,共计3次;(3)每次处理时,对照组PF2只加入等量水;(4)盐分添加完毕后,每2 d浇1次水,维持75%相对土壤含水量下培养20 d。处理结束后,分别摘取幼苗新梢顶端第二对叶片,立即用液氮速冻后保存于-86 ℃冰箱中备用。
1.2 白花泡桐sRNA文库的构建及测序
本试验采用pBIOZOL(杭州博日)植物提取试剂提取白花泡桐总RNA,而后使用NanoDrop 2000超微量分光光度计和Agilent 2100生物分析仪对总RNA进行质量检测。本试验的PF2和PF2-S4文库构建及测序交由深圳华大基因研究院采用高通量测序平台Illumina HiSeqTM2000完成。
1.3 白花泡桐sRNA信息分析
高通量测序所得的50 nt序列,通过过滤得到可信的目标序列,对这些序列的质量、长度及样品间公共序列进行统计。通过目标序列分类注释,可以获得样品中包含的各组分及表达量信息.对未注释的小RNA片段来进行新miRNA预测.随后对已知miRNA和新miRNA进行差异分析。
1.4 已知miRNAs的鉴定
利用SOAP软件将长度在18~30 nt的高质量片段(clean reads)匹配到白花泡桐转录组(http://trace.ncbi.nlm.nih.gov/Traces/sra/)。随后完美匹配序列用Blast软件(http://www.ncbi.nlm.nih.gov/staff/tao/URLAPI/blastall/)与NCBI GeneBank (http://www.ncbi.nlm.nih.gov/)和Rfam 11.0 (http://rfam.janelia.org/)数据库比对,筛选和去除同rRNAs, scRNAs, snoRNAs, snRNAs, tRNA等相关的序列。将这2文库中剩下的序列比对到miRBase 21.0(http://www.mirbase.org/ftp.shtml)中的已知植物miRNAs上鉴定白花泡桐已知miRNAs。
1.5 新miRNAs的鉴定
将未注释的sRNA用于白花泡桐新miRNA的预测。使用mireap软件 (http://sourceforge.net/projects/mireap/files/mireap/)预测2个文库中新miRNA,绘制新miRNA的二级结构图。如果序列存在Meyers等人描述的标准:序列的长度范围在18~25 nt之间,最低自由能(minimum free energy,MFE)不高于-18 kcal·mol-1,miRNA与miRNA*之间的对称度为7 nt、最长距离为100 nt、碱基配对数为10 nt,可以看作白花泡桐中新miRNA[17]。
1.6 2个文库中miRNA的差异表达分析
根据每个miRNA分别在PF2和PF2-S4库中的相对表达丰度确定miRNA的差异表达,如果miRNA的fold change值 ≥1或者≤-1且P-values≤0.05,定义其在2库中差异显著表达;如果miRNA的fold change值≥1或者≤-1且P-values≤0.01,则定义其在2库中差异极显著表达。
具体计算方法如下:
(1)miRNA在各自库中表达量归一化值=miRNA表达量/clean reads总数*1 000 000,如果结果是0,将其变更为0.01。
(2)某一miRNA的fold change值=log2(PF2-S4库中miRNA表达量的归一化值/PF2库中miRNA表达量的归一化值)。
(3)P-values计算公式。
其中:N1代表PF2文库序列总数;N2代表PF2-S4文库序列总数;x代表某一miRNA在PF2文库的表达量;y代表某一miRNA在PF2-S4文库的表达量。
1.7qRT-PCR验证差异表达miRNAs
为了验证盐胁迫下白花泡桐中差异表达miRNAs及其靶基因的存在和表达模式,随机选择了5个差异表达miRNA进行qRT-PCR验证。将生长状况良好且生长一致的白花泡桐组培苗进行0.4%浓度的盐胁迫处理,处理方法同1.1,对对照组和处理组叶片进行总RNA的提取,且总RNA提取与检测方法同1.2。
PCR操作程序参照SuperScriptIIIplatinumSYBRGreenone-stepqRT-PCRkit(Invitrogen,USA)试剂盒说明进行。使用CFX96TMReal-TimePCRSystem(Bio-Rad,Hercules,CA)进行实验。其反应体系总体积为20μL,其中,10μLSSIIIandplatinumMix、0.4μL5×buffer、0.4μLReverseprimer(10μmol·L-1)、0.4μLForwardprimer(10μmol·L-1)、0.4μLReverseuniverseprimer(10μmol·L-1)、500ngRNA,最后,用DEPC处理过的无菌水补至20μL。扩增程序为:50 ℃,3min;95 ℃,5min;40循环(95 ℃,15s;55 ℃,30s); 40 ℃, 10min。每个样品进行3次技术重复。数据处理使用2-△△CT方法计算相对表达量,以内参U6rRNA进行标准化,将白花泡桐(对照样品PF2)的表达量作为“1”,计算出处理样品的表达量,并作图分析。
2 结果与分析
2.1 白花泡桐sRNA文库的构建及测序
根据邓敏捷等人的不同质量浓度盐处理下白花泡桐生理响应研究结果,选用0.4%土壤含盐量作为测序处理组处理质量浓度[18]。利用Solexa/Illumina测序技术分别构建PF2和PF2-S4 2个小RNA的cDNA文库。在PF2和PF2-S4库中,分别有12 248 047条和11 942 362条rawreads。然后分别去除测序质量较低的序列、带有5′接头污染的序列、无插入片段的序列、没有3′接头序列、包含polyA的序列、小于18nt的片段,共得到12 060 908条和11 855 951条cleanreads。在2个样品间共有序列种数1 176 068条,占库中所有序列百分数为16.53%;共有序列总量17 053 689条,占库中所有序列百分数为71.30%。测序结果表明2个样品间序列种类上的差异比较大,但公共部分的序列表达比较集中,说明了2个样品在测序整体上的一致性是比较好的。
对PF2和PF2-S4文库中sRNA进行长度作图分析(图1),2个文库中24nt长度的sRNA峰值最高,分别占到sRNA总量的43.70%(PF2)和41.76%(PF2-S4);其次是21nt长度序列,分别占到sRNA总量的22.06%(PF2)和24.84%(PF2-S4)。相似的结果也出现在毛泡桐[4]和南方泡桐[15]等其他泡桐种的测序结果中。同时长度处于20~24nt间的sRNA总数分别占其sRNA总量的92.96%(PF2)和91.88%(PF2-S4),表明了2个样品的sRNA文库中均富含miRNA。
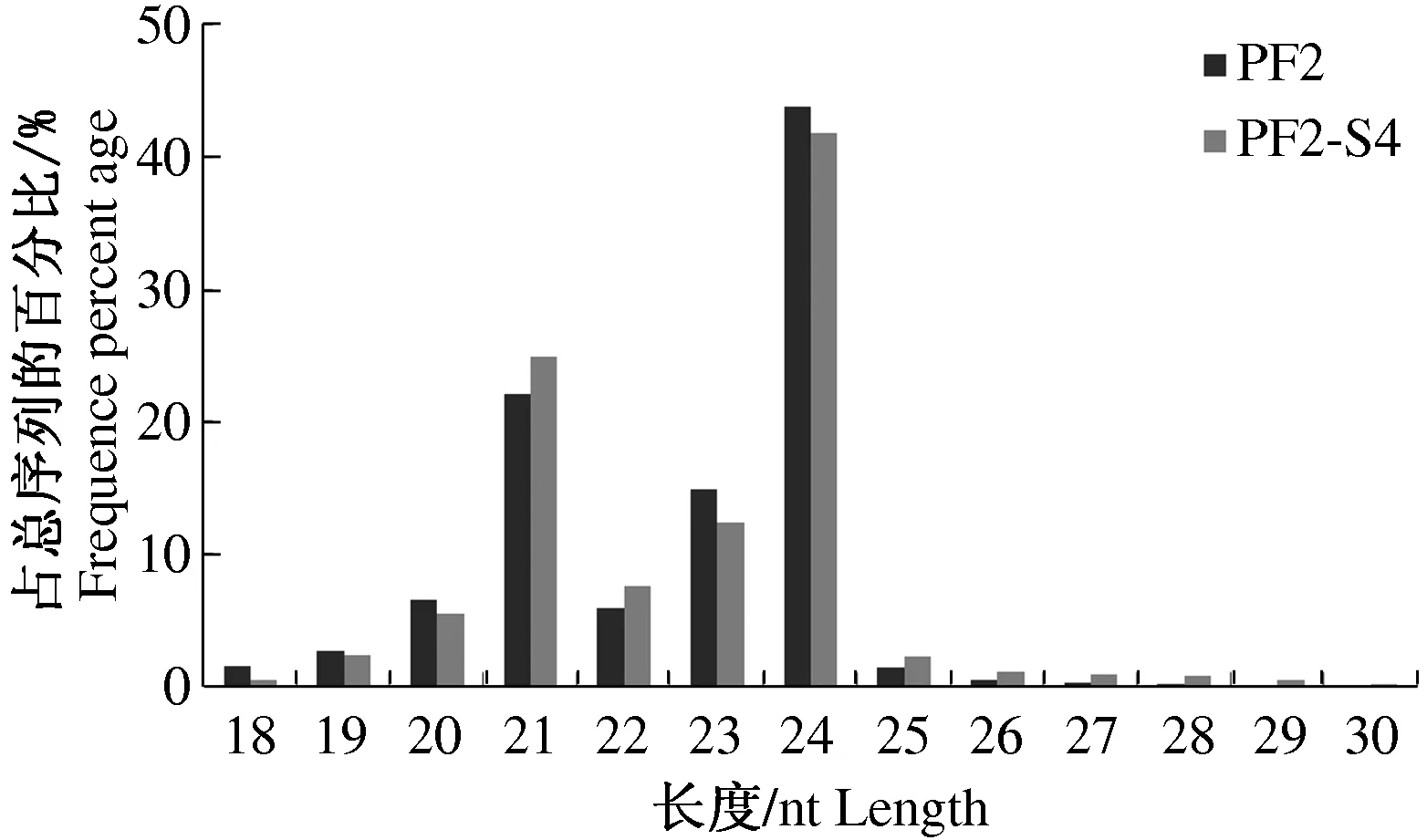
图1 对照组(PF2)和处理组(PF2-S4)白花泡桐样品中小RNAs的长度分布
2.2 sRNA分类注释
将sRNA匹配到Genbank数据库和Rfam数据库中进行sRNA的分类注释确保每一个unique sRNA都有唯一的注释。按照Genbank数据库的优先级大于Rfam数据库且rRNAetc>known miRNA> piRNA>repeat>exon>intron的优先级顺序将sRNA遍历,没有比上任何注释信息的sRNA用unannote表示。其中分类注释结果中的rRNA总量可以作为一个样品的质控标准,植物样品中的rRNA总量所占比例应低于60%(表1)。PF2和PF2-S4中rRNA总量所占比例分别为3.43%和4.22%,远远低于其质控标准。说明2个样品的sRNA是测序所得的真实可信的数据。
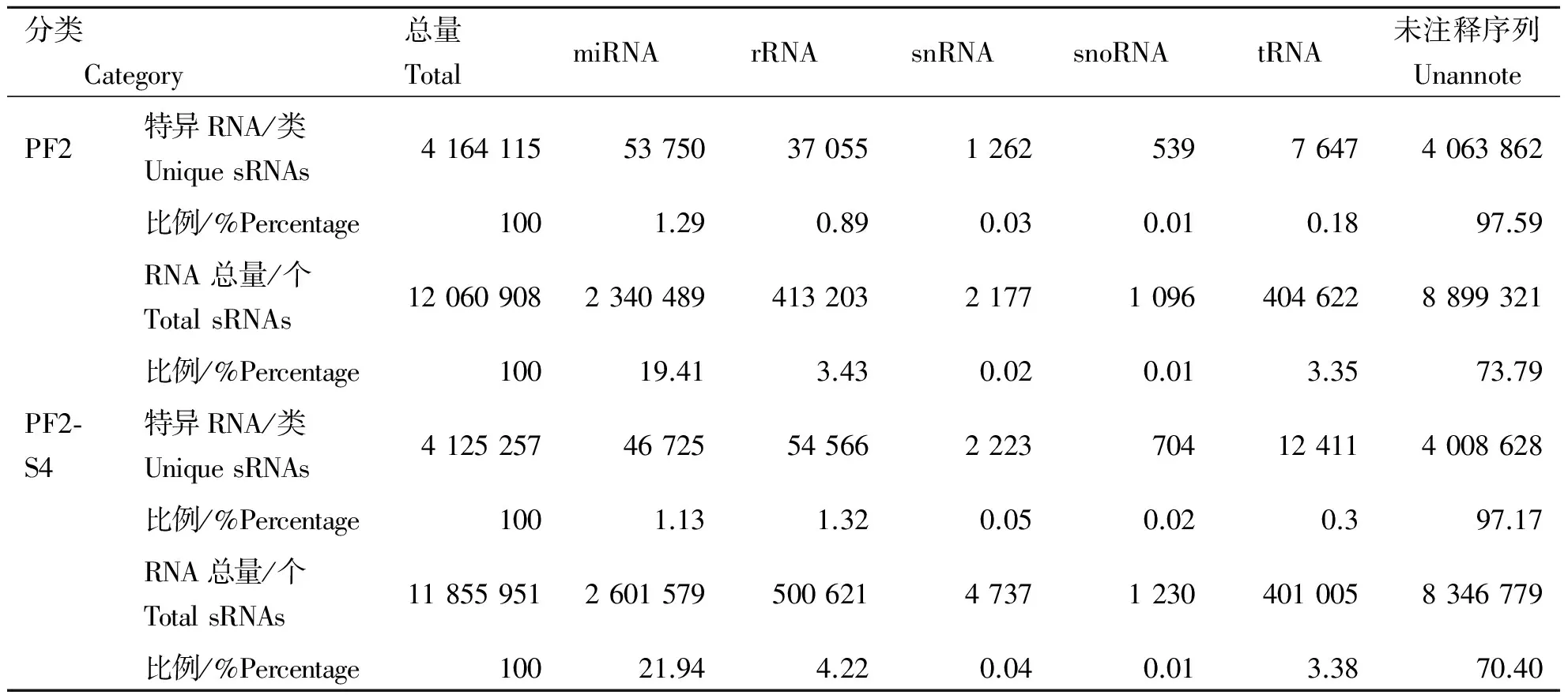
表1 白花泡桐对照组(PF2)与处理组(PF2-S4)小RNA分类
2.3 白花泡桐中已知miRNAs的鉴定
将PF2和PF2-S4库中完全匹配到白花泡桐转录组的序列与miRBase 21.0数据库成熟的miRNA序列进行同源性比对,鉴定出白花泡桐已知miRNA序列33条,其中对照组(PF2)和处理组(PF2-S4)中均特有6条,2库共有21条。鉴定出的33个已知miRNA中,有20个miRNAs分别属于12个家族,13个miRNAs的家族未知。平均前体长度和平均最低自由能分别为155 nt和-58.2 kcal·mol-1。MIR156家族、MIR166家族、MIR167-1家族、MIR168家族和MIR169-1家族在2库中均有较高的表达量。不同的家族表达量差异较大,而且即使是同一家族中的不同成员,其表达量也有较大差异。比如MIR167-1家族中pfo-miR167d和pfo-miR167e在2 个文库中的表达量明显低于其他3个家族成员。结果中有趣的是已知miRNA中只有20 nt,21 nt和22 nt这3个序列长度,长度为21 nt的miRNA最多,共有21个,其次有9个22 nt长的miRNA。这个结果与其他植物的研究结果一致,即长度为21 nt 的miRNAs 表达丰度最高[19],这也说明了鉴定出的白花泡桐中和与盐胁迫相关的已知miRNAs的准确性。
2.4 白花泡桐中新miRNAs的鉴定
如表1所示PF2库中没有注释(unannote)的sRNA有4 063 862种,共计8 899 321条;在PF2-S4库中没有注释的sRNA有4 008 628种,共计8 346 779条。利用Mireap软件对2个库中unannote序列进行miRNA二级结构等特征的筛选,共获得新miRNA序列80条,其中PF2库中特有序列34条,PF2-S4库中特有序列29条,PF2库和PF2-S4库中共有序列17条。新miRNA的序列长度在20 nt到23 nt之间,其中长度为21 nt的新miRNA数量最多,有37条。平均前体长度和平均最低自由能分别为156 nt和-47.7 kcal·mol-1。与预测出来的2个库中已知miRNAs的表达量相比,新miRNAs的表达量总体上呈现较低的水平。
2.5 PF2和PF2-S4文库中miRNAs的差异表达分析
对白花泡桐miRNA在PF2库和PF2-S4库中表达量进行差异分析。与对照组PF2库相比,在处理组PF2-S4库中有27个miRNAs(3个已知miRNA和24个新miRNA)差异显著表达;45个miRNAs(13个已知miRNA和32个新miRNA)差异极显著表达(表2)。这些显著差异和极显著差异表达的miRNAs可能在白花泡桐的抗盐胁迫中发挥重要的调控作用。
2.6 qRT-PCR验证差异表达miRNA
为了进一步验证测序结果的准确性,本研究随机选取了5个差异表达的胁迫相关miRNAs进行实时荧光定量PCR验证,分析盐胁迫相关miRNAs的相对表达差异(图2)。结果显示,与测序结果相比,除pfo-mir890之外,pfo-miR159、pfo-miR167d、pfo-miR5021a、pfo-mir5301的表达量变化与测序的结果完全相同,说明测序结果的可靠性。
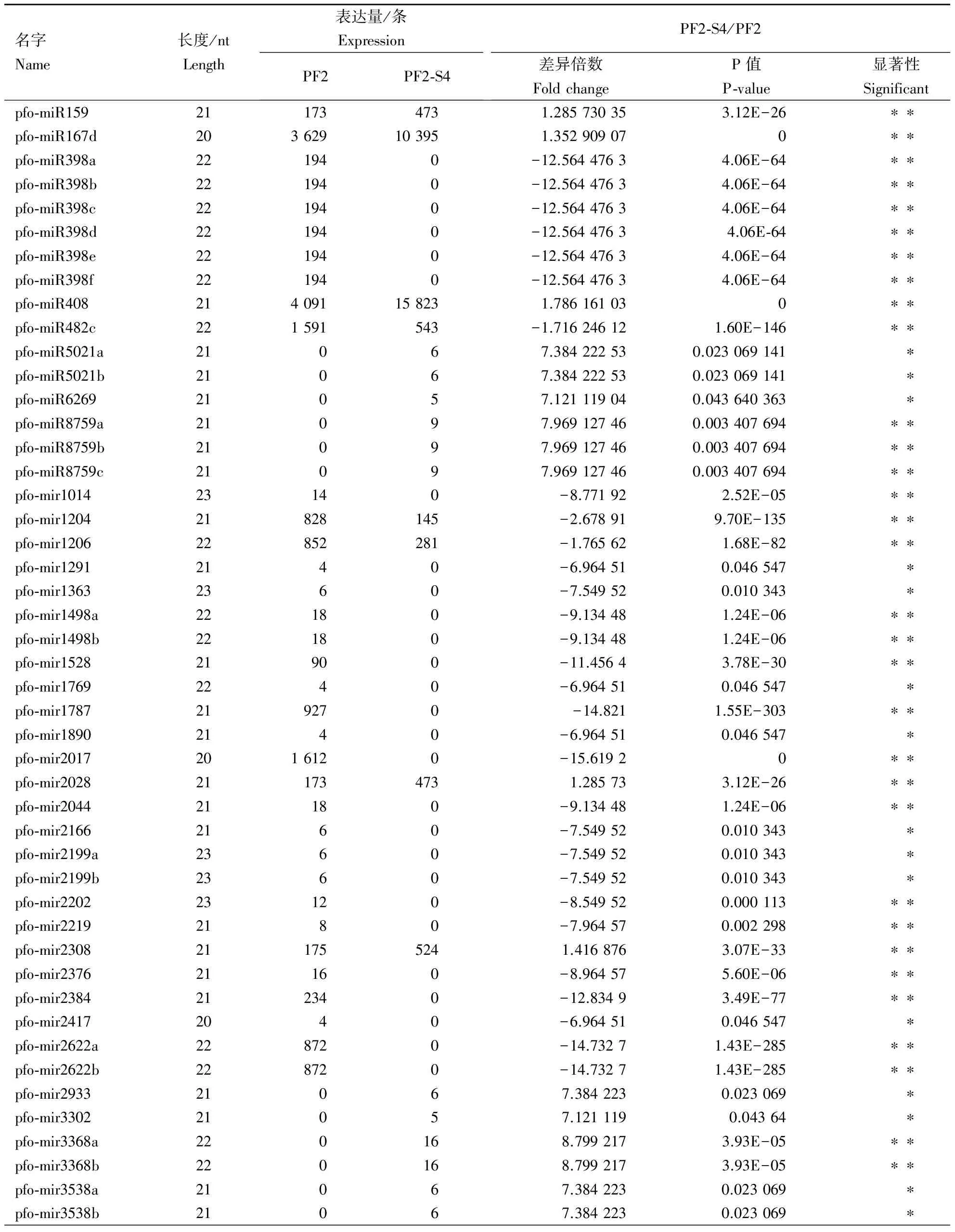
表2 PF2和PF2-S4文库中miRNA的差异表达分析

续表 Continuing table
注: * :差异显著;**:差异极显著。
Note:*:significant difference; **:more significant difference.
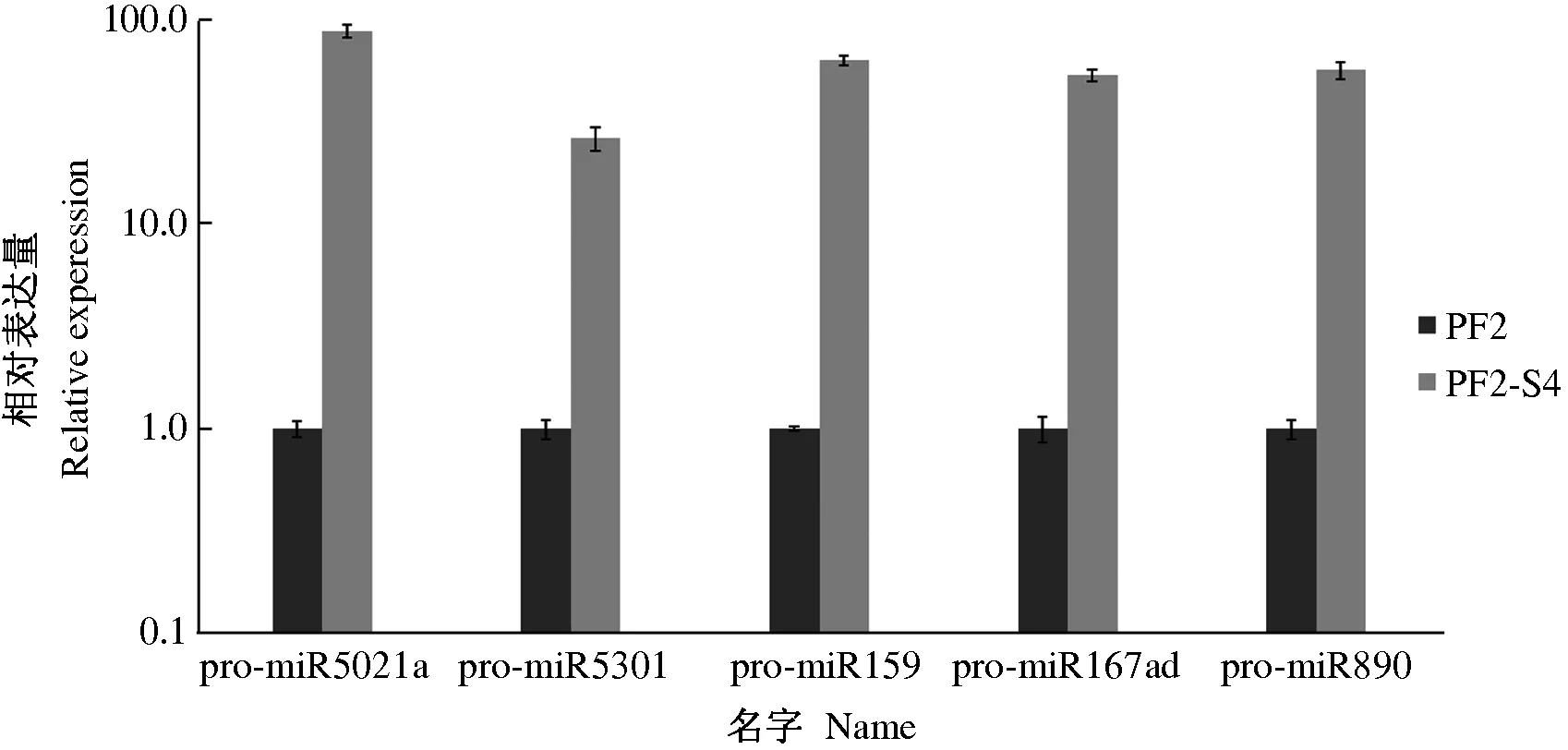
图2 差异表达miRNAs的qRT-PCR验证
3 结论与讨论
本试验采用高通量测序技术对PF2和PF2-S4 2个sRNAs文库中的已知miRNA和新miRNA进行鉴定。通过对2个文库的小RNA长度分布图进行分析得出了2样品中均富含miRNA,说明测序结果是值得信赖的;尽管有一些sRNA可能是由降解途径产生,但PF2和PF2-S4的长度分布没有明显的差异,说明了sRNAs的长度分布可能独立于盐胁迫而被植物其他机制控制[20]。
根据PF2和PF2-S4中miRNAs的表达分析,找到了72个与盐胁迫相关的miRNA。其中,一些miRNAs在白花泡桐正常的生长发育中不容易检测到,但在受到盐胁迫时会受到特异性表达而被鉴定出来;相反,一些miRNA在白花泡桐正常的生长发育中可以特异表达,但受到盐胁迫后会被抑制从而在处理后不能被鉴定出来;还有一些miRNAs在受到盐胁迫前后发生差异表达,这些差异显著表达和差异极显著表达的miRNA通过上调或者下调其表达量来响应盐胁迫。对鉴定出来的miRNAs进行差异表达分析,发现分别在PF2和PF2-S4库中共12个特有的已知miRNA均差异显著表达或差异极显著表达;2个库中共63个特有的新miRNA中,有52个miRNAs差异显著表达或差异极显著表达,这些miRNAs很可能在泡桐抗盐胁迫相关的过程中发挥重要的调控作用。
总之,本试验是国内外首次基于泡桐转录组对白花泡桐抗盐胁迫miRNA进行研究,采用高通量测序分析方法鉴定出了白花泡桐与盐胁迫相关的miRNA。然后采用qRT-PCR方法验证了白花泡桐miRNAs的存在。通过miRNA在2个小RNAs文库的差异表达情况,找到与盐胁迫相关的miRNA,有关白花泡桐抗盐胁迫的miRNA研究将为研究木本植物的耐盐机制提供直接和有效的参考。
[1] ATES S, NI Y, AKGUL M, et al. Characterization and evaluation ofPaulowniaelongotaas a raw material for paper production [J]. African Journal of Biotechnology, 2008, 7(22): 4153-4158.
[2] 孙文喜, 孙文鑫, 周大旺, 等. 植物抗盐性生理生化研究进展及提高植物抗盐性对策 [J]. 农业科技通讯, 2013 (6): 174-178.
[3] DONG Y, FAN G, DENG M, et al. Genome-wide expression profiling of the transcriptomes of fourPaulowniatomentosaaccessions in response to drought [J]. Genomics, 2014, 104(4): 295-305.
[4] FAN G, ZHAI X, NIU S, et al. Dynamic expression of novel and conserved microRNAs and their targets in diploid and tetraploid ofPaulowniatomentosa[J]. Biochimie, 2014, 102: 68-77.
[5] CAO X, FAN G, DENG M, et al. Identification of Genes Related toPaulowniaWitches’ Broom by AFLP and MSAP [J]. International Journal of Molecular Sciences, 2014, 15(8): 14669-14683.
[6] CAO X, FAN G, ZHAO Z, et al. Morphological changes ofPaulowniaseedlings infected phytoplasmas reveal the genes associated with witches' broom through AFLP and MSAP [J]. PloS one, 2014, 9(11): 533-542.
[7] LIU R, DONG Y, FAN G, et al. Discovery of Genes Related to Witches Broom Disease inPaulowniatomentosaxPaulowniafortuneiby a De Novo Assembled Transcriptome [J]. Plos One, 2013, 8(11):238-247.
[8] DONG Y, FAN G, ZHAO Z, et al. Transcriptome Expression Profiling in Response to Drought Stress inPaulowniaaustralis[J]. International Journal of Molecular Sciences, 2014, 15(3): 4583-4607.
[9] CUI L, SHAN J, SHIMIN G, et al. The miR156-SPL9-DFR pathway coordinates the relationship between development and abiotic stress tolerance in plants [J]. Plant Journal, 2014, 80(6): 1108-1117.
[10]LI B, DUAN H, LI J, et al. Global identification of miRNAs and targets inPopuluseuphraticaunder salt stress[J]. Plant Molecular Biology, 2013, 81(6): 525-539.
[11]SHUAI P, LIANG D, ZHANG Z, et al. Identification of drought-responsive and novelPopulustrichocarpamicroRNAs by high-throughput sequencing and their targets using degradome analysis [J]. Bmc Genomics, 2013, 14:233-241.
[12]BARRERA-FIGUEROA B E, GAO L, WU Z, et al. High throughput sequencing reveals novel and abiotic stress-regulated microRNAs in the inflorescences of rice [J]. Bmc Plant Biology, 2012, 12(4).132-145.
[13]HAN J, XIE H, SUN Q, et al. Bioinformatic identification and experimental validation of miRNAs from foxtail millet (Setariaitalica) [J]. Gene, 2014, 546(2): 367-377.
[14]JIA L, ZHANG D, QI X, et al. Identification of the Conserved and Novel miRNAs in Mulberry by-High-Throughput Sequencing [J]. Plos One, 2014, 9(8):409-417.
[15]NIU S, FAN G, XU E, et al. Transcriptome/Degradome-Wide Discovery of MicroRNAs and Transcript Targets in TwoPaulowniaaustralisGenotypes [J]. Plos One, 2014, 9(9):736-745.
[16]NIU S, FAN G, ZHAO Z, et al. High-throughput sequencing and degradome analysis reveal microRNA differential expression profiles and their targets inPaulowniafortunei[J]. Plant Cell Tissue and Organ Culture, 2014, 119(3): 457-468.
[17]GERMAN M A, PILLAY M, JEONG D H, et al. Global identification of microRNA-target RNA pairs by parallel analysis of RNA ends [J]. Nature Biotechnology, 2008, 26(8): 941-946.
[18]邓敏捷, 张晓申, 范国强, 等. 四倍体南方泡桐和四倍体白花泡桐对盐胁迫的生理响应研究 [J]. 河南农业大学学报, 2013, 47(6): 698-702.
[19]CHEN X. Small RNAs and Their Roles in Plant Development [J]. Annual Review of Cell and Developmental Biology, 2009,25: 21-44.
[20]HACKENBERG M, GUSTAFSON P, LANGRIDGE P, et al. Differential expression of microRNAs and other small RNAs in barley between water and drought conditions [J]. Plant Biotechnology Journal, 2015, 13(1): 2-13.
(责任编辑:蒋国良)
Analysis of salt stress-responsive microRNAs inPaulowniafortuneiby high-throughput sequencing
WANG Yuanlong1, CAO Lin1, DENG Minjie1, MA Yiping2,ZHAO Zhenli1,NIU Suyan1, WANG Xiaodan1, FAN Guoqiang1
(1.Institute of Paulownia, Henan Agricultural University, Zhengzhou 450002, China; 2.School of Geography,Beijing Normal University,Beijing 100875,China)
To identify and investigate salt-related microRNAs(miRNAs) inPaulowniafortunei(P.fortunei), we constructed and sequenced two small RNA libraries (PF2 and PF2-S4). A total of 33 conserved and 80 novel miRNAs were identified in the two libraries by high-throughput sequencing. Among these miRNAs, 27 miRNAs (3 conserved and 24 novel) were differentially expressed and 45 miRNAs (13 conserved and 32 novel) were significantly differentially expressed in response to salt stress inP.fortunei, respectively. Finally, 5 of the miRNAs were confirmed by qRT-PCR.
Paulowniafortunei; microRNAs; salt stress; high-throughput sequecning
2015-01-20
王园龙(1989-),男,河南濮阳人,硕士研究生,主要从事泡桐生物技术方面的研究。
范国强(1964-),男,河南禹州人,教授,博士生导师。
1000-2340(2015)04-0461-07
S792.43
A

