地中海拟无枝酸菌“硝酸盐效应”的研究进展
邵志会,赵维,王颖,丁晓明,王金,姜卫红,赵国屏,
利福霉素是安莎类抗生素 (Ansamycins)的一个亚群,临床上广泛地用于治疗由分枝杆菌 Mycobacterium、肺炎球菌 Pneumococcus及其他革兰氏阳性菌引起的各种感染[1-5]。利福霉素可以特异性地抑制依赖于DNA的RNA聚合酶的活性,使病原菌不能正常合成RNA产物,从而达到抑菌目的。地中海拟无枝酸菌Amycolatopsis mediterranei是工业上用来生产利福霉素的稀有放线菌。该菌在1957年首次从地中海沿岸的法国Saint Raphael附近土样中被分离出来,被命名为地中海链霉菌Streptomyces mediterranei[6],后来又被Thiemann等重新鉴定为地中海诺卡氏菌Nocardia mediterranei[7]。直到20世纪80年代中期,Lechevalier等根据其细胞壁的组成缺乏分支酸以及对诺卡氏菌属Nocardia和红球菌属 Rhodococcus的噬菌体不敏感等特征最终将其归到一个新的属,拟无枝酸菌属Amycolatopsis[8]。最初地中海拟无枝酸菌的发酵液分离到了利福霉素 A、B、C、D、E五个组分,其中最主要是 B组分[6];后来人们又从不同的生产菌种中分离到了S、O以及SV等组分[9]。这些组分中活性最高的是利福霉素SV;而利福霉素B虽然活性较小但其产量高,而且可以通过化学反应转化为其他组分,如S、O以及SV等[10-11]。目前,工厂中主要生产利福霉素B或SV,然后在此基础上进行化学修饰来获得利福平、利福喷丁等利福霉素衍生药物。
1 “硝酸盐效应”的发现
20世纪 70年代末,焦瑞身先生等从上海第三制药厂处获得一株高产利福霉素 SV的菌株——地中海拟无枝酸菌 U32。通过传统的育种手段 (如紫外及亚硝基胍 (NTG) 诱变) 多轮筛选后,获得一株产量更高的菌株NG12-4 (注:按照当时的分类标准,U32及 NG12-4最初被称为地中海诺卡氏菌)。由于硝酸盐促进抗生素的合成是一个较普遍的现象,上海第三制药厂的张永昌等发现在工业发酵利福霉素 SV的过程添加硝酸钾能够提高其产量[12]。焦瑞身等进一步通过发酵优化实验确定在发酵起始添加终浓度为 0.8%(80 mmol/L) 的硝酸钾能使地中海拟无枝酸菌NG12-4利福霉素SV的产量提高170%。为了确定究竟是硝酸根离子还是钾离子的影响,早期的研究人员设计了严谨的对照实验,证明了虽然培养基中的钾离子对利福霉素的合成也有部分促进作用,但硝酸根离子是促进利福霉素 SV高产的主要因素[13]。此外,研究还发现硝酸盐不仅能够明显地改变菌体的生理代谢,如引起硝酸还原酶、异柠檬酸脱氢酶、苹果酸脱氢酶等多个酶的活力发生变化;而且还影响了菌体内的脂肪酸含量:不添加 KNO3的菌体内脂肪酸为 13.6%;而添加硝酸盐后,菌体的脂肪酸含量则降到5.7% (图1)。
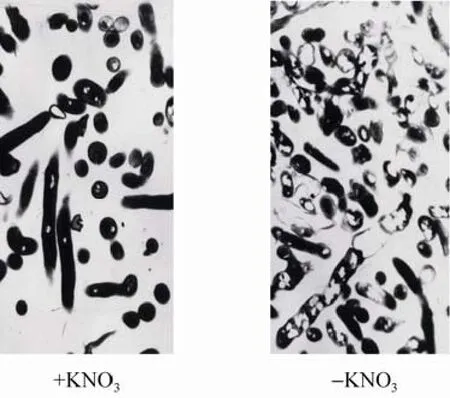
图 1 有无硝酸盐培养条件下地中海拟无枝酸菌 U32菌体电镜下的形态 (本图引自文献[5])Fig. 1 Mycelium morphology of A. mediterranei U32 under electron microscope in the condition with or without nitrate (cited from reference [5]).
2 “硝酸盐效应”分子机制的初步研究
2.1 利福霉素生物合成途径的鉴定
1973年Prelog等确定了利福霉素的化学结构,发现该类化合物为29个碳原子的大环内酰胺,其立体脂肪链桥跨接平面芳香核的结构形成了典型的提桶状结构,并将具有这种大环结构的抗生素称为安莎霉素[17]。此后,许多科研人员对利福霉素的生物合成途径进行了探索:White等曾用同位素标记的丙酸和乙酸阐明了利福霉素大环部分的来源问题[18];焦瑞身等在20世纪70年代末通过诱变方法筛选到利福霉素的非活性突变株,并进行一系列的生化互补实验,从而为进一步阐明利福霉素SV的生物合成途径奠定了基础[19-20]。通过菌体洗涤实验,焦瑞身等发现谷氨酰胺促进利福霉素 SV合成的作用远比谷氨酸显著,而且这一促进作用不受GS专一性抑制剂重氮基-5-氧-L-正亮氨酸(DON) 的抑制,而谷氨酸和天冬酰胺对利福霉素SV的促进作用则受到DON的抑制。在分别利用稳定同位素标记的谷氨酰氨-CO15NH2与谷氨酸-15NH2掺入到利福霉素SV并进行终产物比较实验后发现,前者是利福霉素合成更直接的前体,它促进了利福霉素芳香前体 (AHBA) 的合成[21]。根据以上结果,本实验室提出了利福霉素分子中的氮原子来源于谷氨酰胺。此后,通过稳定性15N标记的硝酸钾掺入实验证明利福霉素中的氮原子是来源于硝酸钾,并经谷氨酰胺最终掺入利福霉素中[22],这也是第一次明确了利福霉素合成中氮原子的掺入途径。
关于利福霉素合成的芳香环碳骨架部分的来源,最初 Ghisaba等认为其主要来自于莽草酸途径[23]。Guo等在2002年又对A. mediterranei S699利福霉素B的生物合成途径进行了修正,他们通过体外实验发现多糖合成的前提物 UDP-葡萄糖和谷氨酰胺经过多步反应可以得到亚氨基-4-磷酸-赤藓糖 (Imino E-4-P)。在此基础上,磷酸烯醇式丙酮酸 (PEP) 和亚氨基-4-磷酸-赤藓糖可以进一步合成得到 5-脱氧-5-氨基-3-脱氢莽草酸(Amino DHS),后者在3-氨基5-羟基苯甲酸合成酶作用下转化为3-氨基5-羟基苯甲酸 (AHBA)[24]。AHBA合成酶 (ahbas) 是合成前体AHBA (又称C7N母核) 的关键酶。黄健强等利用来源于利福霉素B产生菌A. mediterranei S699的ahbas基因作为探针从U32基因组文库中筛选到了ahbas基因 (rifK,AMED_0627);中断了该基因的突变菌株基本不产生利福霉素,证明了AHBA是利福霉素生物合成过程中必不可少的前体[25]。
在利福霉素生物合成途径方面,1998年Floss和Hutchinson研究组率先从地中海拟无枝酸菌S699中克隆、测序了利福霉素B的生物合成基因簇,并在实验及前期文献分析的基础上,对利福霉素B的合成途径进行了详细描述[26]。2010年,本实验室对A. mediterranei U32进行了全基因组序列测定和注释,进一步明确了利福霉素 SV生物合成及前体供给的途径 (图2)[27]。U32的染色体大约为10.2 Mb,环形,含有9 228个基因以及 52个tRNA。通过全基因组序列分析并结合以前的实验结果研究定位了与利福霉素SV生物合成及前体供给相关的大多数基因,并鉴定了从利福霉素 SV到 B的关键 P450酶(AMED_0653,Rif16)[27]。
2.2 “硝酸盐效应”机制的解析
通过对利福霉素生物合成过程中关键酶的活力测定和相关基因的转录分析,我们提出硝酸盐通过促进利福霉素生物合成前体的供给和生物合成酶系的表达来提高菌体利福霉素的产量的假说。
2.2.1 利福霉素前体
利福霉素的前体主要包括 AHBA (碳骨架和氮原子) 和二碳/三碳延伸单位 (图2)。
1) 碳骨架合成:UDP-葡萄糖是 AHBA 合成的重要底物[24]。U32基因组的工作对催化从1-P-葡萄糖合成 UDP-葡萄糖的编码基因 galU(AMED_8311) 进行了注释[27]。最近通过RNA-seq实验,我们对该基因的转录水平也进行了分析,发现硝酸盐能够促进galU的转录上调近7倍左右 (Manuscript under revision),UDP-葡萄糖和谷氨酰胺经多步反应可得到利福霉素合成的中间前体AHBA[28]。
2) 氮原子掺入:虽然利福霉素中的氮原子来源于谷氨酰胺,但是焦瑞身等早期通过菌体洗涤实验发现蛋白胨等氮源不能代替硝酸盐来提高利福霉素产量,因此,硝酸盐的作用并不仅仅作为氮源被菌体利用[13]。在添加蛋白质合成抑制剂——氯霉素的培养条件下,硝酸盐便不能促进利福霉素的合成[29],这说明氯霉素抑制了硝酸盐所诱导的酶系的合成,并进而抑制了“硝酸盐效应”。总结下来,我们认为硝酸盐一方面作为氮源提供利福霉素生物合成中所需的氮原子;同时,也通过某种途径诱导一系列关键酶的表达调控,从而协同调控初级、次级代谢,最终促进利福霉素产量的提高。
硝酸盐还原酶是硝酸盐作为氮源利用的关键酶,催化硝酸盐同化的第一步反应。实验表明,利福霉素高产和低产菌株的硝酸还原酶活力存在差异;而且在以硝酸盐为唯一氮源的培养基上,低产菌株生长较差。这表明硝酸同化酶与利福霉素产量之间可能存在一定的相关性[30]。通过对硝酸还原酶的酶学性质进行研究,发现A. mediterranei U32的硝酸还原酶同其他同化型硝酸还原酶相似,而且其合成受铵盐的阻遏,受硝酸盐的诱导[31]。随后,通过对U32的硝酸盐同化基因簇 (Nitrate assimilatory cluster, nas)进行克隆和鉴定,发现其由nasACKBDE等6个基因组成,其中nasAC编码硝酸还原酶,nasK编码亚硝酸盐排出蛋白,而nasBDE则编码亚硝酸还原酶[32]。U32中U32唯一的同化型硝酸盐还原酶是NasA,其敲除突变株在以硝酸盐为唯一氮源的培养基上不能生长;同时,也失去了“硝酸盐效应”,说明硝酸盐同化在“硝酸盐效应”中起着至关重要的作用,并为利福霉素生物合成提供唯一的氮原子。另一方面,硝酸盐仍然能够激活突变株中利福霉素合成基因簇及其他受硝酸盐诱导的相关基因的转录[32],这也在一定程度上证明了我们前面提到了关于硝酸盐作用机制的猜想,即硝酸盐自身 (或其他相关中间代谢物) 可能扮演着信号分子的作用。
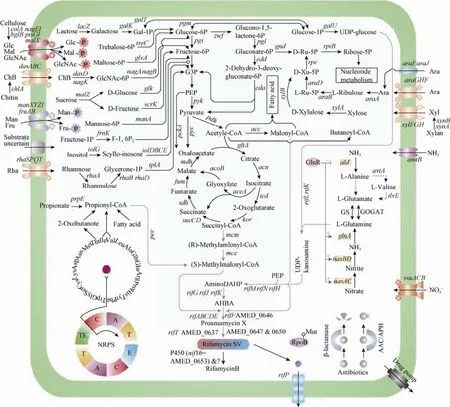
图2 地中海拟无枝酸菌U32的利福霉素SV合成途径 (本图引自文献[27])Fig. 2 RifamycinSV biosynthesis pathway of A. mediterraneiU32 (cited from reference [27]).
硝酸盐在还原为氨之后,会被谷氨酰胺合成酶 (GS) 进一步固定,生成谷氨酰胺,从而进入氮代谢循环;同时,谷氨酰胺也能为利福霉素提供唯一的氮原子。在 U32中存在两条氨同化途径,即丙氨酸脱氢酶 (AlaDH) 和谷氨酰胺合成酶/谷氨酸合酶 (GS/GOGAT) 途径[33-34]。在硝酸盐条件下,菌体主要采用GS/GOGAT途径,并且 GS的酶活与利福霉素的产量之间存在正相关性 (图3);而AlaDH则与利福霉素的产量呈负相关性[34-35]。除硝酸盐外,在其他氮源条件如以脯氨酸、硝酸钾、丙氨酸、谷氨酸、尿素及谷氨酰胺为唯一氮源下,这种相关性仍然存在[35]。我们推测合理的解释是:在高 GS活力情况下,菌体内的谷氨酰胺(Gln) 浓度比较高,因此能够保证利福霉素合成中氮原子的充分供给,从而导致利福霉素的产量较高。
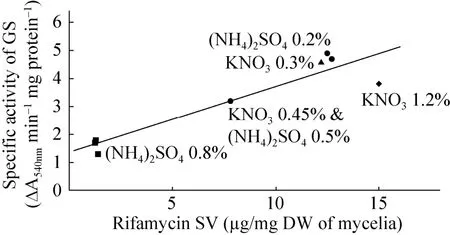
图3 不同氮源种类及浓度培养地中海拟无枝酸菌U32的GS活力和利福霉素的相关性 (本图引自文献[21])Fig. 3 Correlation between yield of rifamycin SV and specific activity of GS in A. mediterranei U32 cultivated in different sort and concentration of nitrogen sources (cited from reference [21]).
在U32中存在有6个GS编码基因,但唯有 glnA (AMED_1229) 编码有生物功能的GS[36]。U32的GS属于GSI类,由12个亚基组成,其中每 6个亚基呈六角形排列,且两个六角形结构重叠排列。早期研究发现,与常规的GS受到腺苷化修饰不同,U32中GS虽然含有保守的腺苷酰化调节区域,其不被腺苷化修饰调控,其活力调节机制主要发生在酶量水平上。其具体原因未知,推测U32在长期的菌种诱变过程中发生突变,使腺苷酰化调节的通路受到影响[34,36-37]。最近的转录组实验结果发现当添加硝酸盐之后,U32中glnA的转录上调13倍,而编码AlaDH的基因ald (AMED_7939)[38]则下调41倍之多,与早期测得的酶活变化趋势完全一致[28]。
3) AHBA的合成:UDP-葡萄糖和谷氨酰胺首先在 RifL和 RifK 的催化下合成UDP-Kanosamine,然后在RifM、RifN和RifH的催化下进一步合成 AminoDAHP,最后在RifG、RifJ和RifK的催化下合成AHBA[24]。编码这些酶类的基因全部集中在利福霉素生物合成基因簇中[27]。近期的转录组测序工作发现硝酸盐能够促进基因簇中的大部分基因 (包括AHBA合成中涉及的基因) 在整个发酵过程中都保持高表达;而没有添加硝酸盐的菌体中,该基因簇只在对数前期高表达,随着培养时间的延长,转录水平急剧下降,这也与硝酸盐促进AHBA生物合成的结论一致[28]。
4) 二碳/三碳单位延伸:除了芳香部分的AHBA外,利福霉素的合成还由 3个乙酸和 8个丙酸分子分别通过丙二酰CoA和甲基丙二酰CoA合成多聚酮,进而合成环桥链[18]。焦瑞身等通过透射电镜研究发现,添加硝酸盐之后菌体内脂肪堆积减少。定量分析菌体的脂肪酸成分发现,中性脂的总量变低,但其在组成成分上无显著差异;而且在硝酸盐存在下利福霉素SV产量的增加量几乎相当于脂肪含量的减少量[39]。由于利福霉素的环桥链和长链脂肪酸合成共用前体丙二酰 CoA,我们推测硝酸盐抑制了菌体脂肪酸的生物合成,并将用于合成脂肪的乙酰CoA转向利福霉素SV的生物合成。该现象与我们目前的转录组数据一致,例如脂肪酸合成酶系 (FAS) 的基因 (fabD等) 在添加硝酸盐后转录下调了3倍多[28]。
对于利福霉素合成的另外一个重要前体——(2R)-甲基丙二酰CoA,研究认为U32中存在 3条合成途径[40],其关键酶分别为甲基丙二酰 CoA 转羧酶、丙二酰 CoA羧化酶、甲基丙酰 CoA 变位酶 (MCM) 和甲基丙二酰 CoA消旋酶 (MCE)。通过对地中海拟无枝酸菌U32来源的甲基丙二酰CoA转羧酶、甲基丙二酰CoA变位酶和消旋酶进行纯化和酶活测定,证明了它们的酶学性质和其他生物来源的相似[41-42]。在 3条合成途径中,通过分析各个酶活性的时间进程和利福霉素合成时间的相关性,及各个酶的底物亲合力,发现甲基丙二酰CoA变位酶途径是主要负责酶系,该途径催化从琥珀酰CoA生成 (2R)-甲基丙二酰 CoA[40]。尽管丙二酰羧化酶途径不是前体 (2R)-甲基丙二酰 CoA供应的主要途径,但实验发现外源添加10 mmol/L的丙酸盐仍然能促进地中海拟无枝酸菌U32的利福霉素SV产量提高30%[43]。
2.2.2 利福霉素生物合成基因簇
利福霉素合成基因簇上共含 43个基因(AMED_0613-AMED_0655),是U32中最大的基因簇。其中,rifABCDE负责编码PKS模块合成酶类;rifG-rifN负责编码合成AHBA前体酶类;rifP负责编码利福霉素排出蛋白;其他基因则大部分主要负责编码后修饰蛋白和调控蛋白[27]。
通过最近的转录组数据分析,我们发现在有无添加硝酸盐的条件下,利福霉素生物合成基因簇在菌体生长对数期都是处于高水平的转录状态;而到了稳定前期,在添加硝酸盐的条件下该基因簇的转录仍然保持高水平,但无硝酸盐的条件下则转录水平急剧下降。在稳定前期,有、无硝酸盐条件下基因簇内各个基因的转录差异在7–2 960 倍之间[28]。
2.2.3 能量代谢
添加硝酸盐之后,菌体内三羧酸循环中的异柠檬酸脱氢酶、苹果酸脱氢酶的酶活力都显著提高[5]。最近的转录组数据[28]也支持了之前的酶活测定结果,即硝酸盐能够促进can (编码乌头酸酶) 和 icd (编码异柠檬酸脱氢酶) 的转录上调。三羧酸循环中这些酶活力的提高无疑为合成利福霉素提供了更多的能量;同时,由于(2R)-甲基丙二酰CoA主要由三羧酸循环中的琥珀酰CoA生成,三羧酸循环的增强能够促进该前体物质的合成。至于硝酸盐如何调控这些基因的转录,以及在调控转录的同时是否影响酶的修饰,这些都有待于进一步的深入研究。
2.2.4 转录调控
GlnR属于OmpR/PhoB家族,是一个非典型的孤对应答调控蛋白;其负责调控放线菌中与氮代谢相关的绝大多数基因的转录。为了深入了解GlnR的调控机制,多个实验室对其结合保守序列进行了深入研究,提出了GlnR蛋白的保守结合位点模型[44-48]。在U32中,GlnR被发现与硝酸盐效应直接相关:在glnR突变株RK中,添加硝酸盐并不能促进利福霉素产量的提高。目前的实验已经证明,在硝酸盐存在的条件下,GlnR激活了与利福霉素前体供给相关的nas operon[49]和glnA[36,50-51]的转录,同时抑制了ald基因的转录[38],从而促进氮代谢以及体内Gln (谷氨酰胺) 的积累。而在RK中,由于nas和glnA的转录无法被激活,因此菌体内无法生成大量的Gln,从而限制了利福霉素合成过程中氮原子的供给。
作为“硝酸盐效应”乃至整个氮代谢调控中的关键调控蛋白,鉴定GlnR的翻译后修饰及其上游的信号 (信号分子及信号转导通路) 对于我们理解“硝酸盐效应”具有非常重要的意义。我们还对GlnR蛋白N端Receiver结构域的晶体结构进行了解析,发现其缺乏典型的磷酸化口袋,而且保守的 Asp50并不被磷酸化修饰;但Asp50对形成有功能的同源二聚体是不可缺少的,该结构由Arg-Asp-Thr电荷网络形成[52]。
虽然GlnR的Asp50位点不被磷酸化修饰,通过现有的 2D-蛋白凝胶电泳结合 Western blotting技术,我们已经证明GlnR蛋白存在多个未知的翻译后修饰 (数据未发表)。进一步鉴定这些修饰类型及其与 GlnR功能的关系将是我们今后工作的重点之一。
除氮代谢外,硝酸盐还调控了初级代谢中其他与前体供给相关基因的表达,如脂肪酸合成及 (2R)-甲基丙二酰 CoA合成相关基因。此外,目前还不清楚利福霉素生物合成基因簇的转录调控因子及其调控机制。对这些基因调控机制的研究将能够帮助我们更加深入地理解“硝酸盐效应”的分子机制。
3 展望
自20世纪70年代末发现“硝酸盐效应”以来,中国科学院合成生物学实验室 (原微生物次生代谢调控实验室/分子微生物学实验室)在焦瑞身先生、姜卫红研究员、赵国屏院士等的带领下对其进行了系统而深入的研究,并揭示了硝酸盐通过促进利福霉素生物合成前体供给和合成基因簇基因的高表达来最终提高利福霉素的产量。同时,随着氮代谢调控蛋白GlnR的鉴定,我们对硝酸盐调控氮代谢相关基因转录的机理有了初步的认识。然而,目前仍有大量未知的问题有待进一步解决。例如,“硝酸盐效应”中的信号分子及信号转导途径,GlnR蛋白的翻译后修饰类型及机制,利福霉素生物合成基因簇的调控机制以及除氮代谢之外的其他前体代谢途径 (特别是脂肪酸代谢) 的调控机制。近年来,随着各种研究手段的飞速发展,我们可以借助比较基因组分析、RNA-seq、蛋白质组学和代谢流分析等多种方法来进一步研究“硝酸盐效应”,并进而认识地中海拟无枝酸菌乃至整个放线菌属的氮代谢调控机制。由于氮代谢与放线菌的次级代谢密切,对氮代谢的深入理解将能够帮助我们在工业上更好地进行次级代谢产物的生产。同时,放线菌的氮代谢调控与肠细菌及低 GC革兰氏阳性菌 (如枯草芽胞杆菌) 都有显著的差异,对放线菌氮代谢调控的研究将能够丰富人们对微生物氮代谢调控的认识。
致谢:感谢香港中文大学王骏教授对本文的认真修改。
[1] Chiao JS, Xia TH, Mei BG, et al. Rifamycin SV and related ansamycins//Vining LC, Stuttard C,Eds. Genetics and Biochemistry of Antibiotic Production. Boston: Butterworth-Heinemann Press,1996: 477–498.
[2] Wehrli W. Ansamycins: chemistry, biosynthesis and biological activity//Medicinal Chemistry:Topics in Current Chemistry Volume 72. Berlin Heidelberg: Springer, 1977: 21–49.
[3] Lal R, Lal S. Recent trends in rifamycin research.Bioessays, 1994, 16(3): 211–216.
[4] Brogden RN, Fitton A. Rifabutin. A review of its antimicrobial activity, pharmacokinetic properties and therapeutic efficacy. Drugs, 1994, 47(6):983–1009.
[5] Chen YM, Gu WL, Wang W, et al. Studies on the metabolic regulation of biosynthesis of rifamycin by Nocardia mediterranei II. The regulatory effect of nitrate on the metabolic pathway of Nocardia mediterranei. Acta Phytophysiol Sin, 1980, 6(3):291–297 (in Chinese).
陈聿美, 顾薇玲, 王武, 等. 地中海诺卡氏菌(Nocardia mediterranei) 合成力复霉素代谢调节的研究 II. 硝酸盐对地中海诺卡氏菌代谢途径的调节. 植物生理学报, 1980, 6(3): 291–297.
[6] Margalith P, Beretta G. Rifomycin. XI. Taxonomic study on Streptomyces mediterranei nov. sp..Mycopathol Mycol Appl, 1960, 13(4): 321–330.
[7] Thiemann JE, Zucco G, Pelizza G. A proposal for the transfer of Streptomyces mediterranei margalith and beretta 1960 to the genus Nocardia as Nocardia mediterranea (margalith and beretta).Arch Microbiol, 1969, 67(2): 147–155.
[8] Lechavalier MP, Prauser H, Labeda D P, et al. Two new genera of Nocardioform actinomycetes:Amycolata gen. nov. and Amycolatopsis gen. nov..Int J Syst Bacteriol , 1986, 36(1): 29–37.
[9] You JY. Recent advances in semi-synthetic rifamycin antibiotics. Chin J Antibiotic, 1985,10(4): 223–229 (in Chinese).
游金莺. 利福霉素类半合成抗生素的研究概况.抗生素, 1985, 10(4): 223–229.
[10] 李同振. 关于发展利福霉素系列产品的探讨. 医药情报, 1990, (5): 11–13.
[11] 蒋毅. 利福霉素 SV菌种选育与发酵工艺研究[D]. 上海: 华东理工大学, 2005.
[12] 张永昌, 邹美云, 盛静娴, 等. 利福霉素产生菌U-32突变株的选育. 上海: 上海遗传学会 1978年年会, 1978.
[13] Jiao RS, Chen YM, Wu MG, et al. Studies on the metabolic regulation of biosynthesis of rifamycin by Nocardia mediterranei I. The stimulative effect of nitrate on biogynthesis of rifamycin SV by Nocardia mediterranei. Acta Phytophysiol Sin,1979, 5(4): 395–402 (in Chinese).
焦瑞身, 陈聿美, 吴梦淦, 等. 地中海诺卡氏菌(Nocardia mediterranei) 合成力复霉素代谢调节的研究 I. 硝酸盐对地中海诺卡氏菌合成力复霉素 SV的促进作用. 植物生理学报, 1979, 5(4):395–402.
[14] Ni LY, Jiao RS. Effect of potassium nitrate on Nocardia mediterranei U-32. Chin J Antibiotic,1986, 11(3): 211–217 (in Chinese).
倪榴英, 焦瑞身. 硝酸钾对地中海诺卡氏菌U-32的影响. 抗生素, 1986, 11(3): 211–217.
[15] Jin Z, Jiao RS. Stimulative effects of nitrate and magnesium salts on biosynthesis of lincomycin by Streptomyces lincolnensis. Chin Biochem J, 1997,13(6): 709–715 (in Chinese).
金詟, 焦瑞身. 硝酸盐、镁盐对林可霉素生物合成的促进作用. 生物化学杂志, 1997, 13(6):709–715.
[16] Jiang S, Huang WY. Improvement of fermentation conditions for azalomycin B produced by Streptomyes hygroscopicus NND-52-C. Chin J Bioproc Eng, 2004, 2(1): 53–57 (in Chinese).
江曙, 黄为一. 吸水链霉菌 NND-52-C菌株产阿扎霉素B发酵条件的优化. 生物加工过程, 2004,2(1): 53–57.
[17] Prelog V, Oppolzer W. Rifamycins. IV.Ansamycins, a novel class of microbial metabolism products. Helv Chim Acta, 1973, 56: 2279.
[18] White RJ, Martinelli E, Gallo GG, et al. Rifamycin biosynthesis studied with13C enriched precursors and carbon magnetic resonance. Nature, 1973,243(5405): 273–277.
[19] Jiao RS, ChenYM, Zhang XC, et al. Studies of the biochemical complementation of inactive mutants of Nocardia mediterranei and the biosynthetic pathway of rifamycin (I). Screening of inactive mutants capable of biochemical complementation.Acta Phytophysiol Sin, 1979, 5(3): 185–191 (in Chinese).
焦瑞身, 陈聿美, 张雪聪, 等. 地中海诺卡氏菌(Nocardia mediterranei) 突变株的生化互补和力复霉素合成途径的研究 (I) 力复霉素生化互补突变株的筛选. 植物生理学报, 1979, 5(3):185–191.
[20] Chen YM, Zhang XC, Shen MJ, et al. Studies of the biochemical complementation of inactive mutants of Nocardia mediterranei and the biosynthetic pathway of rifamycin II. The biochemical complementation pattern of rifamycin SV. Acta Phytophysiol Sin, 1980, 6(4): 331–335 (in Chinese).
陈聿美, 张雪聪, 沈美娟, 等. 地中海诺卡氏菌(Nocardia mediterranei) 突变株的生化互补和力复霉素生物合成途径的研究 (II) 力复霉素的生化互补图. 植物生理学报, 1980, 6(4): 331–335.
[21] 焦瑞身, 刘慈俊, 金志坤, 等. 力复霉素生物合成中氮原子的参入途径. 中国科学 B辑, 1983,(12): 1097–1104.
[22] Jin ZK, Jiao RS. Further studies on the route of incorporation of nitrogen atom into rifamycin. Acta Microbiol Sin, 1986, 26(3): 265–270 (in Chinese).
金志坤, 焦瑞身. 力复霉素生物合成中15N标记化合物参入的研究. 微生物学报, 1986, 26(3):265–270.
[23] Ghisalba O, Nüesch J. A genetic approach to the biosynthesis of the rifamycin-chromophore in Nocardia mediterranei. IV. Identification of 3-amino-5-hydroxybenzoic acid as a direct precursor of the seven-carbon amino starter-unit. J Antibiot, 1981, 34(1): 64–71.
[24] Guo JT, Frost JW. Kanosamine biosynthesis: a likely source of the aminoshikimate pathway's nitrogen atom. J Am Chem Soc, 2002, 124(36):10642–10643.
[25] Huang JQ, Jiang WH, Zhao GP, et al. Cloning,sequencing and expression of AHBAS gene from Amycolatopsis mediterranei U-32. Chin J Biotech,1999, 15(2): 160–165 (in Chinese).
黄健强, 姜卫红, 赵国屏, 等. 地中海拟无枝菌酸菌 U32的 3-氨基-5-羟基苯甲酸合成酶(AHBAS) 基因的克隆与序列分析. 生物工程学报, 1999, 15(2): 160–165.
[26] Floss HG, Yu TW. Rifamycin-mode of action,resistance, and biosynthesis. Chem Rev, 2005,105(2): 621–632.
[27] Zhao W, Zhong Y, Yuan H, et al. Complete genome sequence of the rifamycin SV-producing Amycolatopsis mediterranei U32 revealed its genetic characteristics in phylogeny and metabolism. Cell Res, 2010, 20(10): 1096–1108.
[28] Shao ZH, Ren SX, Liu XQ, et al. A preliminary study of the mechanism of nitrate-stimulated remarkable increase of rifamycin production in Amycolatopsis mediterranei U32 by RNA-seq.Microbial Cell Factories, 2015, 14: 75.
[29] 李果龙. 地中海拟无枝菌酸菌 U-32硝酸还原酶的纯化及基因克隆[D]. 上海: 中国科学院上海植物生理生态研究所, 1989.
[30] Li GL, Jiao RS. Nitrate assimilation of Amycolatopsis mediterranei U-32 and some properties of its nitrate reductase. Acta Microbiol Sin, 1995, 35(2): 141–148 (in Chinese).
李果龙, 焦瑞身. 地中海拟无枝菌酸菌U-32对硝酸盐的同化及其硝酸还原酶特性. 微生物学报,1995, 35(2): 141–148.
[31] Li GL, Yang YL, Jiao RS. Localization,purification and characterization of nitrate reductase from Amycolatopsis mediterranei U-32.Chin J Biochem Mol Biol, 1998, 14(6): 710–714(in Chinese).
李果龙, 杨蕴刘,焦瑞身. 地中海拟无枝菌酸菌(Amycolatopsis mediterranei) U-32 硝酸还原酶的定位、纯化及性质. 中国生物化学与分子生物学报, 1998, 14(6): 710–714.
[32] Shao ZH, Gao J, Ding XM, et al. Identification and functional analysis of a nitrate assimilation operon nasACKBDEF from Amycolatopsis mediterranei U32. Arch Microbiol, 2010, 193(7): 463–477.
[33] Diao R, Jiao RS. Purification and properties of L-Alanine dehydrogenase from Nocardia mediterranei. Acta Microbiol Sin, 1991, 31(3):206–212 (in Chinese).
刁蓉, 焦瑞身. 地中海诺卡氏菌丙氨酸脱氢酶的纯化和性质. 微生物学报, 1991, 31(3): 206–212.[34] 梅百根. 地中海诺卡氏菌 U32氨同化途径(GS/GOGAT) 的调节研究[D]. 上海: 中国科学院上海植物生理生态研究所, 1983.
[35] Ni LY, Jiao RS. Effect of nitrogen compounds on the ammonia assimilatory enzymes in Nocardia mediterranei U-32. Chin J Antibiotic, 1984, 9(4):301–306 (in Chinese).
倪榴英, 焦瑞身. 几种氮化合物对地中海诺卡氏菌 U-32氨同化酶的影响. 抗生素, 1984, 9(4):301–306.
[36] Peng WT, Wang J, Wu T, et al. Bacterial type I glutamine synthetase of the rifamycin SV producing actinomycete, Amycolatopsis mediterranei U32, is the only enzyme responsible for glutamine synthesis under physiological conditions. Acta Biochim Biophys Sin (Shanghai),2006, 38(12): 821–830.
[37] 吴婷. I利福霉素SV生产菌株U-32谷氨酰胺合成酶的转录调控; II抗乙型肝炎病毒药物的筛选[D]. 上海: 上海医科大学, 2000.
[38] Wang Y, Li C, Duan N, et al. GlnR negatively regulates the transcription of the alanine dehydrogenase encoding gene ald in Amycolatopsis mediterranei U32 under nitrogen limited conditions via specific binding to its major transcription initiation site. PLoS One, 2014, 9(8): e104811.
[39] Gu WL, Lu XY, Geng YQ, et al. The regulatory effect of nitrate on the regulationship betreen the production of rifamycin SV and the biosynthesis of cellular lipid components. Acta Microbiol Sin,1983, 23(4): 313–318 (in Chinese).
顾薇玲, 陆小燕, 耿运琪, 等. 硝酸盐对力复霉素 SV和脂肪合成的调节作用. 微生物学报,1983, 23(4): 313–318.
[40] Zhang WW, Jiao RS. The regulation of methylmalonyl-CoA formation pathways in rifamycin SV-producing Amycolatopsis mediterranei U32. Acta Microbiol Sin, 1996, 36(4):276–283 (in Chinese).
张蔚文, 焦瑞身. 力复霉素前体甲基丙二酰 CoA合成途径的研究. 微生物学报, 1996, 36(4):276–283.
[41] Zhang WW, Jiao RS. Purification and characteristics of methylmalonyl-CoA transcarboxylase from rifamycin SV sythesizing Amycolatopsis mediterranei U32. Chin Biochem J,1996, 12(2):176–181 (in Chinese).
张蔚文, 焦瑞身. 地中海拟无枝菌酸菌甲基丙二酰 CoA 转羧基酶的纯化及酶学性质. 生物化学杂志, 1996, 12(2):176–181.
[42] Zhang WW, Jiao RS. Purification and characterization of methylmalonyl-CoA mutase and methylmalonyl-CoA racemase from a rifamycin SV-producing Amycolatopsis mediterranei U32.Acta Microbiol Sin, 1996, 36(3): 199–207 (in Chinese).
张蔚文, 焦瑞身. 地中海拟无枝酸杆菌甲基丙二酰 CoA变位酶和消旋酶的纯化及性质. 微生物学报, 1996, 36(3): 199–207.
[43] Zhang WW, Jiao RS. Regulatory roles of propionate in the production of rifamycin SV by Amycolatopsis mediterranei U32. Chin J Antibiotic, 1995, 20(4): 268–272 (in Chinese).
张蔚文, 焦瑞身. 丙酸盐对利复霉素生物合成的调控. 中国抗生素杂志, 1995, 20(4): 268–272.
[44] Tiffert Y, Supra P, Wurm R, et al. The Streptomyces coelicolor GlnR regulon:identification of new GlnR targets and evidence for a central role of GlnR in nitrogen metabolism in actinomycetes. Mol Microbiol, 2008, 67(4):861–880.
[45] Sola-Landa A, Rodríguez-García A, Amin R, et al.Competition between the GlnR and PhoP regulators for the glnA and amtB promoters in Streptomyces coelicolor. Nucleic Acids Res, 2013, 41(3):1767–1782.
[46] Pullan ST, Chandra G, Bibb MJ, et al.Genome-wide analysis of the role of GlnR in Streptomyces venezuelae provides new insights into global nitrogen regulation in actinomycetes. BMC Genomics, 2011, 12: 175.
[47] Jenkins VA, Barton GR, Robertson BD, et al.Genome wide analysis of the complete GlnR nitrogen-response regulon in Mycobacterium smegmatis. BMC Genomics, 2013, 14: 301.
[48] Wang J, Wang Y, Zhao GP. Precise characterization of GlnR Box in actinomycetes.Biochem Biophys Res Commun, 2015, 458(3):605–607.
[49] Wang Y, Wang JZ, Shao ZH, et al. Three of four GlnR binding sites are essential for GlnR-mediated activation of transcription of the Amycolatopsis mediterranei nas operon. J Bacteriol, 2013,195(11): 2595–2602.
[50] Yu H, Yao YF, Liu Y, et al. A complex role of Amycolatopsis mediterranei GlnR in nitrogen metabolism and related antibiotics production.Arch Microbiol, 2007, 188(1): 89–96.
[51] Yu H, Peng WT, Liu Y, et al. Identification and characterization of glnA promoter and its corresponding trans-regulatory protein GlnR in the rifamycin SV producing actinomycete,Amycolatopsis mediterranei U32. Acta Biochim Biophys Sin (Shanghai), 2006, 38(12): 831–843.
[52] Lin W, Wang Y, Han XB, et al. Atypical OmpR/PhoB subfamily response regulator GlnR of actinomycetes functions as a homodimer, stabilized by the unphosphorylated conserved Asp-focused charge interactions. J Biol Chem, 2014, 289(22):15413–15425.

