基于结构域的芥子油苷合成相关蛋白质相互作用模型构建和预测1)
孙晓芳 刘亚秋 初砚硕
(东北林业大学,哈尔滨,150040)
责任编辑:王广建。
蛋白质在细胞的各类生物过程中发挥着重要作用,且功能多样广泛(促进化学反应、进行信息传递等)。蛋白质通常是以相互作用的形式完成各种生物学功能。了解蛋白质相互作用(PPIs),可更好的了解蛋白质的功能及参与生物学的过程。
近年来,芥子油苷被证实是重要的抗癌药物前体,能被模式植物拟南芥合成。针对参与芥子油苷生物合成过程的蛋白质相互作用进行预测,构建蛋白质相互作用网络,从蛋白质一级序列抽取了蛋白质结构域信息并建立结构域特征向量,使用一对对称的特征向量表示一对蛋白质关系,应用支撑向量机方法构建蛋白质相互作用预测器。
1 蛋白质相互作用预测研究概况
目前,使用生物实验来判断蛋白质之间发生相互作用的方法有酵母双杂交、免疫共沉淀等,这类生物实验方法费时费力,使用计算方法预测蛋白质相互作用,能辅助生物学实验缩减待测样本数量,提高验证效率。
在众多的基于蛋白质结构的蛋白质相互作用预测方法中,一类方法是从蛋白质的一级结构,即氨基酸序列中找到决定蛋白质相互作用的子序列;另一类方法是从蛋白质的一级结构中提取蛋白质的物理化学性质或者蛋白质的高级结构,通过这类信息进行预测蛋白质相互作用。这些方法特征向量的维度过高、计算复杂,且其特征向量中含有冗余特征。目前对特征向量降维方法有很多种,例如:将20 个氨基酸根据其极性以及配基的分子量、范德华力等压缩为几类氨基酸,或者从样本中选取几类主要特征、使用氨基酸的2 元组(3 元组)代替单位为1 个氨基酸的特征表示形式。
蛋白质的结构域被认为是构成蛋白质的框架结构。认为蛋白质结构域是长期进化而来的构成蛋白质结构和功能的基本单元,蛋白质结构域代表了蛋白质的结构特征。基于蛋白质结构域的蛋白质相互作用预测方法,认为蛋白质相互作用是蛋白质中的结构域和结构域相互作用而导致的。
Chen 等[1-2]使用结构域相互作用数据库中的结构域信息加上文献中的数据作为蛋白质相互作用预测的源数据,这些数据含有3 713 个蛋白质、9 843个相互作用、4 239 个结构域,再加上该对蛋白是否真正发生相互作用,共组成了4240 维的蛋白质相互作用特征向量。使用决策森林作为蛋白质相互作用的学习机,进行蛋白质相互作用预测,然后用两个蛋白质特征向量拼接的方法构建特征向量,拼接后的特征向量作为该组蛋白质对是否相互作用的特征。Jefferson 等[3]提出使用结构域的相互作用信息作为预测蛋白质相互作用,实验结果表明使用结构域相互作用(DDIs)信息可以提高蛋白质相互作用预测的准确性。
本文使用数据库AtPIN[4]中的拟南芥蛋白质相互作用数据作为源数据,用Pfam25.0[5]中的结构域隐马尔科夫模型,从蛋白质一级序列中识别结构域,DOMINE 数据库中的结构域相互作用信息作为额外补充特征。根据以上数据,构建对称的蛋白质相互作用特征向量。使用该特征向量作为训练样本,训练支撑向量机学习机;在参数选择中,使用5 折交叉验证的网格寻优方法,优化高斯径向基核函数的降噪参数(c)和核宽参数(γ)。然后,使用高斯径向支撑向量机作为蛋白质相互作用预测器,使用数据库中的蛋白质相互作用对进行验证,使用预测器预测蛋白质的相互作用。
2 蛋白质相互作用预测器的构建
2.1 蛋白质相互作用特征表示
蛋白质相互作用预测的问题可以形式化为两类问题:每一个蛋白质对作为一个样本,属于相互作用类或者不相互作用类。蛋白质A 和蛋白质B 组成的蛋白质对属于相互作用(或者不相互作用)类,可表述为A 与B 有相互作用(没有相互作用),或者B与A 有相互作用(或没有相互作用)。蛋白质A 和蛋白质B 组成的蛋白质对有无相互作用关系,表示为,那么,向量可以表示为同时使用这两个对称的向量来表示蛋白质对AB 之间的关系(有相互作用或无相互作用)。
源数据集合中共含有1913 个不重复Pfam 结构域,每一个结构域使用数字0 到1912 来一一对应标记。这样每一个蛋白质使用一个1913 维数字向量表示,其每一维对应该位置序号的结构域数量。例如:如果一个蛋白质含有一个标记为5 的结构域,那么特征向量中对应的第5 个数字为1。Pfam25.0 可将蛋白质结构域分为5 个类型(Domain、Motif、Repeat、Family、PfamB)。根据蛋白质结构域的类型,统计一个蛋白质对应的所有由Pfam25.0 发现的各自类型结构域的数目。在原特征向量末尾额外连接5 个特征(D、M、R、F、P)。为表示一对蛋白质的关系,将两个分别表示蛋白质的结构域的特征向量拼接成一个向量。即,蛋白质A 的特征向量表示为VA=[fA0,fA1,fA2,…,fAn-1,DA,FA,RA,MA,PA],其中,n=1 913,fA0表示蛋白质A 中标号为0 的结构域的数量,DA表示蛋白质A 中Domain 类型的结构域的数量,FA表示蛋白质A 中Family 类型结构域的数量,RA表示蛋白质A 中Repeat 的数量,MA表示蛋白质A 中Motif 的数量,PA表示蛋白质A 中PfamB 的数量。蛋白质A 和B 的相互作用关系可以表示为[VA,VB]或[VB,VA]。在结构域相互作用数据库DOMINE中,结构域相互作用按照可信度分为高可信、中可信、低可信预测[6]。在特征向量[VA,VB]和[VB,VA]末尾拼接额外三维向量,分别对应该三类可信度结构域相互作用的数量。最终得到蛋白质A 和B 的相互作用关系的特征向量[VA,VB,HCPs,MCPs,LCPs]和[VB,VA,HCPs,MCPs,LCPs]。进一步对所有特征向量进行归一化,使每一维度数值范围在[0,1]。如式(1)所示,特征矩阵S'm,n中的每一行对应m 个样本中相应的特征向量,特征矩阵归一化方法如式(2)所示,
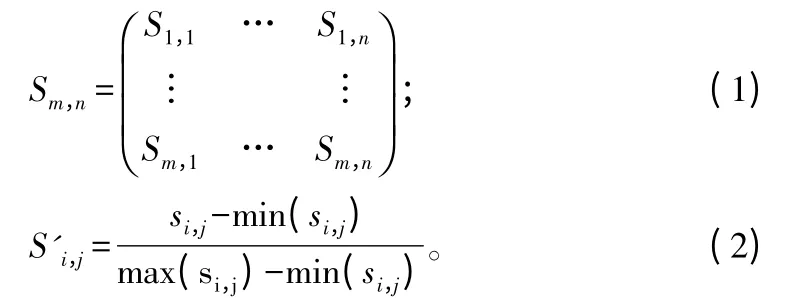
2.2 数据
结构域相互作用数据库DOMINE 搜集了已知和预测的蛋白质结构域相互作用。DOMINE 含有26 219 个结构域相互作用,涉及5 410 个不重复结构域,包含6 634 个直接从PDB 预测而来的DDIs,以及21 620 个由13 种预测算法预测而来的DDIs。在所有DDIs 中,共有2 989 个高可信DDIs、2 537 个中可信DDIs、2 537 个低可信DDIs。
拟南芥蛋白质相互作用数据库AtPIN 收集了拟南芥蛋白质相互作用、本体、亚细胞定位等信息,是一个高可信的拟南芥蛋白质相互作用数据库。在AtPIN 中,所有的蛋白质相互作用被分为2 类。
使用AtPIN 中,至少含有一个结构域的蛋白质对作为正样本。经过滤,我们得到6320 个经过实验验证的蛋白质相互作用对,表示为POS,POS 包含有2695 个蛋白质。使用Pfam25.0 从氨基酸序列中识别结构域。蛋白质对应的氨基酸序列由数据库BiowebDB(http://biowebdb.org/)中下载,该数据库收集了拟南芥中的统一蛋白质资源(UniProt)。本文中使用的负样本由随机生成,生成负样本的方法是在正样本所涉及的蛋白质中随机选取一对不在At-PIN 数据库中的蛋白质对作为一例负样本。本文生成了与正样本同样大小的负样本,共含有2695 对蛋白质。
2.3 参数优化
使用5 折交叉验证的网格搜索高斯径向基核(k(u,v)=exp(-γ*‖u-v‖2))支撑向量机中的降噪参数和核宽参数。参数(c)的网格范围设置为logc2∈[-5,-3,-1,…,13],γ 设置为logy2∈[-15,-13,-11,…,3]。
由图1所示,最佳精确度logc2=1 和logy2=-1,该坐标的5 折交叉验证的高斯径向基核支撑向量机的精确度为85.4549%,最优参数c=2,γ=0.5。
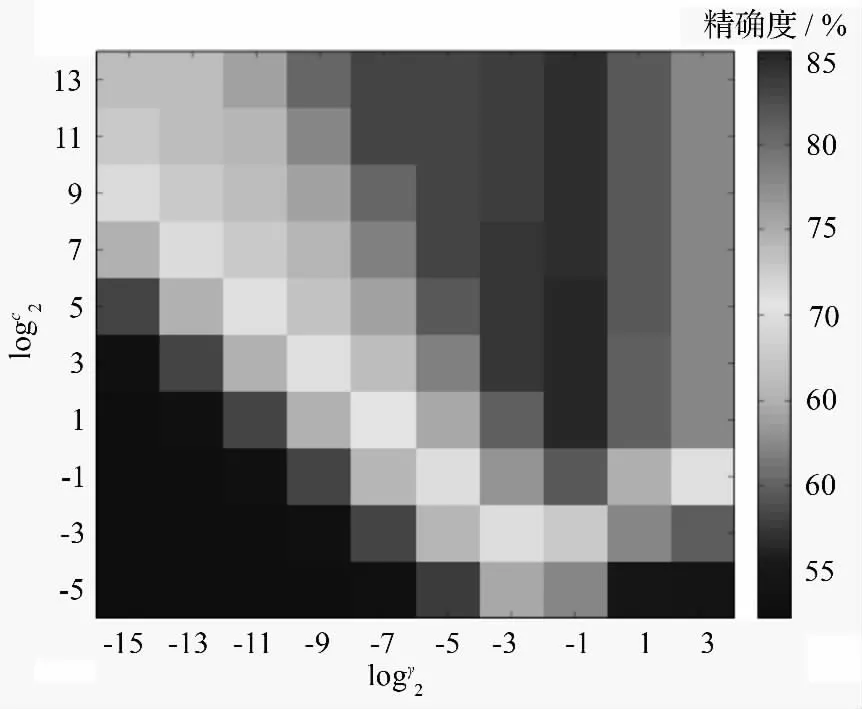
图1 5 折交叉验证网格寻优准确度热力图
图2是根据最优参数c=2,y=0.5 的5 折交叉验证ROC 曲线和最优参数训练,得到支撑向量机的验证ROC 曲线(模型预测)。这两条ROC 曲线的下面积(AUC)分别用AUCCV、AUCMFP。一般地,由所有可用的正样本POS 训练而得的RBF-SVM 的分类性能,优于部分正样本训练而得的RBF-SVM。所以,最终由全部正样本训练得到RBF-SVM 的真实分类性能在这两个下面积之间,即:0.905 72(AUCCV)到0.995 53(AUCMFP)。
3 基于结构域(RBF-SVM)的拟南芥芥子油苷合成相关蛋白质相互作用
芥子油苷是拟南芥中的次代谢产物,富含氮、硫。芥子油苷是模式植物拟南芥与环境发生相互作用的媒介,是植物学研究的热点[7-8]。另外,已证实,芥子油苷与拟南芥中的芥子降解酶发生相互作用时,生成异硫代氰酸盐,该产物具有抗癌活性[9]。
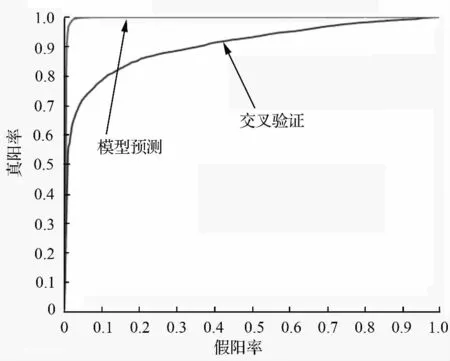
图2 5 折交叉验证和PPI 预测模型的ROC 曲线
在模式植物拟南芥中,芥子油苷是一种次代谢产物,由氨基酸经过乙醛肟、氧化腈、酸式硝基化合物、脱硫芥子油苷等中间产物生成。其中,多种酶在此过程中起催化作用(细胞色素P450(CYS450)、P79(CYS79),谷胱甘肽转移酶(G-ST),S-糖基转移酶(SGT),磺基转移酶(ST)等)。应用PPI 预测器构建拟南芥中芥子油苷合成途径相关的PPI 网络。
AtPIN 中的5697 个预测而得的蛋白质相互作用,所涉及的蛋白质结构域都存在于正样本POS中;AtPIN 中的90043 个预测蛋白质相互作用不是高可信,且其数量巨大,若以该数据构建PPI 网络将给研究拟南芥带来很大的噪声。将AtPIN 中的低可信的预测蛋白质相互作用标记出来,会有助于芥子油苷合成相关的蛋白质网络相互作用的研究。
文献[10]中列出了拟南芥中与芥子油苷合成相关的46 个基因。基于AtPIN 中的蛋白质相互作用,这46 个基因对应的蛋白质作为种子蛋白,来寻找新的与这46 个蛋白质相互作用的蛋白质。在At-PIN 中有4 个实验证实的蛋白质相互作用,237 个预测相互作用。
图3为该芥子油苷相关PPI 网络,基于表1中列出的46 个种子蛋白质而构建的,该PPI 网络的深度为1。

表1 芥子油苷合成相关的46 个基因

续(表1)
4 结束语
蛋白质结构域是不同蛋白质家族中结构保守区域。蛋白质相互作用是通过分子间的作用产生,而这种相互作用存在在蛋白质的特定的结构域中,因此,在结构域层面对蛋白质相互作用的理解,能够加深对蛋白质功能的了解。使用结构域信息,预测芥子油苷相关的蛋白质相互作用并构建PPI 网络。用对称训练方法解决蛋白质相互作用的对称问题,即使用一对对称的特征向量表示一对蛋白质的相互作用关系。特征向量基于从氨基酸序列中抽取的结构域和结构域相互作用信息构建。高斯径向基核的降噪参数(c)和核宽参数(γ)由5 折交叉验证的网格寻优而得。使用ROC 曲线下面积来衡量RBF-SVM的预测性能。最优RBF 为k(u,v)= exp(-0.5*‖u-v‖2),最佳降噪参数c=2。使用RBF-SVM 验证了AtPIN 数据库中的预测类蛋白质的可信度,并发 现 了 蛋 白 质 AGI,标 号 为 AT4G14800、AT5G54810、AT5G05730、AT4G18040、AT1G04510、AT5G05260 是相互作用的蛋白质对。目前,拟南芥中实验证实的蛋白质相互作用的数量远远达不到研究所需,同时,在预测类的蛋白质相互作用数据中还含有未知数量的噪声,因此,蛋白质相互作用的预测仍然是一大难题。
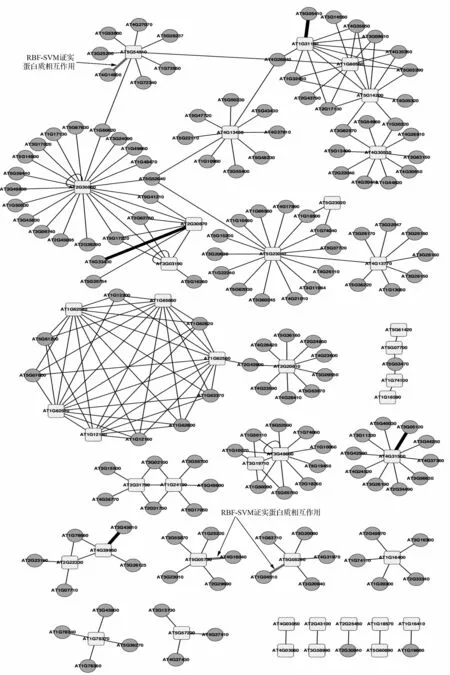
图3 AtPIN 中芥子油苷合成相关的PPI 网络
[1] Xuewen Chen,Mei Liu.Prediction of protein-protein interactions using random decision forest framework[J].Bioinformatics,2005,21(24):4394-4400.
[2] Xuewen Chen,Mei Liu.Domain-based predictive models for protein-protein interaction prediction[J].Eurasip Journal on Advances in Signal Process-ing,2006,2006:1-8.
[3] E R Jefferson,T P Walsh,G J Barton.Biological units and their effect upon the properties and prediction of Protein-Protein interactions[J].Journal of Molecular Biology,2006,364(5):1118-1129.
[4] Brandão M M,Dantas L L,Silva-Filho M C.Atpin:Arabidopsis thaliana protein interaction net-work[J].BMC Bioinformatics,2009,10(1):454-458.
[5] Robert D F,John T,Jaina M,et al.The pfam protein families database[J].Nucleic Acids Research,2008,36:281-288.
[6] S Yellaboina,A Tasneem,D V Zaykin,et al.Domine:a comprehensive collection of known and predicted domain-domain interactions[J].Nucleic Acids Research,2011,39(1):730-735.
[7] Michael Reichelt,Paul D Brown,Bernd Schneider,et al.Benzoic acid glucosinolate esters and other glucosinolates from arabidopsis thaliana[J].Phytochemistry,2002,59(6):663-672.
[8] S J Tonsor,C Alonso-Blanco,M Koornneef.Gene function beyond the single trait:natural variation,gene effects,and evolutionary ecology in arabidopsis thaliana[J].Plant,Cell & Environment,2005,28(1):2-20.
[9] C Douglas Grubb,Steffen Abel.Glucosinolate metabolism and its control[J].Trends in Plant Science,2006,11(2):89-100.
[10] Yazhou Chen,Xiufeng Yan,Sixue Chen.Bioinformatic analysis of molecular network of glucosinolate biosynthesis[J].Computational Biology and Chemistry,2011,35(1):10-18.

