基于系统-合成生物学的人工细胞网络设计
汪建峰 王 勇(中国科学院上海生命科学研究院植物生理生态研究所合成生物学重点实验室,上海 200032)
基于系统-合成生物学的人工细胞网络设计
汪建峰 王 勇
(中国科学院上海生命科学研究院植物生理生态研究所合成生物学重点实验室,上海 200032)
汪建峰,博士,助理研究员,主要研究方向为复杂天然产物异源合成系统的设计。
E-mail:jfwang@sibs.ac.cn
在过去的十多年里,研究人员利用合成生物学方法在模式微生物底盘中设计和重建了一系列重要天然化合物的异源合成模块,大大拓展了微生物发酵在珍稀药物、食品添加剂、生物能源和精细化学品生产领域的应用价值。为了实现异源产物的高产量和高得率,必须综合运用系统生物学和合成生物学方法,对异源模块进行优化设计,强化底盘细胞的前体和能量合成,并改善模块的适配性,最终实现异源化合物的高产。文章综述了近年来在人工细胞网络设计上的进展。

王勇,博士,中国科学院“百人计划”研究员,主要研究方向为天然产物的合成生物学。
E-mail:yongwang@sibs.ac.cn
在过去的十多年里,研究人员利用合成生物学方法在大肠杆菌(Escherichia coli)、酿酒酵母(Saccharomyces cerevisiae)等常见的模式微生物底盘(Chassis)中设计和重建了一系列重要萜类 、苯丙烷素类 、聚酮类 等天然化合物的异源合成模块,成功赋予了原有底盘细胞生产重要外源化合物的能力,大大拓展了微生物发酵在珍稀药物、食品添加剂、生物能源和精细化学品生产领域的应用价值。为能够快速响应环境变化并以最经济的状态生存,由自然进化而来的初始底盘细胞代谢网络往往存在着大量支路代谢、可逆代谢、补偿代谢等途径,而且在基因转录、蛋白翻译、酶活水平等各个层次受到极其复杂、严谨的调控。因此,初始底盘细胞内源代谢仅能向异源代谢模块提供极少量的前体和能量来进行异源产物的生产。为了实现异源产物的高产量和高得率,除了需要对异源模块进行设计优化外 ,另一个迫切的研究内容是如何利用各种工程策略强化底盘细胞的前体和能量合成,并改善内源模块和异源模块的适配性(如空间尺度的接近、时间尺度的同步等)。经典诱变育种方法随机性强、定向筛选困难、过程周期漫长。传统代谢工程的方法仅对局部代谢网络中单个或者少数靶点进行改造,难以达到预期目标。大肠杆菌与酿酒酵母等底盘细胞具有遗传背景清晰、遗传操作手段成熟等优点。针对这些模式微生物,研究人员已经建立了各种成熟的系统生物学研究方法与工具,如组学分析 、全基因组规模代谢模型(GSMM)、in silico的代谢网络分析 等,同时也开发了一整套合成生物学研究工具和技术,如精细可控的表达调控元件、基因回路DNA装配和染色体工程技术 等。因此,结合运用这些系统生物学和合成生物学方法,可以快速鉴定全基因组规模潜在遗传靶点,并对大量靶点实施精准、快速的改造与集成,最终大大提高菌株改造的效率和周期,实现异源化合物的高产 。
1 全基因组规模靶点预测与诊断
1.1 全基因组规模in silico细胞网络分析的靶点预测
随着大量微生物全基因组测序和注释工作的完成,研究人员对微生物复杂的全基因组规模代谢网络有了更全面深入的认识。基于基因-蛋白质-生化反应的相互关系,将全基因组代谢网络抽象为一系列按一定计量关系运行的生化反应,然后可以利用计量系数矩阵模型建立基因组规模代谢模型(GSMM)。以GSMM为基础,运用一系列基于预先假定的约束条件(如最大菌体生长、最大产物合成)进行全细胞代谢网络流量分析,如通量平衡分析(FBA)、最小代谢调整分析(MOMA)等,可以发现大量能改善目标产物合成的潜在途径或基因,特别是一些与产物合成相距较远、表观上无直接关联的靶点。相比于传统方法,这些信息可以更理性地指导发现更多有效的基因靶点,减少实验的盲目性。自从Edwards等 2000年发表了第一个GSMM以来,经过十年的发展,至今已建立了超过100个物种的基因组尺度代谢模型(表1)。随着对细胞代谢网络认识的加深,更多的信息被整合到了模型中,大肠杆菌和酿酒酵母等模式生物的代谢网络模型被不断更新,为更全面、彻底地预测细胞表型和遗传靶点奠定基础。
Ajikumar P K, Xiao W H,Toy K E, et al. Isoprenoid pathway optimization for Taxol precursor overproduction in Escherichia coli. Science, 2010, 330:70-74.
Wang S, Zhang S, Xiao S, et al. Metabolic engineering of Escherichia coli for the biosynthesis of various phenylpropanoid derivatives. Metab Eng, 2015. 29:153-159.
Zhang H, Wang Y, Wu J, et al. Complete biosynthesis of erythromycin A and designed analogs using E. coli as a heterologous host. Chem Biol, 2010. 17:1232-1240.
Koffas M,G Stephanopoulos.Strain improvement by metabolic engineering: lysine production as a case study for systems biology. Curr Opin Biotechnol, 2005. 16:361-366.
Vemuri G N,Aristidou A A.Metabolic engineering in the -omics era: elucidating and modulating regulatory networks. Microbiol Mol Biol Rev, 2005. 69:197-216.
Xu C, Liu L, Zhang Z, et al. Genome-scale metabolic model in guiding metabolic engineering of microbial improvement. Appl Microbiol Biotechnol, 2013. 97:519-539.
Curran K A, Crook N C, Alper H S. Using flux balance analysis to guide microbial metabolic engineering. Methods Mol Biol, 2012. 834:197-216.
Wang H H, Isaccs F J,Carr P A, et al. Programming cells by multiplex genome engineering and accelerated evolution. Nature, 2009. 460(7257):894-898.
Liu Y, Shin H D, Li J, et al. Toward metabolic engineering in the context of system biology and synthetic biology: advances and prospects. Appl Microbiol Biotechnol, 2015. 99:1109-1118.
目前全基因组规模的in silico细胞网络分析已经被广泛应用到了传统工业生产菌株和一些天然化合物异源合成工程菌株的改造中。研究人员通过对全基因组模型的进一步修正和约束算法的优化,不断强化预测的全面性和准确性。Alper等 利用MOMA方法,预测并鉴定了大肠杆菌中能提高番茄红素合成的基因敲除靶点。随后,Choi等 开发了限制目标流量的流量扫描分析方法(FSEOF),不仅预测了大肠杆菌中能提高番茄红素合成的敲除靶点,同时也获得了一些潜在的强化靶点。笔者实验室为了进一步提高in silico预测效率,以经典的FBA和MOMA算法为基础,开发了流量分布比较分析(FDCA)和LMOMA-based两种新的算法 。首先,FDCA算法是以基因组范围的代谢模型为基础,分别采用最大菌体生长和最大产物合成作为约束条件,利用FBA方法计算各自约束条件下代谢通量分布,通过进一步比较这两种状态下的流量分布情况,找出所有显著上调或下调的节点(反应),这些节点即是理论上潜在改造靶点。因此,FDCA实质是in silico代谢流量组的比较分析,它可以从全基因组规模快速找到影响某个表型的所有潜在敲除、强化或弱化靶点。其次,LMOMA-Based算法以LMOMA(线性MOMA)方法为基础发展的,意在实现对全基因组范围模型中的所有靶点进行深入的叠加敲除分析。该算法的每一轮敲除都是对全基因组范围所有靶点的一次全扫描敲除分析,在找到该轮敲除中
Edwards J S, Palsson B O.The Escherichia coli MG1655 in silico metabolic genotype: its definition, characteristics, and capabilities. Proc Natl Acad Sci U S A, 2000, 97:5528-5533.
Weaver D S, Keseler I M, Mackie A, et al. A genome-scale metabolic flux model of Escherichia coli K-12 derived from the EcoCyc database. BMC Syst Biol, 2014. 8:79.
Orth J D,Conard T M, Na J, et al. A comprehensive genome-scale reconstruction of Escherichia coli metabolism-2011, Mol Syst Biol, 2011, 7:535.
Feist A M, Henry C S,Reed J L,et al. A genome-scale metabolic reconstruction for Escherichia coli K-12 MG1655 that accounts for 1260 ORFs and thermodynamic information. Mol Syst Biol, 2007.3:121.
Reed J L,Vo T D,Schilling C H, et al. An expanded genome-scale model of Escherichia coli K-12 (iJR904 GSM/GPR). Genome Biol, 2003. 4(9):R54.
Herrgard M J,Swainston N, Dobson P, et al. A consensus yeast metabolic network reconstruction obtained from a community approach to systems biology. Nat Biotechnol, 2008. 26:1155-1160.
Nookaew I, Jewett M C,Meechai A,et al.The genome-scale metabolic model iIN800 of Saccharomyces cerevisiae and its validation: a scaffold to query lipid metabolism. BMC Syst Biol, 2008. 2:71.
Blank L M, Kuepfer L, Sauer U. Large-scale 13C-flux analysis reveals mechanistic principles of metabolic network robustness to null mutations in yeast. Genome Biol, 2005. 6:R49.影响最显著靶点后,进入下一轮的敲除分析。下一轮的敲除分析采用的模型即是敲除了上轮影响最显著靶点后的新模型。通过这样的叠加敲除,最终可以得到理论上最佳表型。随后笔者研究组及相关合作者将这两种算法应用到了大肠杆菌及酿酒酵母的产萜类或聚酮类化合物的人工细胞网络优化中,并在这些不同的系统鉴定了一系列能提高目标产物产率的新靶点。随着各种微生物全基因组规模细胞网络的完善,全基因组规模的in silico细胞网络分析将在遗传靶点预测与鉴定方面发挥越来越大的作用。
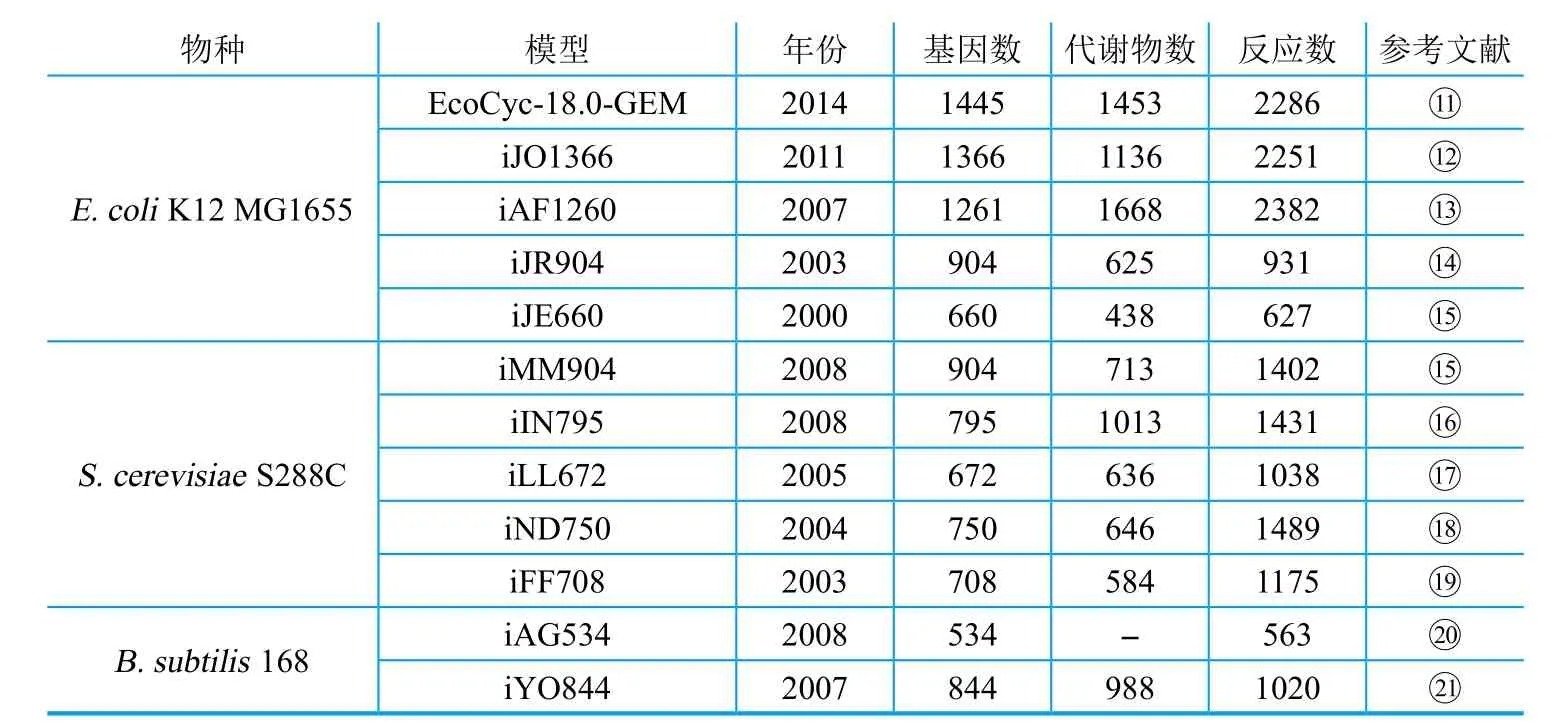
表1 几种重要底盘微生物的全基因组代谢网络模型
1.2 基于组学数据比较分析的细胞网络诊断
基于GSMM计量模型的in silico代谢网络方法可以快速鉴定理论上的敲除、弱化或强化靶点,但目前这种计量模型分析仅仅反应某种理想状态下静态的代谢流量分布,无法体现细胞内代谢状态(如RNA丰度、酶浓度、代谢物浓度等)的动态变化和复杂调控,因而无法指导开展更精细的遗传改造,如基因表达剂量优化、基因表达时间上的开关等,同时也无法反映实验条件下细胞受到的各种环境胁迫作用,如菌株的耐受性等。借助全基因组范围或某个核心代谢模块的转录组、蛋白质组、代谢组、代谢流量组等分析,可有效掌握细胞内酶、底物、中间代谢物及产物浓度的动态变化。这样的系统诊断分析对如何进一步设计高产菌株和选取最有效的改造策略具有重要意义。
细胞内RNA水平直接影响转录和翻译两个重要的基因表达过程。通过基于微阵列芯片和RNA-seq的转录组分析,可以快速鉴定特定条件下大量基因转录水平的变化,从而发现未知的基因表达调控规律以及许多与耐受性相关靶点。Foo 等 利用比较转录组方法分析了大肠杆菌宿主在外源添加高浓度异戊烯醇环境下基因转录水平的变化,发现并验证了与氧化胁迫响应、通用胁迫响应、热激响应和产物转运相关的8个靶点的强化表达能够提高宿主对异戊烯醇耐受性。进一步对高产异戊烯醇工程菌株测试,发现有6个靶点过表达能够强化异戊烯醇的合成,其中蛋氨酸合成调节子MetR能使异戊烯醇产量提高55%。Hess等 借助转录组分析对各种不同环境条件下具有不同异戊二烯产量的枯草芽孢杆菌的基因表达谱进行研究。他们将其中的213个受调控基因的表达与异戊二烯的产量关联起来,并构建了相关预测模型。这些相关的分析可以有效地指导菌株的后续改造及培养条件优化。
Duarte N C, Herrgard M J, Palsson B O. Reconstruction and validation of Saccharomyces cerevisiae iND750, a fully compartmentalized genome-scale metabolic model. Genome Res, 2004. 14:1298-309.
Forster J, Famili I, Fu Patrick, et al. Genomescale reconstruction of the Saccharomyces cerevisiae metabolic network. Genome Res, 2003, 13:244-253.
Goelzer A, Bekkal B F,Martin-Verstraete I, et al.Reconstruction and analysis of the genetic and metabolic regulatory networks of the central metabolism of Bacillus subtilis. BMC Syst Biol, 2008. 2:20.
Oh Y K,Palsson B O,Park S M, et al. Genome-scale reconstruction of metabolic network in Bacillus subtilis based on high-throughput phenotyping and gene essentiality data. J Biol Chem, 2007. 282:28791-28799.
Alper H, Jin Y S,Moxley J F, et al. Identifying gene targets for the metabolic engineering of lycopene biosynthesis in Escherichia coli. Metab Eng, 2005. 7:155-164.
Choi H S, Sang Y L, Kim T Y, et al. In silico identification of gene amplification targets for improvement of lycopene production. Appl Environ Microbiol, 2010. 76:3097-3105.
Meng H, Lu Z,Wang Y, et al. In silico improvement of heterologous biosynthesis of erythromycin precursor 6-deoxyerythronolide B in Escherichia coli. Biotechnol Bioprocess Eng, 2011. 16:445-456.
Meng H,Wang Y, Hua Q, et al.In silico analysis and experimental improvement of taxadiene heterologous biosynthesis in Escherichia coli. Biotechnol Bioprocess Eng, 2011. 16:205-215.
基于选择性监测质谱(selectedreaction monitoring,SRM)的靶向蛋白质组方法可以直接有效地对相关代谢途径中酶丰度进行定量 。相对于转录组分析,蛋白质组信息能够更加直观地反映基因表达状况和细胞代谢状态。Redding-Johanson等 利用靶向蛋白质组学的方法对表达酵母来源MVA途径的产萜类大肠杆菌进行了系统研究。他们发现酵母来源甲羟戊酸激酶(MK)和磷酸甲羟戊酸激酶(PMK)是潜在的代谢瓶颈。基于该结果,通过密码子优化和替换强启动子的方法从翻译和转录两个方面有效地提高了MK和PMK蛋白在细胞内水平,从而使紫槐二烯的产量提高了3倍。代谢组分析技术的进步也进一步便于代谢网络中各种代谢物的定量。Weaver等 结合采用靶向蛋白组和代谢组分析方法对产萜类大肠杆菌中MVA前体途径和紫槐二烯异源合成模块中的酶和代谢分别进行了定量分析。结合局部网络中酶和代谢物的浓度信息,他们利用常微分方程构建了该代谢通路的动力学模型。通过对模型进行全局敏感性分析发现两个重要的信息:①紫槐二烯合成酶的浓度与紫槐二烯的产量相关;②解除FPP对MK酶的反馈抑制作用,不影响产物合成。他们又进一步证实了由动力学模型分析得出的以上两个结论与实验结果是一致的。随着各种组学分析方法可靠性和检测通量的增加,结合运用这些方法可以帮助更准确地理解更大范围的细胞网络动态行为,更加便于对工程菌株进行靶点诊断分析。
Foo J L, Jensen H M,Dahl R H, et al. Improving microbial biogasoline production in Escherichia coli using tolerance engineering. MBio, 2014. 5:e01932.
Hess B M, Xue J F, Markillie L M, et al. Coregulation of Terpenoid Pathway Genes and Prediction of Isoprene Production in Using Transcriptomics. PLoS One, 2013. 8:e66104.
Batth T S, Keasling J D, Petzold C J.Targeted proteomics for metabolic pathway optimization. Methods Mol Biol, 2012. 944: 237-249.
Redding-Johanson A M Batth T S,Chan R, et al. Targeted proteomics for metabolic pathway optimization: application to terpene production. Metab Eng, 2011. 13:194-203.
Weaver L J,Sousa M M L,Wang G, et al. A kinetic-based approach to understanding heterologous mevalonate pathway function in E. coli. Biotechnol Bioeng, 2015. 112:111-119.
Song C W, Lee J, Lee S Y.Genome engineering and gene expression control for bacterial strain development. Biotechnol J, 2015. 10:56-68.
Ma Y, Zhang L, Huang X.Genome modification by CRISPR/Cas9. FEBS J, 2014. 281(23):5186-5193.
2 细胞网络重构的高效遗传操作及DNA装配方法
系统生物学的方法可以为高产菌株的设计提供大量全基因组范围的潜在靶点。如何快速地对所有潜在靶点及各种靶点组合进行精细改造与测试,并将所有关键靶点进行集成以最终获得满足工业化生产的高产菌株,是亟待解决的问题。过去十多年的合成生物学研究,积累了许多精细可控的基因表达调控元件以及元件的定量设计方法,为精细的调控基因的表达提供了丰富的元件。同时,高效的基因编辑技术也被开发来实现染色体范围多靶点快速改造与集成 。以大肠杆菌系统为例,传统同源重组打靶质粒构建,重组效率低。以噬菌体重组系统开发的λ大肠杆菌等重组技术直接用外源线性双链或单链DNA分子打靶染色体上大量靶点,大大缩短了大肠杆菌基因每次敲除的周期,提高了重组效率。尤其是线性单链DNA作为靶分子时,重组效率达到了90%。以线性单链DNA打靶为基础,Wang等 进一步开发了功能更强大的多元自动化染色体改造方法(multiplex automated genome engineering, MAGE),通过设计自动化可重复循环进行染色体改造的实验装备,在数天内快速完成对染色体内大量靶点重新洗牌。笔者以大肠杆菌番茄红素合成工程菌为优化对象,采用MAGE技术对潜在的24个与萜类合成有关的关键基因靶点的RBS区域的序列进行了进化改造。3天后, 从数以亿计的突变克隆中成功地筛选到了高产菌株。该方法实现了染色体上大量位点的并行改造,大大提高了基因编辑的效率。CRISPR/Cas技术是被最新开发的一个更为简易高效的染色体编辑技术 。CRISPR(clustered regularly interspaced short palindromic repeats)/Cas (CRISPR associated)是天然存在于细菌和古细菌中的一种免疫系统。CRISPR/ Cas系统由crRNA、tracrRNA和Cas9蛋白组成。crRNA负责识别入侵的靶点序列,crRNA/tracrRNA与Cas9蛋白结合,然后引导Cas9对DNA双链进行切割。近年来,研究人员利用CRISPR/Cas特异性识别和染色体双链切割功能,将CRISPR/ Cas开发成高效地基因组敲除技术,并在哺乳动物细胞、真菌及细菌等系统中取得成功。该方法重组效率极高,能够有效地进行多靶点的同时改造,因此将在高产菌株全基因组范围靶点的改造中发挥越来越大的作用。对于染色体序列的修改,利用人工设计的非编码RNA可以更加方便地调控相应基因在转录、翻译水平的抑制 。
除高效的染色体编辑技术外,复杂的细胞网络优化也对DNA大片段组装乃至染色体拼接提出了更高的要求 。近年来,研究人员开发了一系列高效的体外拼接方法,包括生物砖系统(BioBricks、BglBricks) 、In-FusionTM同源重组 、Gibson等温法 、非序列及非连接依赖性克隆(SLIC) 等,都能够很好地用于几十kb的DNA片段。相比于体外连接方法,借助酵母强大的同源重组能力,可更快速地实现许多带有特定同源末端的DNA片段连接成大片段的DNA分子,且具有相当高的精确性,该方法显示出了巨大的应用前景 。目前,研究人员已经成功利用该技术重构了番茄红素的生物合成基因簇、宏基因组来源的聚酮生物合成基因簇等。Wingler等 在酵母系统开发了迭代重组方法,实现了多基因途径的快速组装和优化。Sleight等 也开发了随机BioBrick组装方法进行基因回路和番茄红素途径的随机装配,结合高通量筛选方法实现途径的快速优化。虽然DNA合成技术在组装长度、准确性、高通量、自动化、低成本等方面取得了巨大进步,但这些技术仍然存在着各自的缺点,需要进一步的发展和整合,以适应合成生物学发展的需要。
3 人造细胞代谢网络的适配与重编程
3.1 代谢网络的动态适配
对于自然进化而来的微生物,随着其生长阶段和环境的变化,细胞内基因表达也是一个动态变化的过程,以满足细胞以最经济的方式运行。传统方法通过诱导型或组成型启动子调控下游合成途径或强化前体途径限速步骤基因表达、抑或是敲除分支途径基因的方法,虽能在一定程度上提高目标产物的合成,但这些策略无法使关键基因的表达动态地适应菌株生长变化和环境扰动,也无法使细胞实时地处在前体供应与前体消耗的动态平衡下。这些内源代谢途径与下游途径基因表达的不适配,往往会造成细胞在酶水平和代谢物水平的浪费与生长负担。如当胞内前体流量供应改善后,若下游途径的表达维持不变,会引起中间代谢物的大量积累并造成细胞毒性和相关酶的反馈抑制。反之,当胞内前体流量供应降低后,下游途径的表达也可以适量减弱,从而减弱细胞的负担。因此利用动态的调控回路,协同上游途径和下游途径的表达,可以经济有效地提高菌株产物合成 。针对这样的改造目标,能响应细胞内特定代谢物的元件以及合成生物学基因调控回路是非常重要的改造工具。
Sander J D, Joung J K. CRISPR-Cas systems for editing, regulating and targeting genomes. Nat Biotechnol, 2014. 32:347-355.
Chaudhary A K, Na D, Lee E Y. Rapid and high-throughput construction of microbial cell-factories with regulatory noncoding RNAs. Biotechnol Adv, 2015.
Ellis T, Adie T, Baldwin G S. DNA assembly for synthetic biology: from parts to pathways and beyond. Integr Biol (Camb), 2011. 3: 109-118.
Anderson J, Dueber J E, Leguia M, et al. BglBricks: A flexible standard for biological part assembly. J Biol Eng, 2010. 4(1):1.
Sleight S C, Baltley B C, Lieviant J A, et al. In-Fusion BioBrick assembly and re-engineering. Nucleic Acids Res, 2010. 38(8):2624-2636.
Gibson D G.Synthesis of DNA fragments in yeast by one-step assembly of overlapping oligonucleotides. Nucleic Acids Res, 2009. 37:6984-6990.
Li M Z, Elledge S J. Harnessing homologous recombination in vitro to generate recombinant DNA via SLIC. Nat Meth, 2007. 4:251-256.
Wingler L M,Cornish V W.Reiterative Recombination for the in vivo assembly of libraries of multigene pathways. Proc Natl Acad Sci U S A, 2011. 108:15135-15140.
为了实现这样的动态调控,Farmer 等 首次在产番茄红素大肠杆菌中设计了响应细胞内代谢物乙酰磷酸的动态回路,通过乙酰磷酸胞内浓度的动态变化来调控番茄红素下游合成模块的表达,有效促进了番茄红素的合成。Xu等 利用了脂肪酰-CoA响应的转录调控因子FadR构建了基因回路,动态调控乙酰-CoA羧化酶和脂肪酸合酶的表达,从而提高菌株脂肪酸合成。但是在多数情况下,缺乏可用于特定回路设计的响应元件来控制和协同基因的动态表达。结合转录组学等系统生物学方法,可以发现有效的调控因子用于回路设计。研究表明,法呢基焦磷酸(FPP)是倍半萜合成的直接前体, 需要很强的FPP代谢流量才能满足萜类的大量合成需要。但是FPP具有很强的细胞毒性,在引入优化后的MVA途径的E. coli萜类合成高产菌株中,往往由于下游模块的表达水平无法与FPP供应水平相协同,FPP会在细胞内积累并造成细胞生长的抑制。Dahl等 利用转录组分析的方法,发现了能够对萜类代谢中间体FPP作出压力响应的调控元件。利用该调控元件设计了控制紫槐二烯和甲羟戊酸合成途径的动态回路,有效地促进了紫槐二烯的合成。Yuan等 利用实时定量PCR (qRT-PCR)的方法分析了酿酒酵母菌株中相关基因的表达情况,发现ERG11、ERG2、ERG3三个基因的表达在胞内麦角固醇积累时是下调的,并鉴定了受麦角固醇调控的启动子。当利用这些麦角固醇响应启动子调控 ERG9表达时,异源萜类化合物的产量可以提高2~5倍。
Sleight S C, Sauro H M. Randomized BioBrick assembly: a novel DNA assembly method for randomizing and optimizing genetic circuits and metabolic pathways. ACS Synth Biol, 2013. 2:506-518.
McNerney M P, Watstein D M, Styczynski M P. Precision metabolic engineering: The design Styczynski of responsive, selective, and controllable metabolic systems. Metab Eng, 2015,31:123-131.
Farmer W R, Liao J C. Improving lycopene production in Escherichia coli by engineering metabolic control. Nat Biotechnol, 2000. 18:533-537.
Xu P, Li L,Zhang F, et al.Improving fatty acids production by engineering dynamic pathway regulation and metabolic control. Proc Natl Acad Sci U S A, 2014. 111(31):11299-11304.
Dahl R H, Zhang F Z, Alonso-Gutierrez J, et al. Engineering dynamic pathway regulation using stress-response promoters. Nat Biotechnol, 2013. 31:1039-1146.
Yuan J, Ching C B. Dynamic control of ERG9 expression for improved amorpha-4,11-diene production in Saccharomyces cerevisiae. Microb Cell Fact, 2015. 14:38.
Zhao J,Li Q,Sun T, et al.Engineering central metabolic modules of Escherichia coli for improving beta-carotene production. Metab Eng, 2013. 17:42-50.
Wang J F, Meng H L,Xiong Z Q, et al.Identification of novel knockout and up-regulated targets for improving isoprenoid production in E. coli. Biotechnol Lett, 2014. 36:1021-1027.
3.2 代谢网络的系统化重构与适配
为了进一步提高菌株产量,研究人员从细胞全局网络的角度开展了菌株的系统化重构和适配研究,并取得了一系列的成果。Zhao等 采用启动子工程和模块化代谢工程策略对大肠杆菌中心代谢网络中涉及萜类前体合成、NADPH、ATP供应的靶点进行系统的测试与集成改造,成功提高了β-胡萝卜素的合成。Meng等 以大肠杆菌iAF1260模型为基础,将该全基因组in silico代谢流量分析应用到了大肠杆菌萜类合成体系优化中,并由Wang等 成功鉴定了一系列能提高萜类合成的新靶点。利用酿酒酵母的甲羟戊酸途径进行萜类的高效合成已经比较成熟。为了进一步提高酿酒酵母萜类合成的能力,Meng和Sun等 以酿酒酵母iMM904全基因组模型为基础,利用FDCA和MOMA算法,预测并获得了一系列能提高萜类合成、但不显著影响菌体生长的新的改造靶点。随后通过基因敲除实验,以很高的预测准确度发现了SER3、SER33、SOR1等10个靶点的单敲除能够使萜类合成提高8~10倍。此外,为了实现酵母前体供应和下游合成途径代谢流的平衡,Ding等 采用计算机辅助设计的方法预测并发现了最高效的GGPP合成酶,并通过实验成功解决了一个酵母系统紫杉醇关键中间体taxadiene合成的关键瓶颈。
由于异源聚酮合成模块和底盘细胞前体模块之间的不适配,使得红霉素大肠杆菌异源合成菌株的效率非常低。为了改善红霉素等聚酮类化合物在大肠杆菌中的合成,笔者研究组以红霉素前体6dEB合成菌株为基础,首先结合运用基因组尺度的in silico代谢网络分析和比较转录组分析系统地研究了聚酮合成模块(以红霉素前体6dEB合成模块为例)和内源代谢模块在细胞代谢和基因转录两个层次的相互作用 。基因组尺度的in silico代谢网络分析反映了在理论条件下细胞达到6dEB最大合成产率的代谢流量分布情况及潜在的关键靶点。以最大生长速率(Vmax,growth)(理想状况下)和6dEB最大合成速率(Vmax,synthesis)(目标状况下)作为目标函数,通过基于FDCA方法的in silico代谢网络分析发现了大量潜在的改造靶点。另一方面,通过比较转录组分析在启动6dEB合成相关基因表达并添加丙酸盐后,发现共有629个基因的表达产生明显变化。这些基因分布在40个不同的代谢模块中。随后采用了snRNA技术,快速地对大量潜在靶点进行了系统改造,通过弱化提高聚酮化合物产量,发现20多个可使产量提高20%以上的新靶点 。
相比于复杂的细胞网络改造,利用合成生物学方法在体外重构局部代谢网络,可以更好地研究网络特性、指导细胞内代谢网络的重构。Zhu等 首先在体外重构了萜类前体合成甲羟戊酸途径和法尼希下游合成,并对该系统各组分进行分析。然后以此为基础,通过在大肠杆菌中定量分析和表达甲羟戊酸途径与法尼希合成途径中的各个重要酶,成功地提高了大肠杆菌法尼希的合成。
4 结 论
随着DNA合成与装配技术越来越成熟,大量天然化合物异源合成途径被成功组装,高效微生物异源体系设计的焦点已经转移到如何通过对细胞全局网络的设计来实现菌株的高产,并最终可用于生产实践。系统生物学和合成生物学分别为菌株细胞网络分析诊断与遗传改造提供了强大的工具与方法。系统生物学技术,特别是蛋白质组、代谢物组等检测与数据分析方法的日益成熟使生物工程师们可以对复杂的细胞代谢过程有更实时、全局的诊断。基于这些数据建立相关的动态模型,分析这些整合了动力学数据、细胞调控特性的全基因组网络模型,可以更加准确地发现一系列新的改造靶点和靶点组合,这将彻底改变传统以随机诱变和筛选为主的黑箱方法来进行工业菌株改造 。同时,合成生物学技术的发展为菌株的改造提供了强大、高效的DNA操作方法、染色体编辑技术,以及理性的细胞网络设计方法。这些合成生物学方法和策略为快速实现菌株改造和测试奠定了基础。因此,基于系统-合成生物学的人工细胞网络设计与优化方法将成为后基因组时代微生物工业菌株优化的重要策略。
Sun Z Q, Meng H L, Li J, et al. Identification of novel knockout targets for improving terpenoids biosynthesis in Saccharomyces cerevisiae. PLoS One, 2014, 9:e112615.
Ding M Z, Yan H F, Li L F,et al.Biosynthesis of Taxadiene in Saccharomyces cerevisiae : selection of geranylgeranyl diphosphate synthase directed by a computer-aided docking strategy. PLoS One, 2014. 9:1.
王勇,等.一种提高大肠杆菌异源合成聚酮类化合物的方法与用途。中国专利申请号:201510253481.1
Wang J F, Li S Y, Xiong Z Q, et al.Pathway mining-based integration of critical enzyme parts for de novobiosynthesis of steviolg lycosides sweetener in Escherichia coli. Cell Res, doi: 10.1002/biot. 201500351.
Zhu F,Zhong X,Hu X,et al.In vitro reconstitution of mevalonate pathway and targeted engineering of farnesene overproduction in Escherichia coli. Biotechnol Bioeng, 2014. 111:1396-1405.
Ng C Y, Khodayari A,Chowdhury A,et al., Advances in de novo strain design using integrated systems and synthetic biology tools. Curr Opin Chem Biol, 2015, 28:105-114.
■ 反馈服务编码 W3616
10.3969/j.issn.1674-0319.2015.06.005


