高危型HPV感染宫颈病变中E6/E7 mRNA的表达水平
刘甚佳,尹利荣,李洪林
宫颈癌是严重危害妇女健康和生命的恶性肿瘤,发病率位居妇科恶性肿瘤的第二位,是由其癌前病变,即宫颈上皮内瘤变(cervical intraepithelial neo⁃plasia,CIN)逐步发展形成。宫颈癌及其癌前病变的发生和发展与高危型人乳头状瘤病毒(HR-HPV)的感染密切相关。人乳头状瘤病毒(HPV)的早期基因E6/E7具有较强的抗原性,且在宫颈高级别病变中持续高表达,其在感染过程中产生的早期蛋白E6/E7在宿主细胞中的表达是导致宫颈癌的关键因素[1]。目前应用HPV E6/E7 mRNA检测的方法在宫颈疾病筛查中尚未广泛普及,临床资料相对较少,本研究应用支链DNA(b-DNA)技术对HR-HPV感染患者宫颈脱落细胞进行HPV E6/E7 mRNA检测,了解其在宫颈上皮内瘤变和宫颈癌脱落细胞中的表达情况,以评估此方法作为宫颈癌及癌前病变筛查辅助指标的临床应用价值。
1 资料与方法
1.1 一般资料 连续入选2013年11月—2014年5月在我院妇科就诊的HR-HPV感染者265例,研究组共165例,其中CINⅠ级组88例,CINⅡ级组33例,CINⅢ级组28例,宫颈癌组16例;余100例正常宫颈或慢性炎症者为对照组。均无先天性遗传病、严重内科疾病及其他部位肿瘤。
1.2 试剂及仪器 宫颈稳态试剂盒(Diacarta Inc,美国)及96孔检测板购自科蒂亚(新乡)生物技术有限公司;DiaCarta台式冷光仪;恒温箱(型号QS071)。
1.3 方法
1.3.1 HPVE6/E7 mRNA的检测 (1)标本采集:使用液基细胞学Autocyteprep系统专用刷插入受检者子宫颈旋转8~10圈,收集宫颈口及颈管脱落上皮细胞,然后将刷头部放入装有CytoRich保存液的小瓶内供HPVE6/E7 mRNA检测。(2)实验步骤:将液基细胞学标本放入离心管水平离心2次(3 000 r/min,5 min),使标本同质化。加入600 μL细胞裂解液和5 μL蛋白酶K,吹打混匀,放入65℃恒温箱1 h,中间取出标本管振荡2~3次,使细胞充分裂解,待测。按照宫颈稳态试剂盒说明书操作及配置相关溶液并布板,其中阳性对照液,空白对照液均做复孔。经过信号放大,加入标记荧光物质的底物后,在冷光仪上检测。检测结果为光子数,经计算软件转换为拷贝数。可检测HPV16、18、31、33、35、39、45、51、52、56、58、59、68、66、26、55、61、73、83等19种高危亚型的HPVE6/E7 mRNA片段。
1.3.2 薄层液基细胞学(TCT)检测 标本采集方法同上,采用新柏氏TCT检测系统,使用TBS描述性诊断报告,包括正常、非典型鳞状细胞(ASC)、低级别鳞状上皮内瘤变(LSIL)、高级别鳞状上皮内瘤变(HSIL)、宫颈癌等。
1.4 统计学方法 使用SPSS 19.0软件进行统计分析,计数资料以例(%)表示,比较采用χ2检验,计量资料以M(P25,P75)表示,比较采用Kruskal-wallis H检验,检验水准α=0.05,两两比较时校正检验水准为α=0.005。
2 结果
2.1 各组HPVE6/E7 mRNA阳性率及表达量比较 CINⅡ级、CINⅢ级和宫颈癌组的HPV E6/E7 mRNA阳性率及表达量高于对照组和CINⅠ级组(P<0.005)。CINⅡ级、CINⅢ级和宫颈癌组间无明显差异,正常组与CINⅠ级组无明显差异(均P>0.005),见表1。
2.2 不同TCT诊断级别中HPV E6/E7 mRNA表达水平比较 HSIL和宫颈癌组的HPV E6/E7 mRNA阳性率及表达量显著高于正常、ASC和LSIL组(P<0.05),见表2。

Tab.1 Comparison of HPV E6/E7 mRNA positive rate and transcription levels between five groups表1 各组HPV E6/E7 mRNA阳性率及表达水平比较
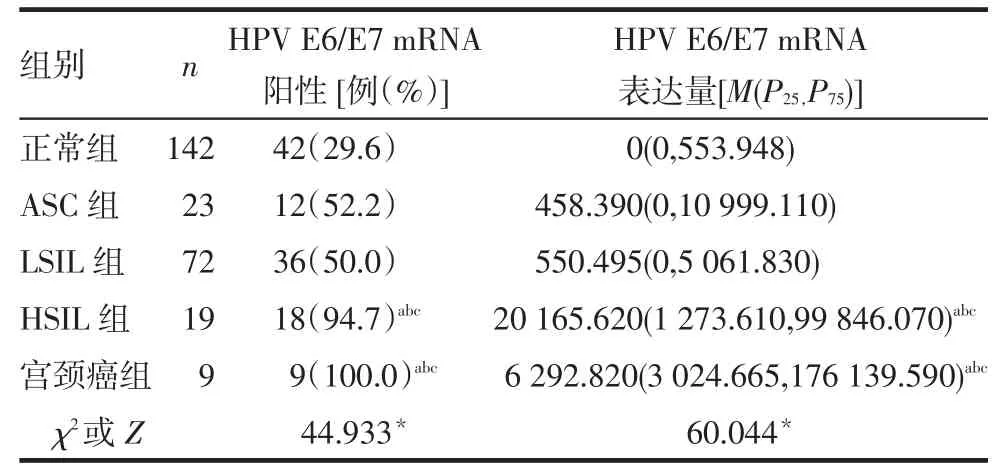
Tab.2 The transcription level of HPV E6/E7 mRNA in different TCT diagnosis level表2HPV E6/E7 mRNA在不同TCT诊断级别中的表达情况
3 讨论
3.1 HPV E6/E7 mRNA在宫颈癌及CIN中的研究现状及意义 宫颈癌是目前唯一病因明确的妇科恶性肿瘤,与HR-HPV的持续感染有关[2]。99.7%的宫颈癌组织中可检测到HPV,未感染HPV者几乎不会发生宫颈癌(阳性预测值>99.0%)[3]。HPV产生的早期蛋白E6/E7在宿主细胞中的过度表达是导致宫颈癌的关键因素。其主要机制为:HPV感染宿主细胞后,其E6/E7致癌基因可显著抑制p53和Rb这2种抑癌蛋白,从而干扰细胞周期调节,促使细胞无规则生长[4]。其中,E6蛋白通过结合宿主细胞p53抑癌基因使之失活,导致细胞凋亡途径失能促使细胞永生化;E7蛋白通过使Rb失活,影响细胞周期,启动端粒酶基因,从而使细胞永生,癌变发生[5]。而HPV E6/E7 mRNA正是病毒癌基因的转录产物,其表达会导致宿主细胞最终向恶性方向转化[6]。然而,大部分女性感染HPV只是暂时性的,即使HPV持续存在,但并不必然导致宫颈癌的发生,80%的HRHPV感染是一过性的[7]。HR-HPV感染的女性中大约只有1%会逐渐发展为宫颈癌[8]。HPV致癌过程不是一个匀速过程,病毒在宿主细胞内的“整合”增加了基因不稳定性,标志着恶性转化的开始[9]。游离状态的病毒在人体正常免疫调节下多数可以自动清除,只有病毒整合至宿主细胞数量到达一定程度时,才会发展为高级别病变甚至发生宫颈癌。CIN存在自然转归的问题,随着CIN级别的提高,病变的逆转率逐渐下降,病变的进展率逐渐升高,说明低级别CIN有可逆性,而高级别CIN与发展为浸润性癌有一定关系[10]。HPV E6/E7 mRNA在宫颈组织中表现了癌基因活性,其水平与病变的严重程度相关。HPV E6/E7 mRNA检测可以筛查出真正高级别病变,为临床筛查方法提供了新选择。
3.2 HPV E6/E7 mRNA检测筛查高级别宫颈病变的诊断性能和优势 本试验采用b-DNA技术对HPV E6/E7 mRNA进行检测,结果显示:在HR-HPV感染者中,HPV E6/E7 mRNA在正常组、CINⅠ、CINⅡ、CINⅢ、宫颈癌组中的阳性表达率和表达量基本随病理级别的升高呈增强趋势,提示HPV E6/E7 mRNA水平与病变的严重程度相关。CINⅡ、CINⅢ、宫颈癌的HPV E6/E7阳性率及表达量明显高于正常组和CINⅠ组,说明这种检测方法在筛查高级别宫颈病变方面有优势,与Liu等[11-12]研究结果一致。低级别CIN(CINⅠ)有可逆性,而高级别CIN (CINⅡ、CINⅢ)与发展为浸润型癌有关,HPV E6/E7 mRNA检测可分流高、低危人群。另外,本试验结果还显示HPV E6/E7 mRNA检测随着TCT诊断级别的提高,其阳性率及表达量也基本呈上升趋势,也说明HPV E6/E7 mRNA检测用于宫颈病变筛查结果与细胞学诊断在一定程度上相符合。有文献报道在检测高级别宫颈病变中,HPVE6/E7 mRNA较HPV DNA具有更高特异性(85%vs 40.83%)[13]。测定E6/ E7 mRNA有助于区分一过性感染(低表达)和发展为癌(高表达)的人群,尤其对于TCT示ASC-US的单纯一过性感染及可逆的低级别宫颈病变(CINⅠ)患者,E6/E7 mRNA检测可给予良好补充和有效提示,可以有效排除风险,从而减轻患者的心理压力,避免过度治疗。
HPVE6/E7 mRNA检测作为一种新的宫颈病变辅助诊断指标,其操作简单,无创、安全、价格低廉的优点适合于临床宫颈病变的筛查,特别对于筛查出高级别的宫颈病变有重要意义。
[1]Lu X,Lin Q,Lin M,et al.Multiple-integrations of HPV16 genome and altered transcription of viral oncogenes and cellular genes are asso⁃ciated with the development of cervical cancer[J].PLoS One,2014,9(7): e97588.doi:10.1371/journal.pone.0097588.eCollection 2014.
[2]Schwarz JK,Siegel BA,Dehdashti F,et al.Metabolic response on posttherapy FDG-PET predicts patterns of failure after radiotherapy for cervical cancer[J].Int J Radiat Oncol Biol Phys,2012,83(1):185-190. doi:10.1016/j.ijrobp.2011.05.053.
[3]LI YL.The clinical research progress of HPV infection and cervical lesion[J].Medical Journal of the Chinese People’s Armed Police Forces,2012,23(2):93-96.[李亚里.HPV感染与宫颈癌前病变的临床研究进展[J].武警医学,2012,23(2):93-96].
[4]Shaikh F,Sanehi P,Rawal R.Molecular screening of compounds to the predicted Protein-Protein Interaction site of Rb1-E7 with p53-E6 in HPV[J].Biomedical Informatics,2012,8(13):607-612.doi: 10.6026/97320630008607.
[5]Belyaeva TA,Nicol C,Cesur O,et al.An RNA aptamer targets the PDZ-binding motif of the HPV16 E6 oncoprotein[J].Cancers(Ba⁃sel),2014,6(3):1553-1569.doi:10.3390/cancers6031553.
[6]Rampias T,Boutati E,Pectasides E,et al.Activation of Wnt signaling pathway by buman papillomavirus E6 and E7 oncogenes in HPV16-positive oropharyngeal squamous carcinoma cells[J].Mol Cancer Res,2010,8(3):433-443.doi:10.1158/1541-7786.MCR-09-0345.
[7]Mehlhorn G,Obermann E,Negri G,et al.HPV L1 detection discrimi⁃nates cervical precancer from transient HPV infection:a prospec⁃tive international multicenter study[J].Mod Pathol,26(7):967-974. doi:10.1038/modpathol.2012.233.
[8]Josefsson AM,Magnusson PK,Ylitalo N,et al.Viral load of human papilloma virus 16 as a detetmination for development of cervical carcinoma in situ:a nested case-control study[J].Lancet,2000,355 (9222):2189-2193.doi:10.1016/S0140-6736(00)02401-6.
[9]Pett M,Coleman N.Integration of high-risk human papillomavirus a key event in cervical carcinogenesis[J].J Pathol,2007,212(4):356-367.doi:10.1002/path.2192.
[10]Discacciati MG,da Silva ID,Villa LL,et al.Prognostic Value of DNA and mRNA E6/E7 of Human Papillomavirus in the Evolution of Cervical Intraepithelial Neoplasia Grade 2[J].Biomark Insights, 2014,13(9):15-22.doi:10.4137/BMI.S14296.eCollection 2014.
[11]Liu TY,Xie R,Luo L,et al.Diagnostic validity of human papilloma⁃virus E6/E7 mRNA test in cervical cytological samples[J].J Virol Methods,2014,196:120-125.doi:10.1016/j.jviromet.2013.10.032.
[12]Li SR,Liu DQ,Hu FY.TNF-α,E2,E6 and E7 in Different Cervi⁃cal Lesion and Correlation Analysis between Them[J].J Int Obstet Gynecol,2014,41(3):321-323.[李世蓉,刘冬青,胡凤英.TNF-α、E2、E6及E7在不同宫颈病变组织中的表达及其相关性分析[J].国际妇产科学杂志,2014,41(3):321-323].
[13]Munkhdelger J,Choi Y,Lee D,et al.Comparison of the perfor⁃mance of the NucliSENS EasyQ HPV E6/E7 mRNA assay and HPV DNA chip for testing squamous cell lesions of the uterine cervix[J]. Diagn Microbiol Infect Dis,2014,79(4):422-427.doi:10.1016/j.di⁃agmicrobio.2014.04.004.

