D-塔格糖-3-差向异构酶及其应用
王琼,王远山
(浙江工业大学 生物工程研究所,浙江 杭州 310000)
1 前言
稀有糖是常见单糖(如半乳糖、葡萄糖等)分子中至少一个羟基被氢原子或其他基团所取代而形成的化合物[1]。据推测,在自然界中约有50种稀有糖,至今已发现34种,D-型稀有糖与L-型稀有糖各占一半[2]。其中D-塔格糖、D-阿洛酮糖、D-阿洛糖、L-系列糖和木糖醇等稀有糖具有及其良好的理化性质及活性功能(表1),广泛应用于甜味剂、医药中间体和保健食品体的生产等领域[2]。目前稀有糖的生产方法主要有化学合成法和生物转化法。化学合成法涉及到催化加氢、加成反应、Ferrier重排等反应,其中关环转换合成法和两步合成法是具有一定竞争力的方法。但这些方法反应存在着反应步骤多、需保护和脱保护、条件苛刻、产率较低和副产物多等不足,还需要改进和完善。近年来,以差向异构酶为基础的稀有糖生物转化法生产由于其良好的立体选择性、温和的反应条件等优点而日益受到重视[1-2]。
差向异构酶,又称变旋酶,是催化单糖分子中某一个不对称碳原子的构型变化的酶。目前报道的可应用于稀有糖大规模生产的差向异构酶,仅有 D-塔格糖-3-差向异构酶 (D-tagatose 3-epimerase,DTEase)和D-阿洛酮糖-3-差向异构酶(D-psicose 3-epimerase,DPEase)。
近年来,利用廉价单糖为原料,通过差向异构酶实现己酮糖C-3位置间的差向异构反应制备稀有糖,已成为生化工程领域中的研究热点。本文主要介绍了DTE酶家族成员DTEase和DPEase的来源、酶学特性、蛋白结构以及DTE酶家族在稀有糖生产中的应用现状等,最后展望了DTE酶家族未来的发展方向。

表1 常见稀有糖及其生物学功能
2 DTE酶家族
2.1 DTE酶家族成员的来源
二十世纪九十年代,日本Izumori团队首次报道了来源于菊芋假单胞菌 (Pseudomonas cichorii ST-24)中的一种酶可催化D-山梨糖(D-sorbose)和D-塔格糖之间的差向异构反应[6],因其最适底物为塔格糖,将其命名为D-塔格糖3-差向异构酶(DTEase)。2006年,韩国Oh团队成功克隆表达了根癌农杆菌 (Agrobacterium tumefaciens)中的DTEase基因,由于该酶对D-阿洛酮糖有较强的底物特异性,后将其更名为D-阿洛酮糖3-差向异构酶[7](DPEase)。 DTEase和 DPEase可催化相似系列的反应,即单糖C-3位置的差向异构反应,因此共同构成了一个新家族—DTE酶家族。至今为止,已先后在类球红细菌(Rhodobacter sphaeroides SK011)[8]、 梭状芽胞杆菌(Clostridium cellulolyticum H10)[9]、瘤胃球菌(Ruminococcus sp.)[10]和 Desmospora sp.[11]中发现了新的 DTE 酶家族成员,如表2所示。
2.2 DTE酶家族的酶学特性
DTEase和DPEase虽催化相同系列的反应,但因来源不同,两者的最适pH、最适温度、半衰期、底物特异性等生化特性存在较大差异,表3列出了不同DTE酶家族成员的生化特性。据表3可知,DTE家族酶适宜中性或弱碱性环境;与其它来源的DTEase相比,R.sphaeroides的DTEase耐热性最差;而C.cellulolyticum中的DPEase半衰期明显比其他来源的DPEase的半衰期长,具有更好的稳定性;A.tumefaciens的DPEase的米氏常数Km值最小,对D-果糖的亲和力最强;在D-果糖与D-阿洛酮糖的差向异构反应中6种酶表现出不同的平衡比,具体见表3所示。

表2 DTE酶家族成员的来源
2.3DTEase和DPEase的结构
为阐明DTE酶家族的催化机制,Kim和Yoshida先后利用X射线衍射技术解析了DPEase和DTEase的蛋白晶体结构[14-15]。
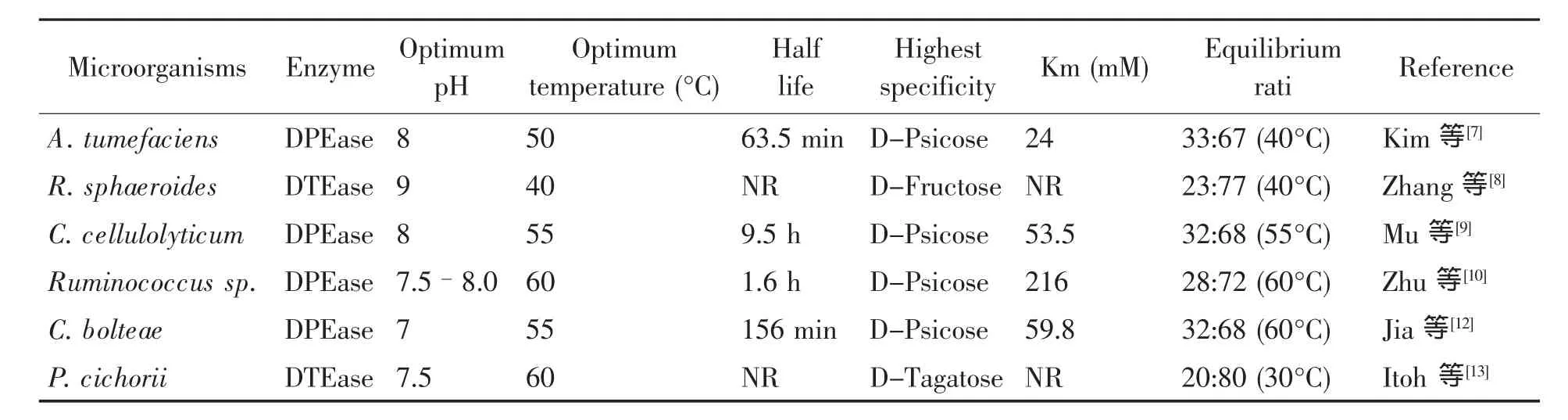
表3 DTE家族酶的酶学特性
2.3.1 A.tumefaciens DPEase晶体结构
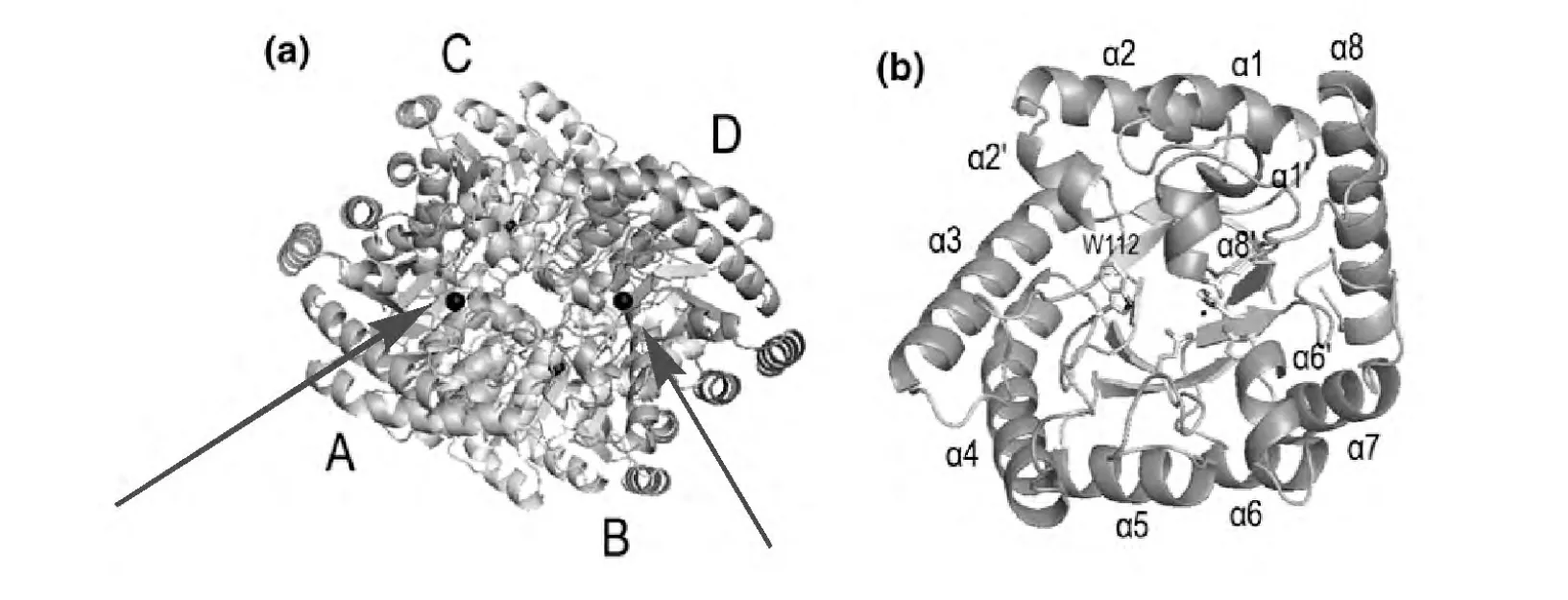
图1 DPEase的分子结构
Kim[14]对DPEase蛋白晶体结构的解析表明,A.tumefaciens DPEase是由A、B、C和D 4个相同的亚基构成的四聚体分子,见图1(a)。图1(a)标出的2个黑点为推测的金属离子结合位点。每个亚基包含8个β折叠和12个α螺旋,而8个β折叠簇被12个α螺旋紧紧环绕,整体呈TIM-桶结构,见图1(b)。该酶为金属离子依赖型酶,金属离子与 Glu150、Asp183、His209 和 Glu244 结合构成活性中心,进行酶催化反应。Zhang等[17]报道Desmospora sp.的DPEase添加Co2+后热稳定性显著提高。

图2 DTEase(a)与 DPEase(b)的分子结构
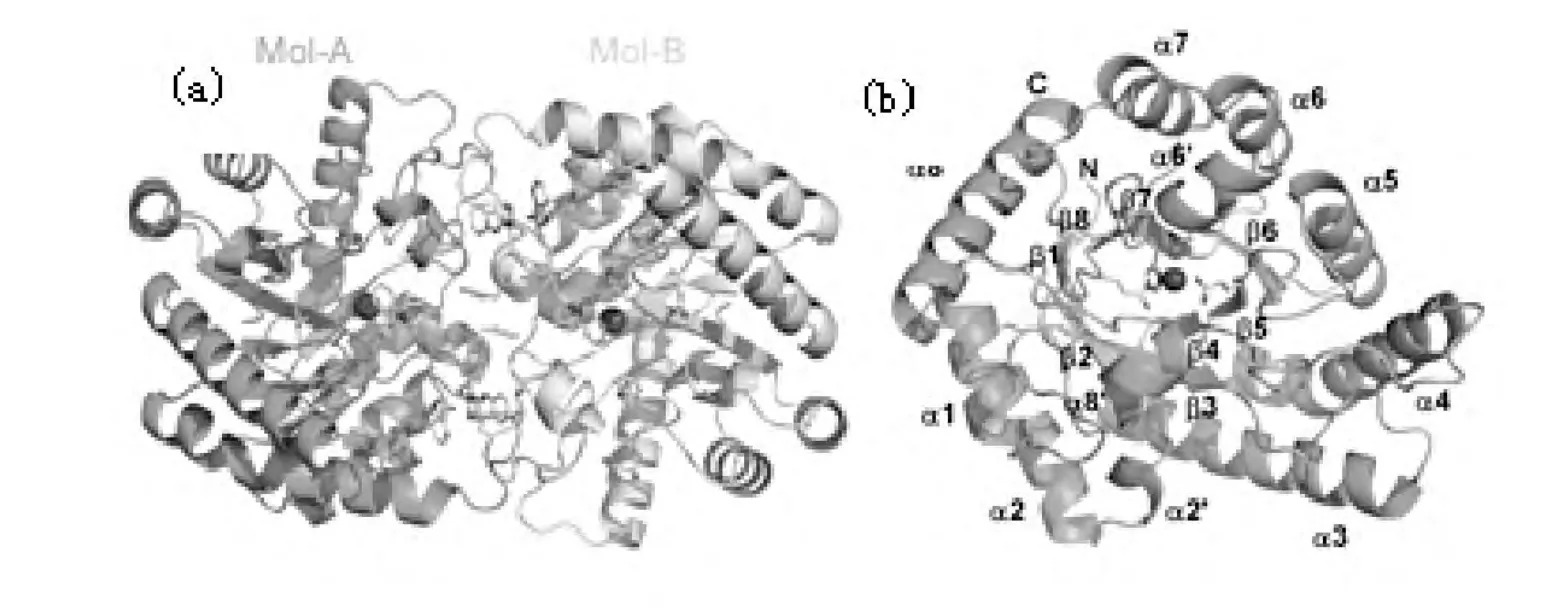
图3 DTEase的分子结构:(a)二聚体结构(b)单体结构
2.3.2 P.cichorii DTEase晶体结构
Yoshida[15]对 P.cichorii DTEase蛋白晶体结构的解析表明,该酶与A.tumefaciens DPEase的晶体结构相似,由Mol-A、Mol-B、Mol-C和 Mol-D组成(图2)。图3(a)显示两个对称的Mol-A和Mol-B亚基构成的二聚体分子。每个亚基包含11个α螺旋和8个β折叠,整体呈现(β/α)8桶状结构,如图 3(b)所示。
3 DTE酶家族在稀有糖生产中的应用
2006年,Izumori团队报道了利用DTE家族酶催化8种己酮糖C-3位置的差向异构反应[16]。图4列出了利用DTEase在D-果糖与D-阿洛酮糖、D-塔格糖与D-山梨糖、L-果糖与L-阿洛酮糖、L-塔格糖与L-山梨糖之间的差向异构反应。除了D-果糖,其余7种己酮糖均为稀有糖[2]。

图4 DTEase催化己酮糖间的差向异构化反应
随后,Izumori等[16]又提出了利用D-塔格糖-3-差向异构酶、醛糖异构酶、醛糖还原酶、氧化还原酶(polyol dehydrogenase,PDH)制备各种稀有糖的Izumoring方法(图5),使得稀有糖的相互转化形成了一个循环网络,对稀有糖的生产具有极其重要的意义。

图5 丁糖、戊糖和己糖类稀有糖的生物酶法转化
3.1 DTE酶家族在D-阿洛酮糖制备中的应用进展
目前,催化D-果糖制备D-阿洛酮糖所利用的酶主要是DTE酶家族的DPEase和DTEase。
2008年,Kim等[17]发现利用DTEase转化果糖制备阿洛酮糖的过程中,添加适量的硼酸盐可以改变原有的化学平衡。因硼酸盐与阿洛酮糖的亲和作用较强,促使反应平衡沿着产物阿洛酮糖方向转化,转化率最高可达64%,相比不添加硼酸盐转化率提高了近两倍。2009年,Lim等[18]通过固定化含有硼酸盐的A.tumefaciens DPEase大规模生产D-阿洛酮糖,该固定化酶催化700 g/L的D-果糖时,D-阿洛酮糖的产量可达441 g/L。2011年,Mu等[19]报道了利用在大肠杆菌中过量表达的梭状芽胞杆菌DPEase催化D-果糖生产D-阿洛酮糖,最终产量可达218 g/L。2013年,Zhang等[11]发现来源于DTE酶家族新成员Desmospora sp.8437中的DPEase与Co2+离子结合后表现出最高酶活,催化500 g/L的D-果糖时D-阿洛酮糖产量达142.5 g/L。目前文献报道的DTE酶家族催化D-果糖生产D-阿洛酮糖的催化反应多在碱性缓冲液中进行,但是在该条件下会产生褐变,同时导致副产物的产生[12]。2014年,Jia等[12]报道了利用异源表达的C.bolteae DPEase在中性缓冲液条件下制备D-阿洛酮糖,转化率达到28.8%。
3.2 DTE酶家族在稀有支链单糖制备中的应用
随着研究的深入开展,研究人员发现DTE酶家族是一种底物谱相对广泛的酶,可以催化稀有支链单糖的差向异构反应。
Rao等[20]报道了利用DTEase催化4-甲基-L-核酮糖(4-C-methyl-L-ribulose)与 4-甲基-L-木酮糖(4-C-methyl-L-xylulose)、4-甲基-D-核酮糖(4-C-methyl-D-ribulose)与 4-甲基-D-木酮糖之间(4-C-methyl-D-xylulose)之间的差向异构化反应,见图6。

图6 DTEase催化4-C-甲基-戊糖的差向异构反应
4 展望
自从Izumori团队首次发现DTEase并借助Izumoring策略成功地实现所有稀有糖的相互转化以来,利用差向异构酶制备各种功能性稀有糖就成为研究热点,研究集中在新的产酶菌株的筛选、催化机理、酶分离纯化、酶固定化等方面。目前已取得了一定进展,为阿洛酮糖等功能性稀有糖的生产提供了一条具有原子经济性和更绿色的途径。
为促进差向异构酶法生产功能性稀有糖的研究和产业化开发,目前仍需在以下方面需加大研究力度:(1)DTE酶家族成员资源的拓展,目前仅有来源于6种菌株中的DTEase和DPEase被确定为DTE家族成员,在底物谱等方面存在着差异。因此,获得新的DTE家族酶将为酶法制备稀有糖的研究提供基础。(2)差向异构酶催化机理的研究,目前对其催化机理研究还处于初步的阶段,对酶的结构与催化功能、立体选择性的关系等尚待深化;(3)差向异构酶催化性能提升,现有的酶的热稳定性、底物特异性等还存在着一些不足,可以利用现代分子生物学技术对其进行分子改造以提高催化性能、拓宽底物谱;(4)差向异构酶应用范围的扩大,要进一步利用其底物特异性,尝试DTE家族在新型的支链稀有糖生产中的应用,以实现功能性稀有糖的高效和绿色生产。
[1]李路,张贵生.稀有糖的合成方法研究[J].有机化学,2008,28(7):1129-1137.
[2]林清泉,刘有才,李丽峰,等.稀有糖的制备及应用最新进展[J].食品与发酵工业.2013, 39(6):146-151.
[3]Oh DK.Tagatose:properties,applications and biotechnological processes[J].Applied Microbiology and Biotechnology,2007,76(1):1-8.
[4]Kubiak,R.L.,R.K.Phillips,et al.Structural and functional studies on a 3'-epimerase involved in the biosynthesis of dTDP-6-deoxy-D-allose[J].Biochemistry,2012,51(46):9375-9383.
[5]Sun Yuanxia,Hayakawa S,Ogawa M,et al.Antioxidant properties of custard pudding dessert containing rare hexose,D-psicose[J].Food Control,2007,18(3):220-227.
[6]Izumori K,Khan AR,Okaya H,et al.A new enzyme,D-ketohexose 3-epimerase from Pseudomonas sp.ST-24[J].Bioscience Biotechnology and Biochemistry,1993,57(6):1037-1039.
[7]Kim HJ,Hyun EK,Kim YS,et al.Characterization of an A-grobacterium tumefaciens D-psicose 3-epimerase that converts D-fructose to D-psicose[J].Applied and Environmental Micro biology,2006,72(2):981-985.
[8]Zhang L,Mu W,Jiang B,et al.Characterization of D-tagatose-3-epimerase from Rhodobacter sphaeroides that converts D-fructose into D-psicose[J].Biotechnology Letters,2009,31(6):857-862.
[9]Mu W,Chu F,Xing Q,et al.Cloning,expression,and characterization of a D-psicose 3-epimerase from Clostridium cellulolyticum H10[J].Journal of Agricultural and Food Chemistry,2011,59(14):7785-7792.
[10]Zhu Y,Men Y,Bai W,et al.Overexpression of D-psicose 3-epimerase from Ruminococcus sp.in Escherichia coli and its potential application in D-psicose production[J].Biotechnology Letters,2012,34(10):1901-1906.
[11]Zhang,W.L.,D.Fang,et al.Characterization of a metal-dependent D-psicose 3-epimerase from a novel strain,Desmospora sp 8437[J].Journal of Agricultural and Food Chemistry,2013,61(47):11468-11476.
[12]Jia M,Mu WM,Chu FF,et al.A D-psicose 3-epimerase with neutral pH optimum from Clostridium bolteae for D-psicose production:cloning,expression,purification and characterization[J].Applied Microbiology and Biotechnology,2014,98(2):717-725.
[13]Itoh H,Okaya H,Khan AR,et al.Purification and characterization of D-tagatose 3-epimerase from Pseudomonas sp.ST-24[J].Bioscience Biotechnology and Biochemistry,1994,58 (12):2168-2171.
[14]Kim K,Jung W,Rhee S.Crystal structure of D-psicose 3-epimerase from Agrobacterium tumefaciens and its complex with true substrate D-fructose[J].Febs Journal,2007,274(1):270-270.
[15]Yoshida H,Yamada M,Nishitani T,et al.Purification,crystallization and preliminary X-ray diffraction studies of D-tagatose 3-epimerase from Pseudomonas cichorii[J].Acta Crystallographic Section F,Structural Biology and Crystallization Communication,2007,63(2):123-125.
[16]Izumori K,Suga A,Takada G,et al.Izumoring:a strategy for bioproduction of all hexoses[J].Journal of Biotechnology,2006,124(4):717-722.
[17]Kim,HJ,Oh DK,et al.Conversion shift of D-fructose to D-psicose for enzyme-catalyzed epimerization by addition of borate[J].American Society for Microbiology,2008,74(10):3008-3013.
[18]Lim BC,Kim HJ,Oh DK,et al.A stable immobilized D-psicose 3-epimerase for the production of D-psicose in the presence of borate[J].Process Biochemistry,2009,44(8):822-828.
[19]Mu W,Chu F,XingQ,et al.Cloning,expression and characterization of a D-psicose 3-epimerase from Clostridium cellulolyticum H10[J].Journal of Agricultural and Food Chemistry,2011,59(14):7785-7792.
[20]Rao D,Yoshihara A,Gullapalli P,et al.Towards the biotechnological isomerization of branched sugars:D-tagatose-3-epimerase equilibrates both enantiomers of 4-C-methyl-ribulose with both enantiomers of 4-C-methyl-xylulose[J].Tetrahedron Letters,2008,49(20):3316-3321.

