面向药物研发的大规模数据语义整合与挖掘模式探索*
□ 钱庆 洪娜 李姣/中国医学科学院医学信息研究所 北京 100020
面向药物研发的大规模数据语义整合与挖掘模式探索*
□ 钱庆 洪娜 李姣/中国医学科学院医学信息研究所 北京 100020
整合并在语义层面上充分互连药物研发数据,将有利于从全局、系统化的视角开展药物研发工作,同时也有助于预测药物的不良副作用、加快药物研发流程、缩减药物开发成本等。文章试图探索语义技术如何支持药物研发数据的整合和挖掘,通过基于知识组织体系的语义标注,以及多类型实体互连策略构建充分互连的药物关联数据,支持药物研究人员对这些大量复杂实体及其关系的查找、探索和知识发现,从而帮助药物研发人员和临床工作者更好地利用大规模药物数据,解决药物研发面临的实际问题。
语义整合,实体互连,RDF,可视化,药物研发
1 引言
药物研发过程是一个从新化合物的发现到药物成功上市的过程,是一项系统的技术创新工程。随着化合物研发、基因工程、临床试验等领域的快速发展,药物研发领域数据的规模不断增长,创新的实验技术产生了大量的数据,包括药物、化合物、蛋白质靶标、基因、生物代谢通路、细胞、疾病和副作用等。然而不同的实验场合产生不同的数据,数据之间相互孤立,无序地分散在公共领域和企业私有领域。例如PubChem Bioassay和ChEMBL是存储化合物如何作用于蛋白质靶标的数据集,UniProt是存储功能和生物学基因通路及蛋白质靶标的数据集,其他诸如存储药物副作用、基因表达、临床表现等数据的数据库。
现代药物研发需要建立在对多种复杂问题的理解之上进行研究,包含对化学基因组学、化合物和药物与人体中的蛋白质和基因之间的复杂相互作用等,需要从全局、微观、分面、多层次上揭示药物研发知识内容及其间的关系,帮助科研人员、实验室工作者和临床工作者更好地、系统化地理解药物研发中的复杂关系。
2 相关研究
药物研发领域的数据有结构化形式的数据库,也有非结构化的文本。数据库资源有ChEMBL、DrugBank、OMIM等,文本型资源有PubMed、ClinicalTrials.gov等,知识组织与概念框架资源有GeneOntology、UMLS、ProteinOntology等。除了大量的数据资源,研究者也从事了大量的系统和工具研发,主要有两大阵营,分别是从数据整合角度和文献挖掘角度设计的系统和工具。数据整合角度的工具有GWAS、BioDW、SAM、ArrayMiner、GenePattern、EnsMart等,文献挖掘角度开发的典型工具有FACTA、BITOLA、Textpresso、Geneways、Pharmspresso、iHOP等。近年来,随着语义Web技术的发展,药物研发数据整合产生了一个重要分支,以语义Web和关联数据为核心技术,开展药物研发应用方面的探索,支持不同的应用,代表性系统有Bio2RDF、LODD、Chem2bio2RDF和OpenPHACTS。可见,利用语义Web,特别是关联数据方法整合结构化数据库中的实体和关系,在药物研发领域是一种新思路。本文重点介绍以下四个新型数据整合系统。
(1)HCLSIG及其LODD项目。HCLSIG[1]是一个开放组织,致力于解决多种问题,包括用于捕捉生物医学科学结论,集成生命科学和临床数据,为蛋白质组学和基因组学数据的发布提供指导等。LODD[2](Linked Open Drug Data)是HCLSIG支持的项目之一,LODD是一个用于药物发现的关联数据,该项目集成了来自多方面的药物信息,从药物对基因表达的影响到对临床试验结果的影响,包括大量的药物、临床、疾病以及制药企业相关的数据集。通过整合LODD、临床试验和病人数据,同时采用TMO[3]作为概念框架,J.S. Luciano等研究者建立了一个基于语义化集成知识库,通过共同的靶标调制深入理解药物再利用,通过作用机制的比较为疾病推荐可替代的治疗方案。
(2)Chem2Bio2RDF。印第安纳大学的Chem2-Bio2RDF[4]项目开创性地证明了大规模语义集成应用于药物研发的可行性。Chem2Bio2RDF集成了大范围药物研发相关的数据集,将其整合为统一的三元组格式,然后采用化学基因组本体Chem2Bio2OWL进行标注。通过将生物信息学和化学信息学的功能嵌入到SPARQL查询中,研究者可以解决简单的集成发现问题,从多来源数据集中发现新的和有意义的关系,并开展多个验证实例,证实了Chem2Bio2RDF利用大规模Linked Data帮助研究者进行药物研发。
(3)OpenPHACTS。OpenPHACTS项目[5]是欧盟第七框架下的创新医学联合计划项目(Innovative Medicines Initiative Joint Undertaking),它很好地实践了语义仓储的理念,真正形成了开放、集成的药物研发语义环境,基于关联数据的技术方法,OpenPHACTS构建了一个开放的药理学空间,OpenPHACTS项目最大的成就在于它密切结合了药物研发过程中的具体研究问题,这些问题是由涉及研究机构、制药企业、临床等药物研发相关机构共同提出的,OpenPHACTS以大规模的数据为背景,应用语义技术和工作流技术,实现对药物研发的全面支持。
(4)Bio2RDF及其在HIV药物研发中的应用。Bio2RDF是一个大规模、分布式生物医学知识库,集成了40多种生物医学信息资源,共包含大约50亿个三元组,Nolin MA等研究者基于Bio2RDF开发了一个针对艾滋病的整合生物资源项目,该项目目的是从显著表达的基因中识别蛋白质相互作用网络,通过开展分子和疾病的关联分析,更好地理解艾滋病药物研究过程中,靶标偏离导致的药物副作用。
这些相关研究对本文的研究思路、技术路线提出了有益的借鉴。此外,许多新兴的科学领域也在探索数据整合分析的可能性,例如系统生物学和系统化学生物学的兴起与发展。可见,将新技术不断深入地应用于药物研发的复杂内容管理是一个值得深入探索的研究领域,结合新兴的语义技术,可以有效推进该领域的研究进程。然而通过对上述研究思路的分析,我们认为当前在药物研发领域,数据整合与利用研究仍有一些问题需要进一步探索:
(1)缺乏统一的药物研发知识组织体系来规范术语的表达。即便是采用本体或词表来辅助数据的组织和规范化,特定用途的本体或词表仍难以涵盖药物研发流程中所有环节的数据内容,这为发现相同实体、相关实体,建立数据之间的语义关系带来许多的障碍。
(2)在深入的数据组织和多层次多类型的语义关联方面研究仍显不足。目前的关系构建主要基于“等同”和“相关”关系的识别,从实体自身的语义关联性考虑,实体映射涉及的关系类型不应当仅仅是实体间的“等同”和“相关”关系,还应涉及其他类型的语义关系,目前此类研究仍然较少。
(3)单一技术角度的药物数据分析是不够的,需要寻求一个多角度的平衡。结构化数据库存在着数据陈旧、更新不及时等问题,新知识和新关联难以被发现。文献挖掘又存在着准确率、可靠性的问题。因此,需要结合数据库资源和科学文献各自的优势,整合最新的研究数据,甚至是研究观点、科学结论,发掘更为广泛、及时地数据互连的可能性。
3 “从数据到语义”推进药物研发的进程
药物研发不同环节持续地产生大规模的不同形式的数据,共同推进药物研发的进程,同时也为药物研发提出新的挑战。语义技术为药物研发提供了一种大数据环境下的解决方案,是实现数据整合与利用的重要支撑技术。
3.1 数据密集时代药物研发的新挑战
大数据环境下的数据存储、分析都面临着新的挑战。药物的安全性、疗效提高与新型药物基因组学研究也都需要建立在大数据集成分析的基础上,需要新的解决思路。总体来看,目前药物研发面临以下三大挑战:
(1)药物研发各环节产生的数据面临大规模增长
药物研发不同的环节持续地产生大规模的不同形式的数据。从典型的现代药物研发流程来看,其包含多个环节,首先将药物物质、基因组数据和蛋白质靶标送入高通量芯片筛选,然后生成先导化合物,以及送入人体开展临床试验,最后形成真正的临床用药。不同环节的数据包含疾病、药物、基因、SNP、蛋白质、小分子、通路、化合物等实体,这些数据为多年的研究所积累,大量的直接和潜在关系蕴藏在这些大规模的数据中,等待着被挖掘,这些联系可能是基因-疾病关系、基因-药物关系、疾病-药物关系、药物-副作用关系、基因调控作用、蛋白质-蛋白质交互作用、蛋白质-小分子相互作用、基因-基因关系、蛋白质-通路关系、化合物-药物关系等,发现这些分散数据之间的联系,有助于解决药物研发中的具体问题。
(2)药物安全有效的转化研究
从药物研发的现状可以看出,缩减开发周期,降低从实验室到临床用药的转化成本,提升药物从基础研究发展成为临床用药的成功几率,仍然是药物研发面临的问题和挑战。当前药物的失败则主要在毒理学和临床安全问题上,即便是靶向性的药物研发方法也没有缓解这个问题,靶向性药物研究者只关注化合物如何作用于特定蛋白质靶标上,并没有考虑化合物在身体中的广泛作用,对意想不到的其他靶标的连锁效应不能很好地预测,这也导致很多药物在临床试验阶段失败了。因此,药物发现仍然面临着“从实验室到临床”安全有效转化的挑战[6]。
(3)生物标记、药物基因组学研究与个性化医疗
药物研发面临着前所未有的挑战,融合新的数据和技术,构建一个合理的现代药物研发发展模式的需求非常强烈。如果能够利用现代技术预测分子在人体试验中的反应,这将会对减少临床试验的时间和成本有很大帮助。尽管预测药物在人体中的临床疗效是个复杂的挑战,生物标记、药物基因组学研究则为此提供了可能[7-10]。寻找有效的方法将基因组数据应用到药物研发过程中,选择和确认新型药物靶标,并遵循以病人为中心的药物研发理念,针对特定的病人开发特定的药物,这是当前药物研发面临的新挑战[11]。
3.2 语义技术助力药物研发
传统的数据集成面临的主要困难包括数据精简和数据标准化两大问题。太过复杂的数据往往是分析工作的负担,这是因为不同的数据集往往从不同角度的多个维度来表达数据。数据标准不一致的问题更为棘手,例如,对于相同的血液蛋白,不同的临床试验机构、实验室采取不同的操作规范和测度结果使用单位。现代药物研发需要寻求一种有效的解决方案来解决如此复杂的集成问题。语义技术则是解决大规模药物研发数据整合与利用的新兴支撑技术,将不同层面的数据按照语义内容有序的组织起来,充分互连、深度挖掘以支持特定药物研发任务的分析挖掘,这对药物研发和药物转化研究有重要的推进作用。关联数据则是实现此目的的一个可行途径,这体现在:
(1)支持多类型、多角度、多层面数据整合。将不同层面的数据按照语义内容有序地组织起来,这对药物研发和药物转化研究有重要的支持作用。通过数据驱动的方式全面促进药物研究从实验室到临床的转化,需要协调运用多学科知识实现内容的整合。
(2)深度知识组织,构建充分互联的语义关系。药物研发相关内容的组织与关联,是提供高质量数据服务与知识发现的前提。知识组织体系的支撑是数据标准化的重要渠道,为数据的整合提供底层语义框架,是建立数据间丰富语义关联的主要途径。
(3)支持开放与共享。语义Web的初衷就是将不同数据源互联起来,构建一张计算机能理解的Data Web,提供丰富的语义关联,支持数据的开放与共享。正如关联数据的原则之一:使用HTTP URI使任何人都可以访问这些数据。
(4)支持数据动态更新。关联数据的存储形式是三元组,而三元组的存储形式相比关系数据库的存储更加便于数据的更新和维护,当有新的数据更新包到来时,只需将新数据转化为三元组,并建立与现有数据的映射,动态导入系统中,这个过程不涉及关系数据库更新所遇到的多表更新不同步、关联表不一致等问题。
4 面向药物研发的大规模数据语义整合与挖掘
在对药物研发领域数据库和文献库的内容、组织结构、获取方式分析的基础上,我们开展了数据RDF化,实体映射、关系抽取、本体映射等理论、方法和系统的调研分析,进而完成了面向药物研发的大规模数据语义整合与挖掘系统的框架设计、方法流程设计以及核心功能的实施。
4.1 总体框架
面向药物研发的大规模数据语义整合与挖掘研究的总体思路是:面向药物研发的具体应用,以解决实际问题为出发点,以开放数据库资源整合为主,以文献中挖掘出来的关系为重要补充,以统一语义框架作为规范和支撑,构建集成药物关联数据网络,通过一系列数据挖掘和图挖掘方法,开发不同应用的不同计算策略,最终支持多种药物研发应用,如图1所示。
药物研发数据网络的构建需要三个关键层次的数据网络支撑,首先构建一个药物研发领域的底层统一语义框架,集成药物研发领域内权威的各级语义知识框架,为后续的知识整合、表示与发现提供互操作和转换的桥梁。其次,在数据整合层面又涉及两个层面的网络,分别是来自结构化数据库的基础网络和来自文献的扩展网络:第一,提取多来源异构数据库中的疾病、基因、SNP、蛋白质、药物等结构化实体及关系,建立跨数据集实体之间的直接映射关系,构建药物基础网络;第二,从非结构化文献、Web中抽取药物相关实体及关系,充分利用结构化数据库中的规范名称作为基础语料,同时采用规则模板和机器学习等方法,从非结构化文本中抽取富语义关系,构建药物扩展网络。最后实现三层网络的融合,构建药物研发数据网络。
4.2 方法流程与核心问题
面向药物研发的大规模数据语义整合与挖掘涉及一系列方法和技术,从异构数据资源的采集、格式转换、预处理,复杂的关系映射方法与算法实现,知识组织体系的支撑,以及文献中关系的发现和补充,直到最后的药物研发应用,是一个完整的处理流程,采用一系列具体的技术方法。
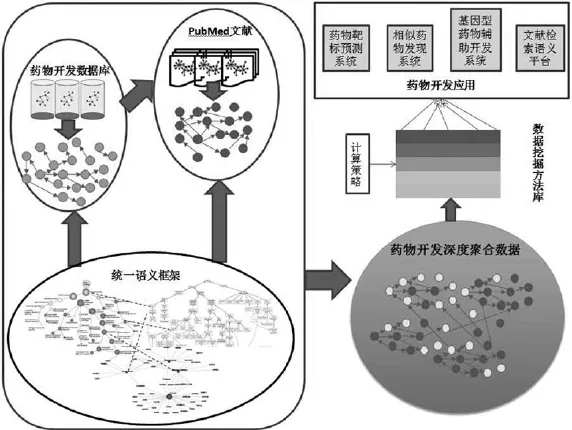
图1 面向药物研发的大规模数据语义整合与挖掘框架
图2揭示了大规模药物数据语义整合与挖掘的方法流程与实施路线,其中,数据的关联策略和算法是整个流程的核心问题,是构建面向药物研发应用的前提和关键,实体映射的类型不是单一的“等同”匹配,而应当是多类型的,涉及的多种类型语义关系有相似关系、上下位关系、包含关系、不相交关系及其他更为具体的语义关系等。
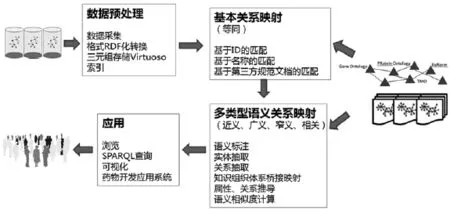
图2 大规模药物数据语义整合与挖掘的方法流程与实施路线
(1)“等同”关系识别与映射
不同来源的药物数据往往存在大小写、单复数、缩写、同义词等不一致的表达形式,“等同”关系识别与映射要解决的问题是识别不一致形式而本质相同的实体。在“等同”关系的识别过程中,我们探索了两种实现方案:①基于ID的映射,一些权威机构发布自己的不同数据集时,通常会采用统一的ID表达某一领域的特定数据,如NCBI发布的基因ID都是相同的,这些ID可以直接建立数据集之间的一部分关联;②数据的发布者往往会同时发布与其他权威数据集ID之间的映射文档,这些规范为ID映射提供了大量的支撑;③基于实体名称的映射,通过语义相似度计算,直接识别药物实体名称之间的语义关联,分别采用基于字符串匹配的方法、基于词法分析的方法、基于规则的方法、基于机器学习的方法等,从字形、语法、语义三个层面来识别实体名称之间的关系。
(2)多类型语义关系识别与映射
从语义上的多类型映射角度入手,本文也探索多类型语义关系的识别与映射方法。相似关系的计算方法类似于等同关系的计算,借助于知识组织体系中的入口词属性来识别,同样需要语义消歧步骤来判断识别出来的关系是否能作为“相似”关系纳入到关联数据网络中。广义关系与狭义关系识别方法的主要思路是,建立在等同关系识别的基础上,然后继承来自知识组织体系内部的广义关系和狭义关系。相关关系的识别则比较特殊,因为作为相关关系的实例又可能是多种多样的,如“药物-靶标”关系、“组成成分”关系、“基因变异位点”关系等,这样的关系非常具体,每一种关系都构成了三元组结构中独立的谓词(Predicate)。它的识别方法也相对复杂,我们采用的主要思路是,除了继承来自知识组织体系内部的相关关系,还借助外部资源PubMed发现两个实体的共现关系以及其他语义相关关系。
4.3 系统实施
(1)数据的选取、采集、RDF化与存储
选取、采集、RDF化和存储均属于数据预处理的工作,是后续研究与应用的前提和基础。选取和采集的资源有:①来自现有开放关联数据的药物研发相关资源,如LODD、OpenPHACTS等;②部分药物研发领域的开放数据库,如DrugBank、EntrezGene、OMIM、PDB、KEGG、DrugBank、dbSNP等;③文献库PubMed药物研发文摘信息。选取和采集之后,分析发现来自不同数据集的数据,具有不同的原始格式,包括excel、fasta等,需要将它们统一转换成RDF三元组进行格式的统一。我们并没有全部转换所有的数据集,尽可能地复用现有的关联数据,例如hgnc、diseasome就来自chem2bio2rdf,但是对于一些特定需求的专业数据集,如dbsnp,我们采取了自行转换的方式转换成三元组。最终所有的数据都以统一的n3格式输入到三元组仓储Virtuoso中。经过采集、转化和统一存储后,支持药物研发的关联数据的数据规模为10个数据集,以22089203多万条三元组的形式存储,如表1所示。
(2)关系构建情况
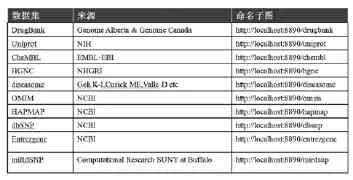
表1 数据选取与采集
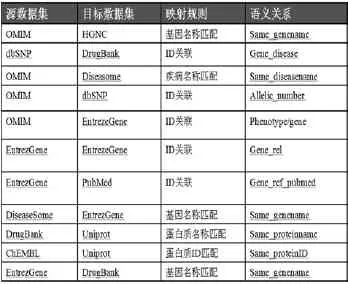
表2 数据映射情况
系统还构建了映射关系12528322条,建立的数据集之间的映射关系,如表2所示。
(3)语义数据的浏览、查询与可视化
我们开发了药物关联数据平台,实现了药物研发领域的大量复杂实体及其关系的浏览、SPARQL查询、开放接口及可视化。对于检索功能,平台支持自动提示,便于用户选择可能的输入词,同时,平台还提供SPARQL查询功能,用户可以通过界面输入查询和SPARQL endpoint接口两种方式调用。基于药物关联数据平台,系统支持多种类型的语义关系查询,进而实现基本的知识发现,输入任何一个疾病,可以发现和它相关的基因,或者蛋白、SNP、药物等。通过浏览和初步查看,如果用户对其中的一个基因感兴趣,希望看到老年痴呆症和该基因具体是如何联系起来的,可以进一步查看具体路径上的节点a,查看具体关系。对于具体关系,平台支持具体路径文字描述(图3)和可视化(图4)两种方式显示,同时还支持从不同角度对路径的排序,例如路径长度、路径加权长度和节点的重要度等。这样做最终是为了帮助用户快速找到有价值的潜在关系。
5 未来应用与展望
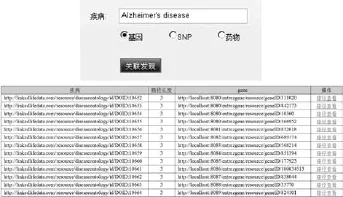
图3 实体间路径及其排序
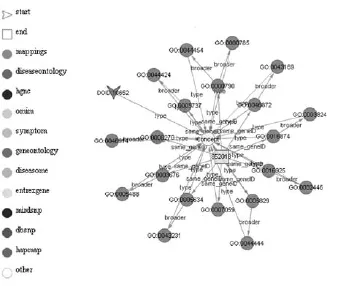
图4 实体关系可视化
基于语义技术的数据整合对药物研发有重要推动作用,随着资源之间的关联不断增多和完善,我们可以构建许多有意义的应用。因此,我们认为,基于语义技术探索药物研发的数据整合,建立和挖掘深层次多角度的药物关系,支持药物的转化研究,这是语义技术发展以来促进药物研发与转化的一种新模式。获取、组织和集成这些来自药物研发流程中不同环节的分布式、异构来源的数据,并对其在语义层次上深度关联、整合和挖掘,有助于解决药物不良副作用的预测、药物研发成本的缩减等药物研发中的具体问题。一个深度组织、互联互通、可智能化推理与预测的药物研发整合数据网络在转化医学的发展过程中将具有重要的作用,将有利于推进药物研发从临床前研究到临床实践的快速转化。由此,我们期望能够进一步解决药物研发中的具体问题,最终能够辅助预测药物副作用,提升药物安全性,促进药物再利用,降低药物研发的成本,辅助药物基因组学新型药物研发,以及促进药物研发的转化研究。
[1] RUTTENBERG A, CLARK T, BUG W, et al. Advancing translational research with the Semantic Web [J]. BMC bioinformatics, 2007, 8(Suppl 3): S2.
[2] Linked Open Drug Data [EB/OL]. [2013-12-21]. http://www.w3.org/ wiki/HCLSIG/LODD.
[3] LUCIANO J S, ANDERSSON B, BATCHELOR C, et al. The Translational Medicine Ontology and Knowledge Base: driving personalized medicine by bridging the gap between bench and bedside [J]. J Biomed Semantics, 2011, 2(Suppl 2): S1.
[4] DONG X, DING Y, WANG H, et al. Chem2Bio2RDF Dashboard: Ranking semantic associations in systems chemical biology space [J]. Future of the Web in Collaboratice Science (FWCS), WWW, 2010.
[5] HARLAND L. Open phacts: A semantic knowledge infrastructure for public and commercial drug discovery research [M]//KnowledgeEngineering and Knowledge Management. Springer Berlin Heidelberg, 2012: 1-7.
[6] VAN Q N, VEENSTRA T D. How close is the bench to the bedside? Metabolic profiling in cancer research [J]. Genome Med, 2009, 1(1): 5.
[7] AUFFRAY C, CHEN Z, HOOD L. Systems medicine: the future of medical genomics and healthcare [J]. Genome Med, 2009, 1(1): 2.
[8] CHEN J H, NI R Z, XIAO M B, et al. Comparative proteomic analysis of differentially expressed proteins in human pancreatic cancer tissue [J]. HepatobiliaryPancreat Dis Int, 2009, 8(2): 193-200.
[9] GREINERT R. Skin cancer: new markers for better prevention [J]. Pathobiology, 2009, 76(2): 64-81.
[10] STIMSON L, LA THANGUE N B. Biomarkers for predicting clinical responses to HDAC inhibitors [J]. Cancer letters, 2009, 280(2): 177-183.
[11] MERVILLE S. Challenges and opportunities in drug discovery and development [EB/OL]. [2013-12-21]. http://www2.mdanderson. org/cancerfrontline/2013/04/challenges-and-opportunities-in-drugdiscovery-and-development.html.
Research on Big Data Semantic Integration and Mining Pattern for Drug Discovery
Qian Qing, Hong Na, Li Jiao/Institute of Medical Information of Chinese Academy of Medical Sciences, Beijing, 100020
Integrating and linking drug discovery data in semantic level is helpful to make drug discovery research implement on global, systematic, and predicable perspectives, also it will accelerate the research flow and reduce the cost of development. In this paper, we explore how semantic technology supports the integrating and mining of drug data, through semantic annotation based on knowledge organization system and multi-type entity linking strategy to construct interlinked data for drug discovery. Our research supports the search, exploration and mining of large scale entities and complex entity relations. The object of our effort is to make the most of drug big data for supporting researchers and clinicians to resolve the real problems existing in drug discovery.
Semantic integration, Entity linking, RDF, Visualization, Drug discovery
2014-02-14)
10.3772/j.issn.1673—2286.2014.03.003
*本文系国家“十二五”科技支撑计划项目课题“科技知识组织体系的协同工作系统和辅助工具开发”(编号:2011BAHl0B02)和国家社会科学基金项目“关联数据中潜在知识关联的发现方法研究”(编号:11CTQ016)的研究成果之一。
钱庆(1970- ),中国医学科学院医学信息研究所研究员,研究方向:知识组织、知识发现、大数据。
洪娜(1980- ),中国医学科学院医学信息研究所副研究员,研究方向:语义Web、关联数据、大数据。E-mail: hong.na@imicams.ac.cn
李姣(1981- ),中国医学科学院医学信息研究所副研究员,研究方向:数据挖掘、文本挖掘、科学数据。

