基于DNA条形码鉴定豆蔻类中药材△
王晓玥,陈晓辰,廖保生,王丽丽,韩建萍
(中国医学科学院 北京协和医学院 药用植物研究所,北京 100193)
基于DNA条形码鉴定豆蔻类中药材△
王晓玥,陈晓辰,廖保生,王丽丽,韩建萍*
(中国医学科学院 北京协和医学院 药用植物研究所,北京 100193)
目的:利用ITS2序列对豆蔻类中药材进行DNA条形码鉴定研究。方法:本文采集了包括豆蔻两个基原白豆蔻Amomumkravanh与爪哇白豆蔻Amomumcompactum、红豆蔻Alpiniagalanga、草豆蔻Alpiniakatsumadai等在内的70份样品,采用试剂盒法提取基因组DNA,应用Primer premier 5.0设计引物,通过聚合酶链式反应(Polymerase Chain Reaction,PCR)扩增其ITS2序列,并对PCR产物进行双向测序,所得序列经CodonCode Aligner V3.71拼接后,采用MEGA5.0 软件进行序列比对,计算种内和种间距离,构建邻接树(Neighbor-joining tree,NJ Tree)。ITS2序列采用基于隐马尔可夫模型(Hidden Markov model,HMM)的注释方法获得。结果:通过新设计引物扩增的序列可以注释到5.8 S与28 S,扩增序列位于ITS2间隔区。采用新设计的引物,所有实验样品均可获得ITS2序列,NJ树结果显示不同种豆蔻类药材分别聚为一支,可以互相区分,且可以与其混伪品相互区分。另外,通过序列比对发现有5个SNP(s)存在于豆蔻与其它物种种间,且有2个稳定的SNP(s)存在于白豆蔻和爪哇白豆蔻种间,可用于二者的准确鉴定。结论:ITS2序列作为DNA条形码可准确稳定地鉴别不同种豆蔻类药材,本研究将为保障临床安全用药提供新的技术手段。
豆蔻;DNA条形码;ITS2;物种鉴定
豆蔻之名始载于晋代药学著作《名医别录》中[1],同时其名也常出现在古代文学作品中,如唐代诗人杜牧有诗《赠别》云:“娉娉婷婷十三余,豆蔻梢头二月初。”说明豆蔻类中药材在我国得到认识已有很久的历史。豆蔻在不同历史时代中代表不同来源中药材,各种豆蔻类药材的名称或外形类似,在本草记载中,同物异名或同名异物等情况常有出现,民间用药混乱。现常用品种为姜科豆蔻属及山姜属植物,如(白)豆蔻、草豆蔻、红豆蔻等。由于姜科物种本身不易辨别,姜科豆蔻属和山姜属内其他物种如草果、益智、砂仁等[1]较豆蔻类药材不易区分,常成为此类药材的混用品。如高良姜与红豆蔻原植物外形相似,但是红豆蔻所含挥发油较少[2-6],香气较淡[7];草果与草豆蔻之间也时常存在混伪现象[8]等。本次实验所选择的十种中药材均被中国药典(2010版)所收录,如豆蔻为白豆蔻AmomumkravanhPierre ex Gagnep.或爪哇白豆蔻AmomumcompactumSoland ex Maton的干燥成熟果实[9],红豆蔻为大高良姜AlpiniagalangaWilld.的干燥成熟果实[10],高良姜为高良姜AlpiniaofficinarumHance的干燥根茎[11]等。各药材之间药效差异较大,如草豆蔻具有行气作用,草果却没有此作用;草果功效截疟,可用以寒湿偏盛之疟疾,而草豆蔻则无此作用[7]。
豆蔻类药材采收年限较长,如高良姜栽培一般4年收获,种后5~6年收获的,根茎含粉质多,质量更好,产量也高[12];益智定植第3年开始开花结果,第5年才进入丰产期[13],故老百姓种植积极性不高,药材市场紧缺。加之其在市场上混伪现象严重,故鉴定区分豆蔻类药材具有重要性及必要性。为了解决几种豆蔻类药材的混伪情况,许多学者已在形态分类、化学成分研究方面做了许多研究。如豆蔻的两种基原植物白豆蔻与爪哇白豆蔻,已有在显微水平上对其鉴定区分的研究[14],但是这些技术要求鉴定者具有丰富的解剖学、组织学或化学知识,对于没有丰富经验的人员而言,鉴定较困难。
DNA条形码(DNA barcoding)技术作为一种新兴的分子鉴定手段,即利用基因组中一段公认的、相对较短的DNA序列来进行物种鉴定的一种分子生物学技术,是传统形态鉴别方法的有效补充,近年来成为国内外生物分类和鉴定的研究热点[15],在Nature,Science等学术刊物和National Geographic News、The New York Times等媒体上大量报道[16],该技术具有便捷、快速、准确等特点,在药用植物的鉴定和濒危物种的保护中具有广阔的应用前景[17-18]。该种方法的出现,极大地提高了物种鉴定效率及其准确性,与传统方法相比,在通用性与可数字化方面具有明显优势[19]。韩建萍等[20]发现,ITS2序列可以作为洋金花及其伪品鉴定用的条形码序列;朱英杰等[21]对重楼属11个物种的ITS2,psbA-trnH,rpoB,rbcL,matK等序列进行分析,结果表明ITS2序列鉴定效率为100%。Chen等[22]通过对大量研究的系统整理提出,ITS2序列可以作为标准DNA条形码鉴别药用植物,并在通过对大样本量药用植物研究的基础上,建立了以ITS2序列为主,psbA-trnH序列为辅的中药材DNA条形码分子鉴定体系[23-24],Yao等[25]也通过大样本量分析证实ITS2序列可以作为植物物种鉴别的通用条形码。
本研究主要从分子水平着手,通过DNA条形码标准化流程,对豆蔻类药材进行鉴定,并且建立该类药材的鉴定方法体系,以满足非形态学或分子生物学专业人员对豆蔻类药材进行鉴定的需求,将为确保药材的质量及临床用药安全提供参考。
1 材料与方法
1.1 材料
以姜科豆蔻属与山姜属10个物种共125个样本为研究对象,实验样品植物材料来源见表1,GenBank登录号等信息详见表2。其中70份实验材料样品来自全国各大药市、药店,如安徽亳州药材市场、河北安国药材市场、北京各大药店、四川成都荷花池药市等;原植物样本采自云南西双版纳热带植物园、华南植物园和中国医学科学院药用植物研究所海南分所及云南分所、广州中医药大学等地,凭证标本存放于中国医学科学院药用植物研究所标本馆。其余55个样品来源于GenBank。
1.2 样品DNA提取、PCR扩增与测序
1.2.1 DNA提取 取种子或果实类样品约50 mg,叶片类样品20 mg;根及根茎类样品取样前,用酒精擦拭表面,切去样品表皮,取内部样约35 mg。用DNA提取研磨仪研磨2 min(30次/s)。采用植物基因组DNA提取试剂盒(Tiangen Biotech Co.,China)提取总DNA。

表1 实验样品信息表

表2 GenBank数据库获得豆蔻类药材及混伪品ITS2序列GenBank登录号信息
1.2.2 PCR扩增 本研究初期,使用通用引物ITS 2F/3R[22]进行序列扩增,但是发现该引物在部分实验所用豆蔻类药材(爪哇白豆蔻、白豆蔻、草果、草豆蔻、红豆蔻、高良姜)序列扩增中效率较低,无法达到测序要求,因此我们针对豆蔻类药材设计特异性引物扩增ITS2区段。利用GenBank下载的同属物种ITS2序列两端的5.8 S和28 S保守序列,使用Primer premier 5.0软件设计引物,检查引物长度、引物二级结构(包括引物自身二聚体、发卡结构、引物间二聚体等)、引物GC含量等是否符合要求,最终设计引物为ITSHF(20 bp)/ITSHR(20 bp),引物交由生工生物工程(上海)股份有限公司北京合成部合成(具体引物信息及扩增程序见表3)。PCR反应体积为25 μL,体系内含MgCl22 μL(25 mM)、dNTP 2 μL(2.5 mmol·L-1)、PCR buffer 2.5 μL(10×)、引物各1.0 μL(2.5 μmol·L-1)、Taq DNA聚合酶1.0 U、总DNA约2 μL。通过优化其PCR扩增程序,实验结果证明该引物对豆蔻类中药材的PCR扩增效果优于通用引物ITS 2F/3R。PCR扩增产物经纯化后,使用ABI 3730XL测序仪双向测序。

表3 PCR引物及扩增程序
1.2.3 序列拼接和数据分析方法 测序峰图釆用序列拼接软件CodonCode Aligner V3.71(CodonCode,Dedham,MA,USA)校对拼接,去除低质量序列及引物区。研究涉及的全部实验以及下载ITS2序列,采用基于隐马尔可夫模型的HMM注释方法去除两端5.8S和28S区段即可获得ITS2间隔区序列[26]。然后,将所有序列用软件MEGA5.0(molecular evolutionary genetics analysis)分析比对[27],用邻接(Neighbor-Joining,NJ)法构建系统聚类树,通过Bootstrap(1 000次重复)检验各分支的支持率,并基于K-2P遗传模型进行遗传距离等分析。
2 结果与分析
2.1 ITS2序列长度及GC含量比较
研究所用10个物种的ITS2片段序列的长度范围为218~309 bp,GC含量范围为55.23%~63.30%,豆蔻类药材及其混伪品之间在ITS2序列长度上存在差异,详见表4。
2.2 豆蔻类药材及其混伪品种间变异分析
利用MEGA 5.0输出的变异位点见图1(序列完全相同样品只列出一个作为代表),种间变异较丰富。应用MEGA5.0对种间和种内K-2P距离进行计算,由表5中K-2P遗传距离结果表明,爪哇白豆蔻与白豆蔻各自的种内遗传距离大于它们之间的种间遗传距离,表示两个物种亲缘关系较近,没有Barcoding-GAP;其余物种的种内遗传距离均小于其与其他物种种间遗传距离,即通过ITS2序列可以很好地鉴别实验物种。

表4 不同物种ITS2长度及GC含量

表5 实验用豆蔻类药材种内种间距离(注:最小距离~最大距离(平均距离))
分析亲缘关系较近、种间差异很小的物种,SNP位点解析是一个有力工具。所以对于利用遗传距离不易区分的爪哇白豆蔻与白豆蔻,可以采用SNP位点进行鉴别。对研究所涉及的10个物种的变异位点(图1)进行分析,结果标明豆蔻的两个基原可以与其它实验药材明显分开(表6)。豆蔻两个基原的ITS2序列长度均为218 bp,对其变异位点(图2)进行分析发现:61 bp处爪哇白豆蔻均为C,而白豆蔻均为T;202 bp处,爪哇白豆蔻均为T,而白豆蔻均为C。由此就可以用两个位点将白豆蔻与爪哇白豆蔻区分开。对GenBank中所示豆蔻的ITS2序列进行分析,结果表明这两个SNP(s)位点稳定地存在于两个基原物种间。综上所述,ITS2序列的SNP位点能明显的将两种豆蔻鉴别开。

图1 MEGA输出各物种单倍型及变异位点(Aligned By Muscle)

表6 豆蔻与其它实验药材SNP位点分析
2.3 豆蔻类药材及其混伪品的NJ树鉴定
基于ITS2序列构建豆蔻类药材及其混伪品NJ树(图3),结果表明本次实验研究的几种豆蔻类药材各自聚为一支,其中,爪哇白豆蔻与白豆蔻亲缘关系较近,在树的一支上,最终分为两小支,说明NJ树可以将爪哇白豆蔻与白豆蔻鉴别开,其它豆蔻类药材之间建树时区分支持率较高。同时豆蔻类药材均可与从GenBank下载的混伪品ITS2序列很明显区分开。因此,ITS2序列作为DNA条形码可准确鉴别不同种豆蔻类药材及其混伪品。

图2 MEGA输出爪哇白豆蔻与白豆蔻变异位点
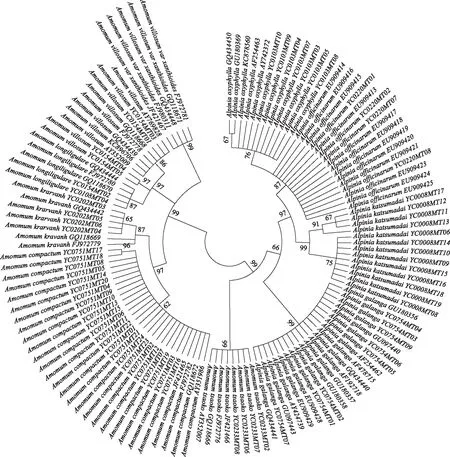
图3 基于ITS2序列构建的豆蔻类药材及其混伪品的邻接(NJ)树
3 讨论
3.1 DNA条形码鉴定准确性与稳定性分析
豆蔻类药材以果实或根茎入药,基原植物均来源于姜科,具有相近气味。有些物种之间形态类似,如白豆蔻与爪哇白豆蔻,显微鉴别时有微小区别;有些物种之间名称类似,如草豆蔻与草果故药用豆蔻类药材混淆现象严重。且豆蔻类药材常作为香料使用,有时要提取香精等,提取时已破坏其原植物或原药材性状特征,使鉴定更加困难,而从分子水平上鉴定不受其形态等影响。豆蔻类药材之间功效不尽相同,混用会造成用药不安全,故鉴别豆蔻类药材是非常迫切的。无论从所选的10个物种的豆蔻类药材种内、种间遗传距离,还是基于ITS2条形码序列构建的NJ树,均证实ITS2条形码序列能够准确地将豆蔻类药材鉴别开来。对于豆蔻的两个基原物种(白豆蔻与爪哇白豆蔻)而言,根据K-2P遗传距离分析,爪哇白豆蔻与白豆蔻各自的种内遗传距离大于它们之间的种间遗传距离,在NJ树上它们各自聚为一支,说明它们利用ITS2序列可以鉴别区分。同时用SNP位点也能很好将两个基原物种区分,说明ITS2条形码序列能够准确地鉴别豆蔻类药材。
所谓鉴定稳定性是指对于不同产地、不同批次的样品而言,通过实验均能够稳定地获得DNA条形码序列。本实验采用豆蔻类药材进行DNA提取,设计特异引物扩增ITS2 序列,70份药材和原植物样品均可成功获取ITS2序列,证明ITS2序列作为豆蔻类药材DNA条形码具有很好的稳定性。
3.2 DNA条形码在实际鉴定工作中的意义
豆蔻类药材是传统的常用中药材,但在不同历史时期或不同地区,对于豆蔻类中药材的认识不同,所使用的药材来源也不相同。如大高良姜(红豆蔻),古代所用红豆蔻为山姜属植物,但很难鉴定到种。现在民间,仍然有把果实红色的山姜属植物用作红豆蔻的情况。由此可见,豆蔻类药材在市场上流通的混伪品较常见,只有具有丰富的药材鉴别能力的人员才能准确区分开正品与混伪品。而DNA条形码优点在于,其在鉴定过程中不受药材性状的影响,是直接从基因水平上提供鉴定依据,因此,即便是没有丰富的药材鉴别经验的人员,通过DNA条形码的标准化流程,即可准确区分药材正伪品。因此可知,采用DNA条形码技术,可以更加准确、快速地完成中药材鉴定工作,对于今后药材市场中药材的流通、市场管理等实际应用均具有极大的价值。
[1] 吴孟华,郭平,陈虎彪,等.豆蔻类中药的本草新析[J].中国中药杂志,2012,37(11):1686-1692.
[2] 董乃维,刘凤芝,甘春丽,等.高良姜素提取工艺改进研究[J].哈尔滨医科大学学报,2006,40(2):168-169.
[3] Bleier W.,Chirikdjian J.J.Flavonoids from galanga rhizome(AlpiniaofficinarumHance)[J].Planta Med.1972,22(2):145-151.
[4] 罗辉,蔡春,张建和,等.高良姜根茎叶挥发油化学成分的比较[J].时珍国药研究,1997,8(4):34-35.
[5] Ly T N,Shimoyamada M,Kato K,et al.Isolation and characterization of some antioxidative compounds from the rhizomes of smaller galanga(AlpiniaofficinarumHance)[J].J Agric Food Chem,2003,51(17):4924-4929.
[6] 罗辉,蔡春,张建和,等.高良姜化学成分研究[J].中药材,1998,21(7):349-351.
[7] 赵中振,肖培根.当代药用植物典[M].上海:上海世界图书出版公司,2008:21-23.
[8] 石亚娜,金航,杨雁,等.草果药用本草考证[J].中国现代中药,2013,15(10):913-916.
[9] 国家药典委员会.中国药典[S].一部.北京:中国医药科技出版社,2010:156-157.
[10] 国家药典委员会.中国药典[S].一部.北京:中国医药科技出版社,2010:142-143.
[11] 国家药典委员会.中国药典[S].一部.北京:中国医药科技出版社,2010:270-271.
[12] 吕侠卿,吕祖建,陈宝树,等.中药培育大全[M].长沙:湖南科学技术出版社,2005:141.
[13] 吕侠卿,吕祖建,陈宝树,等.中药培育大全[M].长沙:湖南科学技术出版社,2005:176.
[14] 丁萍,刘心纯,徐鸿华.两种白豆蔻的显微鉴别[J].广州中医学院学报,1993,10(1):22-24.
[15] 陈士林,庞晓慧,姚辉,等.中药DNA条形码鉴定体系及研究方向[J].世界科学技术-中医药现代化,2011,13(5):747-754.
[16] 陈士林,宋经元,姚辉,等.药用植物DNA条形码鉴定策略及关键技术分析[J].中国天然药物,2009,7(5):322-327.
[17] 陈士林,谢彩香,姚辉,等.中药资源创新方法研究[J].世界科学技术-中医药现代化,2008,10(5):1-9.
[18] 陈士林,庞晓慧,罗焜,等.生物资源的DNA条形码技术[J].生命科学,2013,25(5):451-459.
[19] 陈士林,郭宝林,张贵君,等.中药鉴定学新技术新方法研究进展[J].中国中药杂志,2012,37(8):1043-1055.
[20] 韩建萍,李美妮,罗焜,等.DNA条形码鉴定洋金花及其伪品[J].药学学报,2011,46(11):1408-1412.
[21] 朱英杰,陈士林,姚辉,等.重楼属药用植物DNA条形码鉴定研究[J].药学学报,2010,45(3):376-382.
[22] Chen S L,Yao H,Han J P,et al.Validation of the ITS2 region as a novel DNA barcode for identifying medicinal plant species[J].PLoS ONE,2010,5(1):8613.
[23] Chen SL,Pang XH,Song JY,et al.A renaissance in herbal medicine identification:From morphology to DNA[J].Biotechnol Adv,2014,32(7):1237-1244.
[24] Yao H,Song JY,Liu C,et al.Use of ITS2 region as the universal DNA barcode for plants and animals[J].PLoS ONE,2010,5(10):13102.
[25] 陈士林,姚辉,韩建萍,等.中药材DNA条形码分子鉴定指导原则[J].中国中药杂志,2013,38(2):141-148.
[26] Keller A,Schleicher T,Schultz J,et al.5.8S-28S rRNA interaction and HMM-based ITS2 annotation[J].Gene,2009,430(1-2):50-57.
[27] Tamura K,Peterson D,Peterson N,et al.MEGA5:molecular evolutionary genetics analysis using maximum likelihood,evolutionary distance,and maximum parsimony methods[J].Mol Biol Evol,2011,28(10):2731-2739.
IdentificationofAmomiFructusRotundusBasedonDNABarcoding
WANG Xiaoyue,CHEN Xiaochen,LIAO Baosheng,WANG Lili,HAN Jianping*
(InstituteofMedicinalPlantDevelopment,ChineseAcademyofMedicalSciences,Beijing100193,China)
Objective:In this study,the internal transcribed spacer 2 of nuclear ribosomal DNA(ITS2)sequence was used for identifying the traditional Chinese medicine fromAmomumandAlpiniato ensure the quality medicines and clinical efficacy.Genomic DNA was extracted from 70 samples using Genomic DNA kit and used as templates for Polymerase Chain Reaction(PCR).Sequences assembly and consensus sequence generation were performed by Codoncode Aligner.The intraspecific and interspecific genetic distances were computed and the neighbor-joining tree was constructed by MEGA 5.0 in accordance with the kimura 2-parameter(K-2P)model.The ITS2 regions were obtained by using the hidden Markov model(HMM)-based annotation methods from the ITS2 sequence.Results:With the specific designed primers,ITS2 sequences can be obtained from all samples.Every species clustered into a clade in the neighbor-joining phylogenetic trees(NJ tree)respectively,and can be accurately separated with other varieties and adulterants.In addition,we can find some SNP(s)among Fructus Amomi Rotundus and other experiment species by comparing ITS2 sequences,andAmomumcompactumandAmomumkravanhcan be distinguished well through two stable SNPs.Conclusion:ITS2 region could be used as an efficient barcoder to identify the traditional Chinese medicine fromAmomumandAlpinia.This study could provide a new technique to ensure clinical safety in utilization Chinese medicines.
Amomi Fructus Rotundus;DNA barcoding;ITS2;species identification
10.13313/j.issn.1673-4890.2014.11.003
2014-10-05)
国家自然科学基金资助项目(81473303):太白贝母甾体生物碱生物合成途径的比较转录组学研究
*
韩建萍,副研究员,主要研究方向:中药资源及分子鉴定,Tel:(010)57833198,E-mail:jphan@implad.ac.cn

