厌氧氨氧化细菌物种组成对环境因子响应的研究进展*1
富冰冰,刘吉文,高铭鸿,张晓华
(中国海洋大学 海洋生命学院,山东 青岛 266003)
厌氧氨氧化细菌物种组成对环境因子响应的研究进展*1
富冰冰,刘吉文,高铭鸿,张晓华*
(中国海洋大学 海洋生命学院,山东 青岛 266003)

厌氧氨氧化细菌;物种组成;丰度;厌氧氨氧化活性;环境适应性
1 厌氧氨氧化细菌概述

厌氧氨氧化细菌属于浮霉菌门,具有由厌氧氨氧化体、核糖细胞质、外室细胞质组成的特殊细胞结构,针对其的研究正在深入。2014-01,Teeseling等首次描述了CandidatusKuenenia stuttgartiensis细胞最外层为蛋白质亚基晶体排列组成的蛋白质层,进一步完善了厌氧氨氧化细菌的细胞模型[3]。目前,厌氧氨氧化细菌共包括5个属13个种(表1),其中CandidatusScalindua属的CandidatusScalindua sorokinii为首次在自然环境中发现的厌氧氨氧化细菌种类,发现于黑海厌氧底层水中[4]。CandidatusScalindua zhenhei I、CandidatusScalindua zhenhei II及CandidatusScalindua zhenhei III为2011年命名的3个新种,发现于南海表层沉积物中[5]。
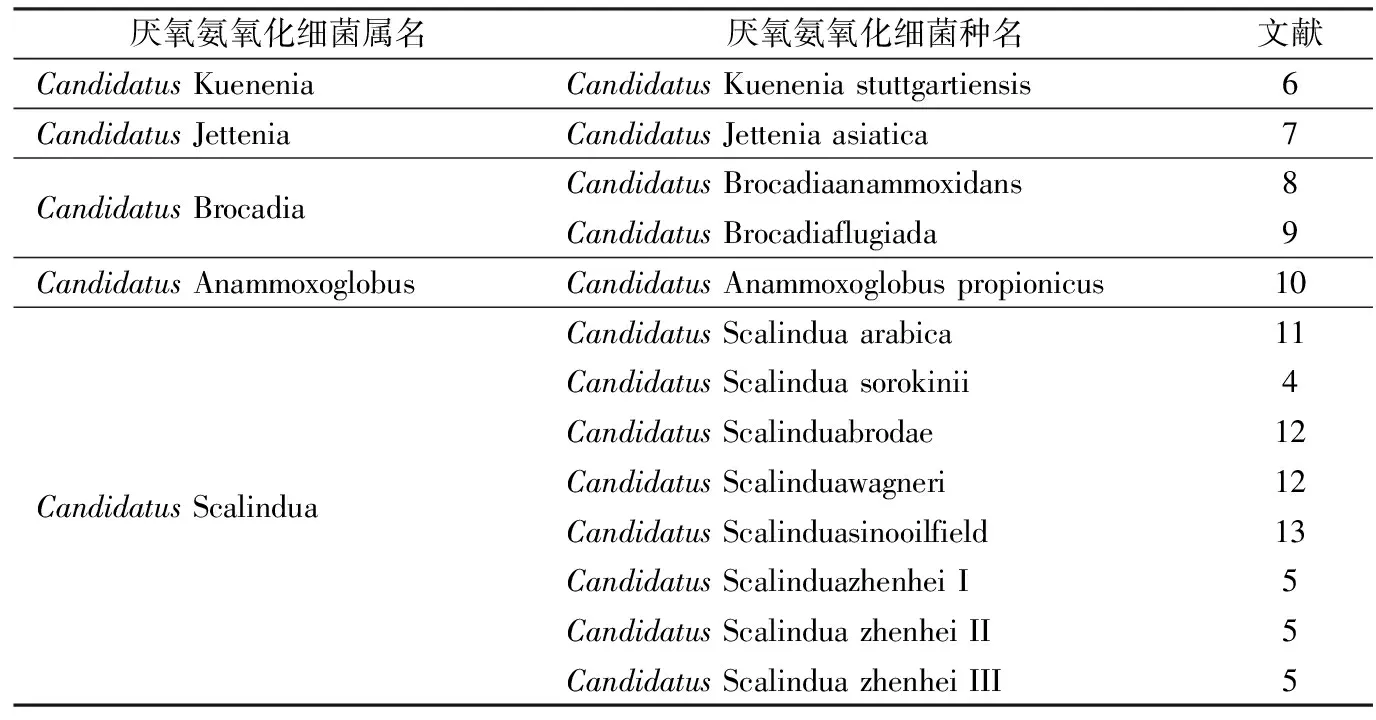
表1 厌氧氨氧化细菌种属Table 1 The genera and species of anammox bacteria
厌氧氨氧化细菌在海洋及多种生态环境中广泛存在,其物种组成、丰度、厌氧氨氧化活性以及三者对环境因子响应的研究,有助于全面认识厌氧氨氧化过程在海洋氮循环中发挥的作用。了解厌氧氨氧化细菌类群对不同环境因子的适应性,为其在污水处理等方面的实际应用提供了理论基础。现已发现厌氧氨氧化细菌物种组成、丰度及厌氧氨氧化活性受多种环境因子的影响。在不同环境中,发挥显著影响作用的环境因子不同;对于同一环境因子,不同研究者发现的结果存在差异。目前,对于厌氧氨氧化细菌在海洋及其它生态环境中的存在现状及其物种组成、丰度、活性与环境因子之间的关系缺乏系统的归纳总结及分析比较。
2 海洋厌氧氨氧化细菌环境适应性研究的技术基础
海洋中绝大多数微生物难以利用传统微生物培养方法进行纯培养,分子生物学技术的发展为更好地认识海洋微生物提供了基础。基因组学及生物信息学等分子生物学技术由于具有无需纯化培养、直接从环境中提取DNA等优点,避免了培养过程中细菌多样性严重损失的弊端,广泛应用于海洋微生物种群结构、丰度等方面的研究。目前,厌氧氨氧化细菌物种组成及丰度等方面的分子生物学技术主要包括针对16S rRNA,hzo,nirS,hzs和细胞色素C等基因的克隆文库构建[14-16]、新兴的第二代高通量测序以及宏基因组分析技术[12]、荧光定量PCR[17]及荧光原位杂交技术[4]等。扩增各基因所用的特异性PCR引物及特异性荧光定量PCR引物分别见表2和表3。

表2 扩增厌氧氨氧化细菌各基因的常用PCR引物Table 2 PCR primers for amplification of anammox bacterial genes

表3 扩增厌氧氨氧化细菌各基因的常用荧光定量PCR引物Table 3 Real time-PCR primers for amplification of anammox bacterial genes
3 海洋厌氧氨氧化细菌的环境适应性
厌氧氨氧化细菌在海洋及其他生态系统中广泛分布,但种类上却存在明显差异,如海洋环境中存在的厌氧氨氧化细菌属于CandidatusScalindua,而在废水处理厂活性淤泥或淡水环境中的厌氧氨氧化细菌多属于CandidatusBrocadia。环境因素是引起以上差异的主要原因。了解厌氧氨氧化细菌物种组成及分布与环境因子之间的关系,将为厌氧氨氧化细菌作为生物指示物用以评估环境特点及环境中氮污染程度这一科学构想提供理论基础。
3.1 盐 度
2003年Jetten等报道了CandidatusScalindua对盐度有极高的耐受力[21],随后的研究结果有力地证实了这一发现。在南太平洋东部的秘鲁上升流[20]、日本海[22]以及巴伦支海、黑海等11个不同海区(盐度为17~35)[23]均发现厌氧氨氧化细菌的存在,且只属于CandidatusScalindua。而CandidatusBrocadia被报道适于生活在淡水环境中[21]。2012年,王善雨等人在盐度为0.09~0.18的珠江口沉积物中观察到CandidatusBrocadia在淡水环境中占优势(占厌氧氨氧化细菌的53.6%)[24]。2013年侯立军等在盐度为0的2个长江口沉积物样品中观察到厌氧氨氧细菌全部属于CandidatusBrocadia[25]。另外,在盐度为1.0~18.0的长江口沉积物样品中观察到CandidatusKuenenia的存在[25],表明CandidatusKuenenia对寡盐(0.5~5)及中盐(5~18)环境有一定的适应性[18],虽然Kartal等曾推断CandidatusKuenenia是适于生活在淡水环境中的类群[26]。在凯普菲尔河口[18]及长江口[25]沉积物中观察到厌氧氨氧化细菌优势类群随盐度的升高从CandidatusBrocadia转变为CandidatusScalindua,这一分布模式清楚地展现了CandidatusBrocadia、CandidatusKuenenia及CandidatusScalindua对盐度变化的不同响应。相关性分析表明盐度是导致上述河口区厌氧氨氧化细菌物种组成差异分布的最主要因素[18,25]。目前,厌氧氨氧化细菌物种组成与盐度之间关系的研究已较为明确,CandidatusBrocadia、CandidatusKuenenia及CandidatusScalindua分别适应淡水、寡盐、海水环境的特性,有力地证实盐度是导致厌氧氨氧化细菌物种组成及分布差异的重要环境因子。
3.2 温 度
厌氧氨氧化细菌存在于一个广阔的温度范围。-2.5 ℃的北极海冰(固态)[27]是目前发现厌氧氨氧化细菌的最低温度环境,厌氧氨氧化细菌的细胞数约为1.1×105个/mL海冰。表面流体温度为100 ℃的深海热液喷口[28]是目前发现厌氧氨氧化细菌的最高温度环境,发现的厌氧氨氧化细菌属于CandidatusScalin-dua和CandidatusKuenenia。此外,在8~27 ℃的切萨皮克海湾[29]等多种适宜温度的海洋环境中均观察到厌氧氨氧化细菌的存在,但都属于CandidatusScalindua。厌氧氨氧化细菌对温度的广泛适应性在淡水及陆源环境中同样被证实,24~65 ℃的温泉[30]、55~75 ℃的石油储油池[13]、3.2~34 ℃的长江口[25]均观察到厌氧氨氧化细菌的存在。相关性分析表明,温度影响长江口厌氧氨氧化细菌丰度及活性,温度较高时丰度及活性较高。厌氧氨氧化细菌对温度的广泛适应性已证实,但其群落组成在不同温度的海洋环境中均以CandidatusScalindua占绝对优势。厌氧氨氧化细菌对温度的极限耐受范围;温度是否影响海洋厌氧氨氧化细菌群落组成;海洋厌氧氨氧化细菌丰度、活性与温度之间的关系,需在今后深入研究。
3.3 溶解氧
厌氧氨氧化细菌是一种专性厌氧菌[31]。迄今为止,对厌氧氨氧化细菌的研究几乎全部集中于低氧或厌氧环境。2014-03,Daigger等研究表明厌氧氨氧化过程须有少量氧(1.71 mg O2/mg氨转化为氮气)的参与[32]。另外,在未达到低氧标准(<3 mg/L)的秘鲁海水体中通过基因扩增及类脂分析技术,检测到厌氧氨氧化细菌的存在[20]。厌氧氨氧化细菌存在于有氧水体中这一现象,有2种可能原因:1)表明厌氧氨氧化细菌对氧气有一定的耐受力,厌氧氨氧化过程在氧浓度略高的环境中仍存在;2)观察到的厌氧氨氧化细菌为受到氧气抑制作用处于休眠状态的细胞,氧气仍然是厌氧氨氧化过程的重要限制因素。由于基因扩增和类脂分析技术不能区分细胞的活性状态和休眠状态,因而目前无法断定氧气对厌氧氨氧化过程的具体影响。厌氧氨氧化细菌对氧气的耐受范围及氧气对其的具体影响会成为今后一段时期研究的一个热点和重点。
3.4 氮 盐
3.5 深度和pH
尚未见有关海洋中厌氧氨氧化细菌随水体或沉积物深度变化的研究报道。但是,2008年舒青龙等通过对南海不同深度沉积物中与厌氧氨氧化细菌相关的浮霉菌门细菌的研究发现,浮霉菌门细菌多样性随深度增加而减少,且其存在的极限深度为5 m[35]。在水稻土壤中观察到厌氧氨氧化细菌多样性随深度增加而减少的明显趋势。表层水稻土壤中包括CandidatusKuenenia、CandidatusJettenia和CandidatusAnammo-xoglobus三个属,随深度的增加多样性降低,深层(表层下方1 m处)水稻土壤中只发现CandidatusBrocadia的存在[36]。该研究同时也发现了厌氧氨氧化细菌丰度存在垂直差异,16S rRNA基因丰度最高处为表层土壤下方40~50 cm处,为1.1×10716S rRNA基因copies/g[36]。海洋厌氧氨氧化细菌多样性是否同陆源研究结果相同,有关海洋环境中厌氧氨氧化细菌多样性、丰度随深度的变化趋势以及其对深度的极限适应范围需进一步研究。目前发现厌氧氨氧化细菌的海洋或淡水环境都有较适pH(6~8.9),研究环境并未包括显著酸性或碱性条件。因此,厌氧氨氧化细菌对pH的耐受范围,是下一步深入研究的方向。
3.6 季 节
厌氧氨氧化细菌物种组成、丰度及活性在冬、夏两季表现出明显差异。季节差异性研究目前多集中于淡、海水混合的河口环境,海洋环境中相关研究应在日后加强。
2011年李蒙等人在香港米浦湿地自然保护区河口沉积物中观察到,夏季厌氧氨氧化细菌包括CandidatusKuenenia、CandidatusScalindua和CandidatusAnammoxoglobus三个属,而在冬季则只包括CandidatusScalindua和CandidatusAnammoxoglobus两个属。CandidatusKuenenia只存在于夏季该地区沉积物中。他们推断人类活动和陆源输入是引起上述厌氧氨氧化细菌物种组成季节差异的主要原因。夏季周边河流的输入,引入了丰富的营养盐以及适应寡盐水环境的Candidatus Kuenenia[36]。与此相似,在珠江口沉积物中也观察到厌氧氨氧化细菌物种组成的季节差异。但不同的是物种组成从夏季到冬季由CandidatusJettenia、CandidatusBrocadia和CandidatusKuenenia转变为CandidatusBrocadia和CandidatusKuenenia[24]。这些研究表明,厌氧氨氧化细菌夏季的多样性高于冬季的,但具体物种组成的改变在不同地区存在差异。
厌氧氨氧化细菌丰度、活性在冬、夏两季也表现出明显不同。在香港米浦湿地沉积物中夏季厌氧氨氧化细菌丰度高于冬季的[37]。然而,在珠江口沉积物中却发现了相反的变化趋势,厌氧氨氧化细菌的丰度在冬季较高,为(1.4~20)× 108hzs基因copies/g,在夏季仅为(1.3~12)× 106hzs基因copies/g[24]。同时,厌氧氨氧化细菌活性在冬季明显高于夏季的,且已证实丰度与活性之间无明显关系[24]。
季节影响包括温度、人为输入、水体流动速度等多种环境因素。现已证实,河口地区冬、夏两季相比,厌氧氨氧化细菌多样性在夏季较高;活性在冬季较高,但具体的影响因素目前尚不明确。海洋环境具有受人为干扰小、营养盐含量相对稳定等特点,在分析季节性差异与环境因子相关性方面具有优势。海洋中厌氧氨氧化细菌物种组成、丰度及活性的季节变化;导致季节差异性的具体环境原因,可作为今后海洋厌氧氨氧化细菌研究的重点。

4 海洋厌氧氨氧化细菌环境适应性研究展望
4.1 对环境因子响应研究的深入
作为海洋氮循环研究的新问题及热点问题,环境因子对厌氧氨氧化细菌物种组成、丰度及厌氧氨氧化活性的影响现已在多个海区、多种环境中被证实并具有一定规律。但仍有相关研究需进一步深入,我们认为以下方面可以作为今后海洋厌氧氨氧化细菌环境响应研究的重点:1)海洋中厌氧氨氧化细菌多样性随深度的变化规律——多样性是否随深度增加呈现递减趋势或呈现先增后减的趋势;2)导致厌氧氨氧化细菌物种组成、丰度及厌氧氨氧化活性季节性差异的具体环境因子——是否不同季节氮素等营养盐人为排放导致的环境污染是其中最主要的影响因素;3)氧浓度对厌氧氨氧化细菌细胞活性及厌氧氨氧化过程的抑制作用——较高的氧浓度是否会导致厌氧氨氧化细菌细胞处于休眠状态;4)厌氧氨氧化细菌对温度、深度、溶氧量及pH的最高和最低耐受值。此外,目前海洋厌氧氨氧化细菌的研究多集中在近海区域,缺乏远海及深海环境中厌氧氨氧化细菌的研究数据,大大限制了对海洋中厌氧氨氧化过程的准确评估,今后应加强对深海及远海厌氧氨氧化细菌的研究。
4.2 扩增引物的改进
分子生物学技术作为目前研究厌氧氨氧化细菌的主要生物学手段,同样广泛应用于海洋厌氧氨氧化细菌的研究中。但现有的某些技术在检测厌氧氨氧化细菌方面不能达到理想效果。如目前有多种引物可用于扩增厌氧氨氧化细菌16S rRNA基因序列,但扩增效果并不理想。扩增产物除了厌氧氨氧化细菌相关序列之外,还包括非厌氧氨氧化细菌序列,如γ-变形菌门[38]、疣微菌门[19]等。李蒙等曾报导在用Brod541F/Brod1260R引物对扩增厌氧氨氧化细菌16S rRNA基因时,还会同时扩增出隶属于霍乱弧菌、生丝微菌、产左聚糖微杆菌的基因序列[19]。另外,16S rRNA基因和hzo功能基因在分析结果上也存在一些差异:对于同一样品,16S rRNA基因进化分析会发现CandidatusBrocadia而未发现CandidatusAnammoxoglobus,相反,hzo功能基因的进化分析常常发现CandidatusAnammoxoglobus却不能发现CandidatusBrocadia[13]。以上结果可能是由于引物的特异性和覆盖度不同所引起的,因此,为了更好地了解厌氧氨氧化细菌的多样性,应该加强对更为有效的扩增引物的设计。
[1] THAMDRUP B, DALSGAARD T, JENSEN M M, et al. Anaerobicammonium oxidation in the oxygen-deficient waters offnorthern Chile[J]. Limnol. Oceanogr., 2006, 51(5): 2145-2156.
[2] HONG Y G, LI M, GU J D, et al. Bacterial anaerobic ammonia oxidation(Anammox)in the marine nitrogen cycle-A review[J]. Acta Microbiologica Sinica, 2009, 49(3): 281-286. 洪义国,李猛,顾继东,等.海洋氮循环中细菌的厌氧氨氧化[J].微生物学报,2009, 49(3): 281-286.
[3] TEESELING M C F, ALMEIDA N M, KLINGL A, et al. A new addition to the cell plan of anammox bacteria:CandidatusKuenenia stuttgartiensis has a protein surface layer as the outermost layer of the cell[J]. Journal of Bacteriology, 2014, 196(1): 80-89.
[4] KUYPERS M M M, SLIEKERS A O, LAVIK G, et al. Anaerobic ammonium oxidation by anammox bacteria in the Black Sea[J]. Nature, 2003, 422: 608-611.
[5] HONG Y G, LI M, CAO H, et al. Residence of habitat-specific anammox bacteria in the deep-sea subsurface sediments of the South China Sea: analyses of marker gene abundance with physical chemical parameters[J]. Microbial Ecology, 2011, 62(1): 36-47.
[6] SCHMID M, TWACHTMANN U, KLEIN M, et al. Molecular evidence for genus level diversity of bacteria capable of catalyzing anaerobic ammonium oxidation[J]. Systematic and Applied Microbiology, 2000, 23:93-106.
[7] QUAN Z X, RHEE S K, ZUO J E, et al. Diversity of ammonium-oxidizing bacteria in a granular sludge anaerobic ammonium-oxidizing (anammox) reactor[J]. Environmental Microbiology, 2008, 10:3130-3139.
[8] DAMSTE J S S, STROUS M, RIJPSTRA W I C, et al. Linearly concatenatedcyclobutane lipids form a dense bacterial membrane[J]. Nature, 2002, 419(6908): 708-712.
[9] KARTAL B, NIFTRIK L, RATTRAY J, et al.CandidatusBrocadia fulgida: an autofluorescent anaerobic ammonium oxidizing bacterium[J]. FEMS Microbiology Ecology, 2008, 63: 46-55.
[10] KARTAL B, RATTRAY J, NIFTRIK L A, et al.Candidatus“Anammoxoglobus propionicus” a new propionate oxidizing species of anaerobic ammonium oxidizing bacteria[J]. Systematic and Applied Microbiology, 2007, 30: 39-49.
[11] WOEBKEN D, LAM P, KUYPERS M M M, et al. A microdiversity study of anammox bacteria reveals a novel Candidatus Scalindua phylotype in marine oxygen minimum zones[J]. Environmental Microbiology, 2008, 10(11): 3106-3119.
[12] KLOTA M G, STEINTEIN L Y. Nitrifier genomics and evolution of the nitrogen cycle[J]. FEMS Microbiology Letters, 2008, 278(2): 146-156.
[13] LI H, CHEN S, MU B Z, et al. Molecular detection of anaerobic ammonium-oxidizing (anammox) bacteria in high-temperature petroleum reservoirs[J]. Microbial Ecology, 2010, 60(4): 771-783.
[14] HIRSCH M D, LONG Z T, SONG B. Anammox bacterial diversity in various aquatic ecosystems based on the detection of hydrazine oxidase genes (hzoA/hzoB)[J]. Environmental Microbiology, 2011, 61(2): 264-276.
[15] HARRY R H, ROY M L, ALEN T, et al. Hydrazine synthase, a unique phylomarker with which to study the presence and biodiversity of anammox bacteria[J]. Applied and Environmental Microbiology, 2012, 78(3): 752-758.
[16] LI M, HONG Y G, CAO H L, et al. Community structures and distribution of anaerobic ammonium oxidizing and nirS-encoding nitrite-reducing bacteria in surface sediments of the South China Sea[J]. Microbial Ecology, 2013, 66(2): 281-296.
[17] DANG H Y, CHEN R P, WANG L, et al. Environmental factors shape sediment anammox bacterial communities in hypernutrified JiaozhouBay, China[J]. Applied and Environmental Microbiology, 2010, 76: 7036-7047.
[18] DALE O R, TOBIAS C R, SONG B. Biogeographical distribution of diverse anaerobic ammonium oxidizing (anammox) bacteria in Cape Fear River Estuary[J]. Environmental Microbiology, 2009, 11(5): 1194-1207.
[19] LI M, HONG Y G, KLOTZM G, et al. A comparison of primer sets for detecting 16S rRNA and hydrazine oxidoreductase genes of anaerobicammonium-oxidizing bacteria in marine sediments[J]. Applied Microbiology and Biotechnology,2010, 86(2):781-790.
[20] HAMERSLEY M R, LAVIK G, WOEBKEN D, et al. Anaerobic ammonium oxidation in the Peruvian oxygen minimum zone[J]. 2007, Limnology and Oceanography, 52(3): 923-933.
[21] JETTEN M S M, SLIEKERS O, KUYPERS M, et al. Anaerobic ammonium oxidation by marine and freshwater Planctomycete-like bacteria[J]. Applied Microbiology and Biotechnology,2003, 63(2):107-114.
[22] AMANO T, YOSHINAGE I, OKADA K, et al. Detection of anammox activity and diversity of anammox bacteria-related16S rRNA genes in coastal marine sediments in Japan[J]. Microbes Environ., 2007, 22:232-242.
[23] SCHMID M C, RISGAARD-PETERSEN N, VOSSENBERG J, et al.Anaerobic ammonium-oxidizing bacteria in marine environments:widespread occurrence but low diversity[J]. Environmental Microbiology,2007, 9(6): 1476-1486.
[24] WANG S Y, ZHU G B, PENG Y Z, et al. Anammox bacterial abundance, activity, and contribution in riparian sediments of the Pearl River Estuary[J]. Environmental Science and Technology, 2012, 46(16): 8834-8842.
[25] HOU L J, ZHENG Y L, LIU M, et al. Anaerobic ammonium oxidation (anammox) bacterial diversity, abundance, and activity in marsh sediments of the Yangtze Estuary[J]. Journal of Geophysical Research Biogeosciences, 2013, 118(3): 1237-1246.
[26] KARTAL B, KOLEVA M, ARSOV R, et al. Adaptation of a freshwater anammox population to high salinity wastewater[J]. Journal of Biotechnology,2006, 126(4):546-553.
[27] RYSGAARD S, GLUD R N. Anaerobic N2production in Arctic sea ice[J]. Limnology and Oceanography, 2004, 49(1): 86-94.
[28] BYRNE N, STROUS M, CREPEAU V, et al. Presence and activity of anaerobic ammonium-oxidizing bacteria at deep-sea hydrothermal vents[J]. ISME Journal, 2009, 3: 117-123.
[29] RICH J J, DALE O R, SONG B, et al. Anaerobic ammonium oxidation (anammox) in Chesapeake Bay sediments[J]. Microbial Ecology, 2008, 55(2): 311-320.
[30] JAESCHKE A, CAMP H J M, HARHANGI H, et al. 16S rRNA gene and lipid biomarker evidence for anaerobic ammonium-oxidizing bacteria (anammox) in California and Nevada hot springs[J]. FEMS Microbiology Ecology, 2009, 67(3): 343-350.
[31] JETTEN M S M, STROUS M, PAS-SCHOONEN K T, et al. The anaerobic oxidation of ammonium[J]. FEMS Microbiology Reviews, 1999,22(5): 421-437.
[32] DAIGGER G T. Oxygen and carbon requirements for biological nitrogen removal processes accomplishing nitrification, nitritation, and anammox[J]. Water Environment Research, 2014, 86: 204-209.
[33] MEYER R L, RISGAARD-PETERSEN N, ALLEN D E. Correlation between anammox activity and microscale distributionof nitrite in a subtropical mangrove sediment[J]. Applied and Environmental Microbiology, 2005, 71(10): 6142-6149.
[34] TRIMMER M, NICHOLLS J C, DEFLANDRE B. Anaerobic ammoniumoxidation measured in sediments along the Thames estuary, United Kingdom[J]. Applied and Environmental Microbiology, 2003, 69: 6447-6454.
[35] SHU Q L, JIAO N Z. Profiling planctomycetales diversity with reference toanammox-related bacteria in a South China Sea, deep-sea sediment[J]. Marine Ecology,2008, 29(4): 413-420.
[36] ZHU G B, WANG S Y, WANG Y, et al. Anaerobic ammonia oxidation in a fertilizedpaddy soil[J]. ISME Journal,2011, 5: 1905-1912.
[37] LI M, CAO H L, HONG Y G, et al. Seasonal dynamics of anammox bacteria in estuarial sediment of the MaiPo Nature Reserve revealed by analyzing the 16S rRNA and hydrazine oxidoreductase (hzo) genes[J]. Microbes Environ., 2011, 26: 15-22.
[38] GALAN A, MOLINA V, THAMDRUP B, et al. Anammox bacteria and the anaerobic oxidation of ammonium in the oxygen minimum zone off northern Chile[J]. Deep-Sea Research II, 2009, 56(16): 1021-1031.
ProgressinStudyonResponseofAnammoxBacterialSpeciesCompositiontoEnvironmentalFactors
FU Bing-bing, LIU Ji-wen, GAO Ming-hong, ZHANG Xiao-hua
(CollegeofMarineLifeSciences,OceanUniversityofChina, Qingdao 266003, China)
anammox bacteria; community structure; abundance; anammox activity; environmental adaptability
March 27, 2014
2014-03-27
国家自然科学基金项目——南太平洋环流区洋底沉积物及海水的微生物群落演化及功能活性研究(41276141)
富冰冰(1988-),女,满族,吉林柳河人,硕士研究生,主要从事海洋微生物学方面研究.E-mail:mufashabing@126.com
*通讯作者,张晓华(1965-),女,博士,教授,主要从事海洋微生物学方面研究.E-mail:xhzhang@ouc.edu.cn
(高 峻 编辑)
Q938.1
A
1671-6647(2014)03-0427-08

