恒河猴samhd1基因编码区多态性及其对蛋白结构和功能的影响
熊圣文,王 卫,董志会,陈 霆,魏 强
(北京协和医学院比较医学中心,中国医学科学院医学实验动物研究所,卫生部人类疾病比较医学重点实验室,国家中医药管理局人类疾病动物模型三级实验室,北京 100021)
SAMHD1(SAM domain and HD domain 1) 是近年来发现的对HIV-1等逆转录病毒复制具有明显限制作用的天然抗病毒蛋白之一[1,2],而在恒河猴体内也存在抑制猴免疫缺陷病毒(simian immunodeficiency virus, SIV)复制的samhd1基因[3]。前期研究表明,Trim5α和Apobec3G等天然抗病毒蛋白在进化过程中,会出现影响抗病毒功能的核苷酸突变;进化分析也证实,samhd1基因部分氨基酸残基位点可能影响Vpx对SAMHD1的敏感性[3]。但有关恒河猴samhd1基因多态性,及对蛋白结构和功能的影响尚未见报道。本研究旨在通过基因测序,序列比对,蛋白质结构预测等技术找到恒河猴Samhd1基因中影响抗SIV功能的cSNP位点,从而为探索SAMHD1抗病毒机制和优化SIV/SHIV/恒河猴模型提供研究基础。
1 材料和方法
1.1 实验动物与样品采集
中国恒河猴138只,购自北京协尔鑫生物资源研究所(合格证编号:SCXK(京)2005-2005);分别采集动物外周血1 mL,EDTA抗凝,样品采集后4 h内提取RNA。
1.2 外周血RNA的提取及反转录
实验猴EDTA抗凝全血使用RNA blood mini kit(购自Qiagen公司)进行总RNA的提取,提取过程参照试剂盒说明书;提取RNA后立即对其进行反转录。
1.3 RT-PCR扩增
Samhd1基因扩增采用巢式PCR,第一轮RT-PCR使用One Step RNA PCR Kit (AMV)(TaKaRa,大连)进行,操作过程参照试剂盒说明书;第二轮PCR使用KOD-Plus试剂盒(TOYOBO, 上海)进行,操作过程参照试剂盒说明书。Samhd1基因引物由引物设计软件Primer Premier 5.0设计完成,由Invitrogen公司合成。外套引物序列为:outer-F 5′-CCC GAA GGG CTC AAC TGT CA-3′; outer-R 5′-AGA CTT TAA TAA TAC TAA AAA TAG GCA AGG-3′,退火温度为44.5℃。内套引物序列为:inner-F 5′-CAA TAC TGC TTG GAC TCC CCG CC-3′;inner-R 5′-ATT CTA GGA GTG AAG CCC CTA AAT GAA TT-3′,退火温度为51℃。PCR在System 9700 PCR仪上进行(Applied Biosystems公司)。
1.4 序列比对和生物软件分析
DNA测序由北京诺赛基因公司完成。利用DNAstar软件进行核苷酸和氨基酸序列比对[4],标准恒河猴[Macacamulatta] SAMHD1 cDNA序列为:NM_001271642.1;标准恒河猴[Macacamulatta] SAMHD1氨基酸序列为:NP_001258571。
分别用Predictprotein、SWISS-MODEL软件进行蛋白质二级和三级结构的预测。
1.5 SNaPshot分型检测
SNP的分型检测采用SNaPshot技术,即基于荧光标记单碱基延伸原理的分型技术。该检测过程交由北京阅微基因公司,基于3730XL平台完成。
2 结果
2.1 中国恒河猴Samhd1基因编码区cSNP位点的分布
提取29只恒河猴全血RNA,逆转录PCR后进行测序比对(参照序列为NCBI上公布的Samhd1 cDNA序列,NP_001258571),一共找到11个突变位点,分别位于基因的N端、SAM结构域、链接区、HD结构域和 C端(表1);通过使用DNAstar软件进行翻译和氨基酸序列比对,发现其中5个突变位点为非同义突变,在Samhd1编码区上的位置分别是:15 bp (C/G)、 320 bp (C/T)、 547 bp (G/A)、 552 bp (A/T)、839 bp (T/C)[5](图1)。其中G183S、E184D、V280A分布在HD结构域上;S107L分布在SAM结构域上;D5E分布在N端非结构域上。
2.2 非同义cSNP对SAMHD1蛋白二级结构的影响
Predictprotein软件分析结果显示,5个非同义cSNP位点突变影响了SAMHD1蛋白的二级结构,具体的,在264aa位置附近一个α螺旋结构变成β片层,530aa附近增加了一个α螺旋。295aa附近的蛋白质结合位点移动到540aa附近,其它结构均未发生变化(图2,见彩插1)。但当单独出现280aa或 183aa和184aa的突变时,软件分析结果显示并不引起此二级结构变化(图未列出)。
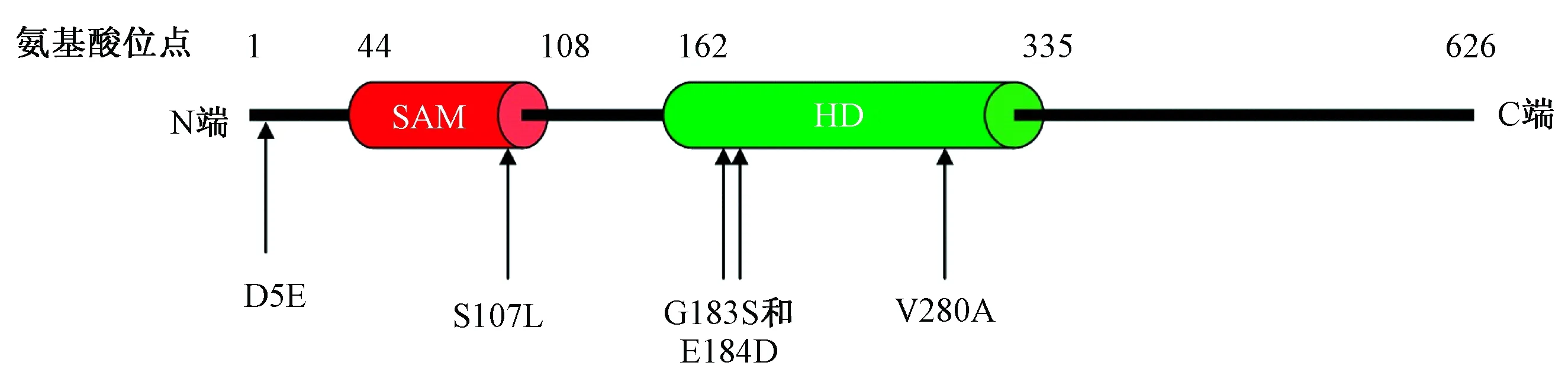
图1 恒河猴SAMHD1的蛋白质结构及SNP分布情况
表1恒河猴samhd1基因突变位点
Tab.1samhd1 nucleotide mutants of the rhesus macaques
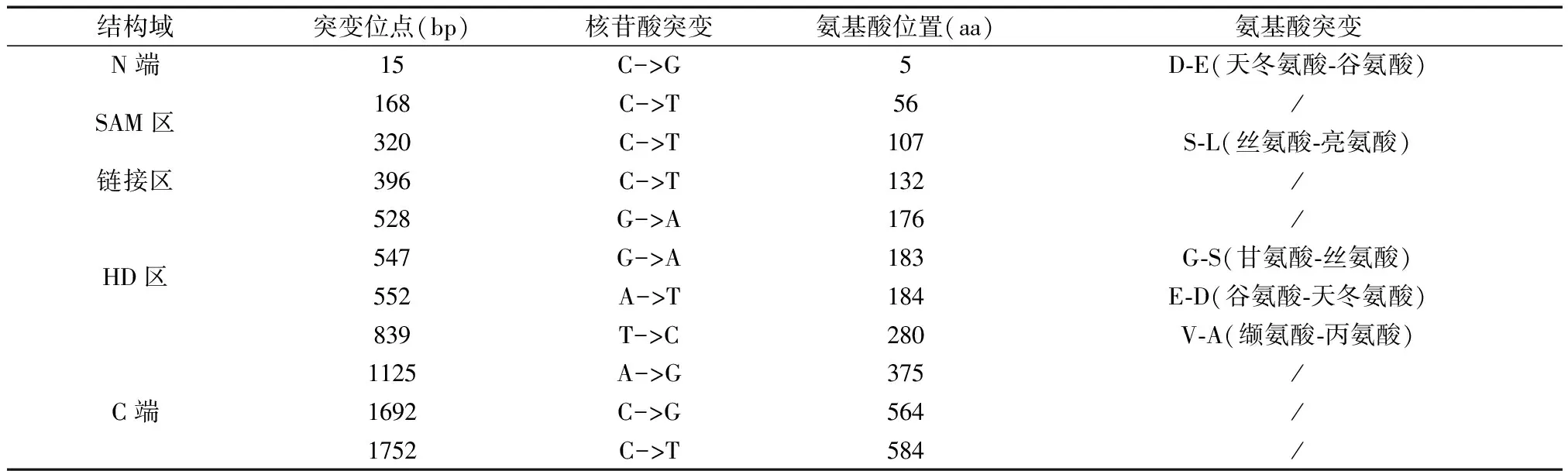
结构域突变位点(bp)核苷酸突变氨基酸位置(aa)氨基酸突变N端15C->G5D-E(天冬氨酸-谷氨酸)SAM区168C->T56/320C->T107S-L(丝氨酸-亮氨酸)链接区396C->T 132/528G->A176/HD区547G->A183G-S(甘氨酸-丝氨酸)552A->T184E-D(谷氨酸-天冬氨酸)839T->C280V-A(缬氨酸-丙氨酸)1125A->G375/C端1692C->G 564/1752C->T584/
2.3 非同义cSNP对SAMHD1蛋白功能的影响
为预测cSNP对SAMHD1蛋白功能的影响,使用SWISS-MODEL软件对SAMHD1蛋白不同结构域进行结构预测。
图3A所示samhd1基因SAM结构域的三级结构分析图,绿色标出文献中通过进化分析找到的影响Vpx对SAMHD1敏感性的位点,其中,包括红圈中的S107L位点,说明S107L影响病毒Vpx蛋白对SAMHD1的敏感性。
图3B为samhd1基因HD结构域的三级结构分析图,可以看到G183S、E184D、V280A三个位点均分布在蛋白质表面,且和文献中已知的重要位点(绿色标出)距离较远,其中V280A处于蛋白质活性中心(凹槽部位)的袋口处,类似于盒盖的结构上,可能会影响到SAMHD1的脱氧核苷酸三磷酸水解酶催化活性;而G183S、E184D两个位点均分布在凹槽的背面,但文献中报道的一些可能影响Vpx对SAMHD1敏感性的位点也位于类似位置(如图3B“凹槽”下方的绿色位点),所以不能排除G183S、E184D两个位点会影响SAMHD1与Vpx结合的可能性(图3见彩插1)。
2.4 基因分型及cSNP位点的频率测定
根据发现的核苷酸有义突变位点,利用SNaPshot技术对138只中国恒河猴进行了基因分型并验证是否为cSNP位点。通过SNaPshot检测,发现138只中国恒河猴中C15G基因频率占2.17%;C320T的基因频率为6.88%;G547A的基因频率为13.41%;A552T的基因频率为4.35%;T839C的基因频率为17.39%,这5个位点均可判定为SNP(基因频率大于1%)。另外还发现G547A和T839C存在较强的连锁关系,连锁频率占21.01%。对基因分型结果进行分析,本实验共找到了6种突变基因型(根据5个cSNP在基因中的前后顺序,将正常基因型表示为:… C…C…GA…T…),详情见表2。

表2 恒河猴samhd1突变基因型及比率
3 讨论
SAMHD1主要在单核细胞、单核细胞来源的巨噬细胞、树突状细胞等髓系细胞以及静息CD4+T细胞中表达[1]。SAMHD1具有磷酸水解酶活性,受dGTP的调控,它对HIV-1复制的限制是通过降解细胞内dNTP的水平[6,7], 使胞内dNTPs的水平低于病毒复制的需要水平,从而切断HIV-1逆转录所需底物的来源;这一过程被称为“核苷酸仓库耗竭”。 研究发现SAMHD1对HIV-1 的复制具有明显的限制作用,但可以被HIV-2和SIV编码的Vpx蛋白拮抗[2]。另外,有些Vpr蛋白也能起到类似作用[3]。2011年,Powell et al通过功能实验找到了20多个与SAMHD1蛋白功能有关的氨基酸残基位点[8]。2012年,Lim et al通过正向选择分析找到6个氨基酸残基位点可能影响Vpx对SAMHD1的敏感性[3]。因此,氨基酸残基位点的突变对于SAMHD1的抗病毒功能将可能产生很大影响。
非同义cSNP碱基序列的改变可使以其为模板翻译的氨基酸序列发生改变,从而影响蛋白质的功能。这种改变常是导致生物性状改变的直接原因。因此,本研究主要分析了SAMHD1分子的编码区非同义SNP位点。
和人类相似,在进化过程中恒河猴SAMHD1分子上也存在着大量的SNP位点。 2012年,Coon et al在分析人samhd1基因多态性时,发现SNP (rs1291142)与SAMHD1表达量显著相关[7]。因此,我们对恒河猴SAMHD1分子的编码区突变情况进行了大样本的筛查,总共有5个位点被鉴定为非同义cSNP,在群体中均占有较高的频率,其中G183S、E184D、V280A分布在具磷酸水解酶活性的HD结构域上;S107L分布在参与蛋白-蛋白或蛋白-RNA 相互作用的SAM结构域上。
为了判断这些cSNP是否能影响到蛋白质的功能,我们先进行了蛋白高级结构和功能预测分析。用软件对SAMHD1二级、三级结构的分析发现,5个cSNP中G183S、E184D、V280A三个位点最有可能影响蛋白质的功能和结构,在二级结构上,这三个位点所处的HD结构域发生了一个α螺旋到β片层的转变,一个蛋白质结合位点也因为这三个cSNP的出现而发生了移位;在三级结构上,V280A正好处于蛋白质活性中心(凹槽部位)的袋口处,类似于盒盖的结构上,可能会影响到SAMHD1的磷酸水解酶催化活性。另外,发现S107L与文献中报道的可能影响病毒Vpx蛋白对SAMHD1敏感性的位点重合,说明此位点也有较大可能会改变蛋白质的功能。D5E未分布于重要的蛋白结构域上,目前还没有能改变蛋白功能的证据支持,但也有文献报道,SAMHD1蛋白的N端能够与艾滋病毒相互作用,所以有待进一步的实验验证。如果实验验证这些位点与抗病毒功能有关,则说明该蛋白与SIV在相互进化过程中发生了选择作用。
这些分析为之后蛋白功能实验提供了大量的参考依据,同时也为SAMHD1抗病毒机制的进一步研究发掘了新的靶点。我们后续还将分别对在SAMHD1上发现的6种基因型进行蛋白功能实验验证。
参考文献:
[1] Baldauf HM, Pan X, Erikson E, et al.SAMHD1 restricts HIV-1 infection in resting CD4(+) T cells [J]. Nat Med, 2012, 18(11):1682-1687.
[2] Laguette N, Sobhian B, Casartelli N, et al.SAMHD1 is the dendritic- and myeloid-cell-specific HIV-1 restriction factor counteracted by Vpx [J]. Nature, 2011. 474(7353):654-657.
[3] Lim ES, Fregoso OI, McCoy CO, et al. The ability of primate lentiviruses to degrade the monocyte restriction factor SAMHD1 preceded the birth of the viral accessory protein Vpx [J]. Cell Host Microbe, 2012, 11(2):194-204.
[4] 刘克剑, 丛喆, 金光, 等. 表现神经症状的SIVmac251感染猴大脑基底节病毒gp120序列变异分析 [J].中国实验动物学报, 2012, 2:37-42.
[5] 李佩璐, 陈倩倩, 张驰宇. 髓系单核细胞来源的 HIV-1 限制性因子—SAMHD1 [J].动物学研究, 2012, 33(5):537-541.
[6] Powell RD, Holland PJ, Hollis T, et al. Aicardi-Goutieres syndrome gene and HIV-1 restriction factor SAMHD1 is a dGTP-regulated deoxynucleotide triphosphohydrolase [J]. J Biol Chem, 2011, 286(51):43596-43600.
[7] Goldstone DC, Ennis-Adeniran V, Hedden JJ, et al.HIV-1 restriction factor SAMHD1 is a deoxynucleoside triphosphate triphosphohydrolase [J]. Nature, 2011, 480(7377):379-382.
[8] Coon S, Wang D, Wu L. Polymorphisms of the SAMHD1 gene are not associated with the infection and natural control of HIV type 1 in Europeans and African-Americans [J]. AIDS Res Human Retroviruses, 2012, 28(12): 1565-1573.

