Biolog ECO和DGGE 数据几种分析方法的比较研究
,, ,
(1.云南农业大学 农业生物多样性研究中心,云南 昆明 650201;2.宣威市农业局农技推广中心,云南 宣威 655400)
过去的研究中,微生物多样性的数据采集方法主要包括:Biolog ECO[1]、变性梯度凝胶电泳(DGGE)[2-3]、磷脂脂肪酸(PLFA)[4]、宏基因组(Metagenome)[5]、随机扩增多态性DNA标记(RAPD)[6]、扩增片段长度多态性(AFLP)[7]等技术。其中,Biolog ECO和DGGE是微生物研究的重要工具。微生物多样性的数据是多元变量,目前,多元变量分析方法有无监督分析方法及有监督的分析方法2种。无监督分析方法中的主成分(PCA)常用于不同环境条件下微生物多样性差异的研究,但是无监督分析方法存在以下问题:无法建立模型及进行可靠性检测;无法进行分类,无法识别样本间微小差异,目标性弱,结果分散,效率低;临界值的判定依赖PCs的得分系数,目前无统一的临界值。有监督的分析方法有最小二乘法(PLS)[8]、正交最小二乘法(OPLS)[9-10]、kernel 函数正交最小二乘法(K-OPLS)[11]、最小二乘法判别法(PLS-DA)[12]、最小二乘法增强判别法(PLS-EDA)、正交最小二乘法判别法(OPLS-DA)[13]、kernel函数正交最小二乘法判别法(K-OPLS-DA)[14]等。有监督的分析方法克服无监督分析方法的弱点,可对微生物数据进行分组、分类,结果直观,目标性强,采用VIP(Variable important)≥1.00作为临界值。目前,有监督的分析方法仅有最小二乘法(PLS)[8]用于微生物多样性的研究。
为更好地解释不同环境对土壤微生物的碳源代谢及物种多样性影响,研究通过比较PCA、PLS-DA、PLS-EDA及PLS、OPLS分析法,来实现微生物在不同环境中差异及差异代谢碳源的鉴定及物种的鉴定。
1 材料与方法
1.1 土壤取样
于2011年,分别在昭通靖安及鲁甸、昆明寻甸取玉米与马铃薯间作(种植模式2∶2)收获时期的土壤样品,每个点取3~5份。用于Biolog ECO分析的土壤样品以最快速度置于4℃冰箱保存,用于DGGE分析的土壤样品用1.5 ml离心管装好置于-80℃冰箱保存。
1.2 基质利用的评价
采用Biolog ECO(Biolog .Inc.CA)平板作为基质,平板培养及数据读取参照文献进行[1],获得的数据选择120 h进行多元变量分析。
1.3 土壤DNA的提取及PCR-DGGE
土壤DNA采用百泰克(E.Z.N.A.®Soil DNA Kit)按照说明书提取。
1.3.1PCR。选取细菌16SrRNA V6-V9高变区引物:F-968-GC (5’-CGC CCG GGG CGC GCC CCG GGCGGG GCG GGG GCA CGG GGG GAA CGC GAA GAA CCT TAC-3’)和R-1401(5’-CGG TGT GTA CAA GAC CC-3’),PCR产物的片段为500 bp,PCR方法采用TD-PCR,94℃ 8 min,94℃ 1 min,60℃-50℃ 1 min,72℃ 1 min 10个循环,94℃ 1 min,50℃ 1 min,72℃ 1 min 25个循环,72℃ 7 min,4℃保存,反应结束后置于-20℃冰箱储存备用。反应体系:2×Master Mix 25 μL,dd H2O 21.50 μL,BSA 0.5 μL,F-968-GC 1 μL,R-1401 1 μL,模板1 μL,采用1.2%琼脂糖电泳确认PCR产物。
1.3.2DGGE。100 μL PCR产物用Bio-Rad旋蒸仪浓缩5~6倍,The Dcode Universal Mutation Detection Delivery System(Bio-Rad )用于灌制30% ~ 65%梯度胶。具体过程为:各取15 mL30%和65%的变性胶,加340 μL的溴酚蓝染液到65%高浓度胶中,混匀。然后,分别在30%和65%的变性胶加入170 μL过硫酸铵(10%)和17 μL TEMED,快速灌胶,聚合1 h ~ 2 h。将1×TAE在Bio-Rad Dcode Mutant detect system电泳槽中预热到60℃,浓缩样品与等体积的Loading buffer混合后,取20 μL上样到聚丙烯酰胺变性凝胶6% 梯度为30%~65%胶孔中,150 V,电泳8 h,1/10000 EB染色10 min,1×TAE脱色15 min,紫外光成像系统Gensnap拍照获取图谱。
1.4 数据分析
Simca-p 11.5(umertric)用于PLS 及OPLS及潜在标记的发现;采用MultiBase进行PCA、PLS-DA、PLS-EDA分析。选取Biolog ECO培养120,在590 nm条件下,31种碳源吸光度经消除空白后作为分析的原始数据;采用Quantity one 4.6.2软件对DGGE图谱数据化,统计条带数、亮度、位置。采用Microsoft office Excel 2010对条带按位置进行对齐处理,采用亮度作为分析的原始数据。DGGE和 Biolog ECO数据分析过程如下,首先在Microsoft office Excel 2010加载MultiBase 2014的宏,选中要分析的数据设置变量及处理,对数据进行分类,最后进行计算导出图形。
2 结果与分析
2.1 DGGE和 Biolog ECO数据的PCA、PLS-DA、PLS-EDA分析
图1为DGGE结果图,左边5条泳道表示昭通鲁甸5个取样点;右边5条泳道表示昭通靖安5个取样点。DGGE数据分析(图2-图4)表明:PCA的PC1为27.1%,有2个样品无法区分;PLS-DA的PC1为33%,有1个样品无法区分;PLS-EDA把昭通的2个试验点样品完全分开,PC1为16.8%。Biolog ECO数据分析(图5-图7)表明:PCA的PC1为35.1%,有6个样品无法区分;PLS-DA的PC1为36.3%,有4个样品无法区分;PLS-EDA把昭通的2个试验点和寻甸样品完全分开,PC1为25.5%。

图1 变性梯度凝胶电泳(DGGE) 图Fig.1 DGGE profile注:L,J表示昭通鲁甸和靖安试验点。
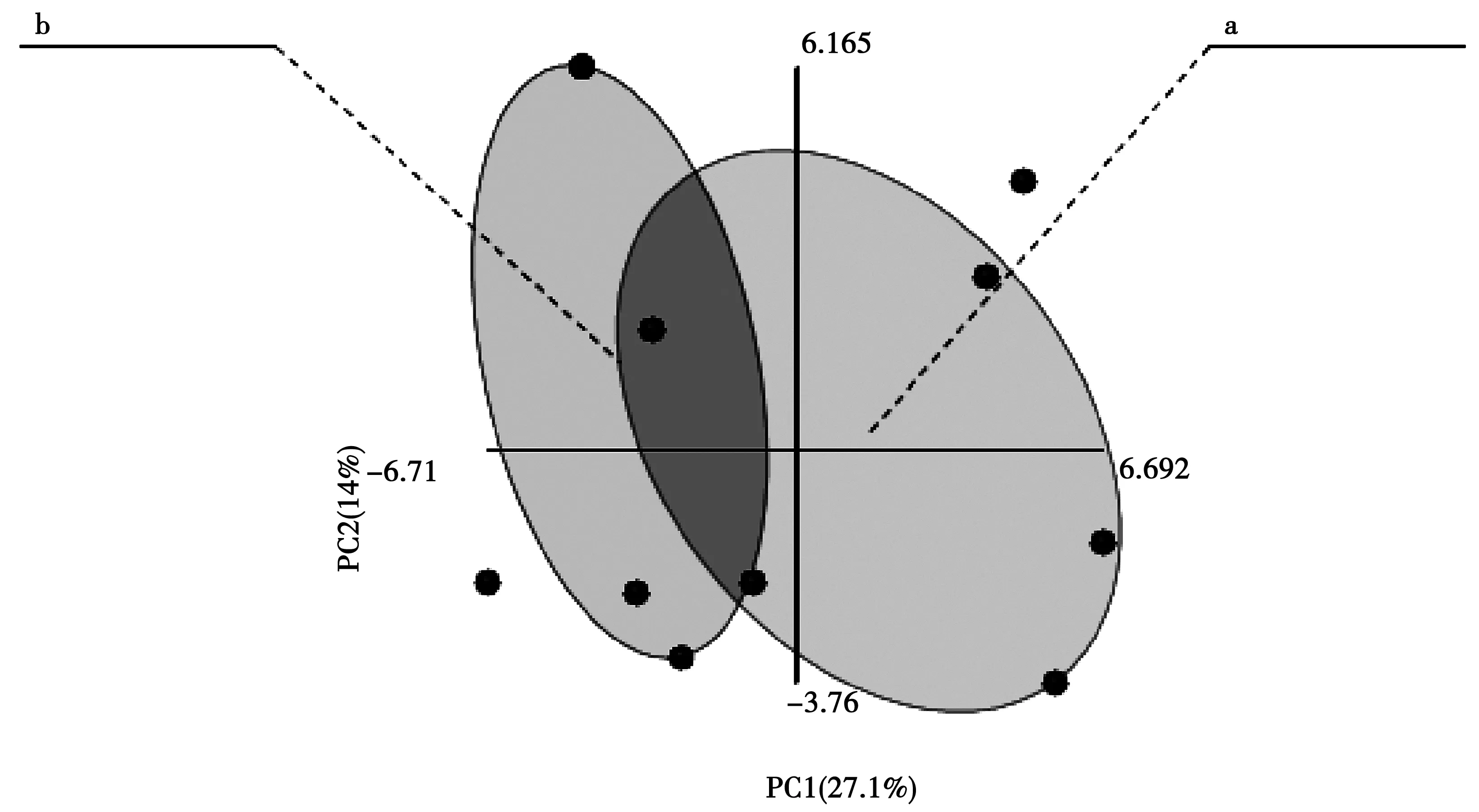
图2 变性梯度凝胶电泳(DGGE)数据的主成分分析(PCA)Fig.2 PCA analysis of DGGE data 注:a,b表示昭通两个不同的试验点,图3、图4同。

图3 变性梯度凝胶电泳(DGGE) 数据的最小二乘法判别法分析(PLS-DA)Fig.3 PLS-DA analysis of DGGE data

图4 变性梯度凝胶电泳(DGGE) 数据的最小二乘法增强判别法分析(PLS-EDA)Fig.4 PLS-EDA analysis of DGGE data
2.2 基于PLS和OPLS筛选环境微生物标记
采用PLS和OPLS筛选VIP≥1.00的条带和碳源作为潜在生物标记,见表1,筛选发现DGGE PLS 和OPLS筛选差异较大,PLS筛选出19个带型作为标记,OPLS筛选出11个带型作为标记;Biolog差异较小,OPLS与PLS相比多2种碳源,应综合考虑PLS和OPLS的筛选结果。DGGE考虑到后续克隆条带进行微生物种类鉴定,因此,可选PLS的VIP≥1.00的条带作为潜在生物标记。

表1 DGGE 和Biolog微生物候选标记Tab.1 Candidate for microbial makers in DGGE and Biolog
注:1-4、24-26、15、17-18、34-35、48、50-56、59、84、86 表示DGGE图谱分析结果;C2、D1、A8、D7、D5、D2、D4、D8、C4、C6、B6、C8、B4、A4表示Biolog ECO碳源种类。

图5 Biolog ECO数据的主成分分析(PCA)Fig.5 PCA analysis of Biolog ECO data注:a、b、c分别表示寻甸及昭通3个不同的试验点,图6、图7同。

图6 Biolog ECO 数据的最小二乘法判别法分析(PLS-DA)Fig.6 PLS-DA analysis of Biolog ECO data

图7 Biolog ECO数据的最小二乘法增强判别法分析(PLS-EDA)Fig.7 PLS-EDA analysis of Biolog ECO data
3 讨 论
土壤微生物Biolog ECO和DGGE数据分析多采用PCA[15-16],该方法区分不同环境样品依赖PCs得分系数,但是样品离散程度大,无法直观看出环境对土壤微生物多样性的影响,也有研究使用DCCA(Detrended canonical correspondence analysis)及CCA(Canonical correspondence analysis)方法反应环境对土壤微生物多样性的影响[17-19],但是目前CANCOCO是收费软件,操作复杂,况且DCCA及CCA方法还有无法克服的问题。也有较少的研究者采用PLS[8],PLS是一种有监督的分析方法,对环境数据进行回归分析,建立可解释变量的模型,但是需对模型的可靠性进行判定,才能确定是否合乎逻辑与事实。因此,PCA及PLS不能有效的解释环境变异与微生物多样性的关系[20-21]。
PLS-DA、PLS-EDA在代谢组学数据分析中有强大的优势,尤其是分析环境条件与代谢物的关系[13,22-23]。土壤微生物多样性的数据和代谢组数据具有相似性,都属于多元变量。因此,研究认为,该方法同样适用于微生物多样性与环境关系的研究。研究中采用PCA、PLS-DA、PLS-EDA 3种方法对Biolog ECO和DGGE数据分析作对比,发现PLS-EDA能够将不同环境的样品区分开来,PLS-DA能够区分出大多数样品,PCA无法区分不同环境的样品。因此,PLS-DA、PLS-EDA可作为分析微生物多样性数据的新方法。
已有的研究认为PCA 可从Biolog ECO和DGGE数据中能够确定哪些碳源或哪些条带对微生物群落结构变化起到主导作用,但是PCA结果过于笼统和模糊,依赖于碳源和带型在PCs上的荷载大小,但目前无统一荷载大小临界点值作为特征碳源或带型,一些研究中采取荷载筛选与多元方差分析结合,对特征碳源或带型进行显著性检测[15-16],此方法比较繁琐,因此,很难筛选出有价值的碳源和物种。研究采用PLS 和 OPLS两种方法筛选微生物标记,参照代谢组学特异物筛选方法,规定VIP≥1.00的碳源和物种作为环境潜在代谢和物种标记[24]。筛选结果表明:OPLS及PLS都可选出一批在不同环境中的土壤微生物潜在代谢和物种标记,能为进一步分析微生物的生态功能提供有价值的信息。此方法为研究不同环境下土壤微生物多样性的特征标记物进行规范化鉴定。
近年来,多元变量分析广泛使用于生物统计中,大量分析软件的出现,不仅提高了数据分析效率,而且结果更直观易懂[24]。在复杂样本及复杂环境数据分析中,多元变量分析为挖掘重要信息提供无以伦比的优势。PLS-EDA、OPLS、PLS作为近年来发展出的新方法,同样可作为土壤微生物多样性数据分析最有效、最重要的方法。
参考文献:
[1] Choi K,Dobbs F C.Comparison of two kinds of Biolog microplates (GN and ECO) in their ability to distinguish among aquatic microbial communities[J].Journal of Microbiological Methods,1999,36(3): 203-213.
[2] Muyzer G,Smalla K.Application of denaturing gradient gel electrophoresis (DGGE) and temperature gradient gel electrophoresis (TGGE) in microbial ecology[J].Antonie van Leeuwenhoek,1998,73(1): 127-141.
[3] Muyzer G,Brinkhoff T,Nübel U,et al.Denaturing gradient gel electrophoresis (DGGE) in microbial ecology[M].In: Akkermans ADL,van Elsas J D,de Bruijn F J,eds.Molecular Microbial Ecology Manual.Dordrecht:Kluwer Academic Publishers,1997.
[4] Green C T,Scow K M.Analysis of phospholipid fatty acids (PLFA) to characterize microbial communities in aquifers[J].Hydrogeology Journal,2000,8(1): 126-141.
[5] Rondon M R,August P R,Bettermann A D,et al.Cloning the soil metagenome: a strategy for accessing the genetic and functional diversity of uncultured microorganisms[J].Applied and environmental microbiology,2000,66(6): 2541-2547.
[6] Borneman J,Skroch P W,O'Sullivan K M,et al.Molecular microbial diversity of an agricultural soil in Wisconsin[J].Applied and Environmental Microbiology,1996,62(6): 1935-1943.
[7] LIU B R,JI G M,Chen J,et al.A review of methods for studying microbial diversity in soils[J].Pedosphere,2006,16(1): 18-24.
[8] Frostegård Å,Bååth E,Tunlio A,et al.Shifts in the structure of soil microbial communities in limed forests as revealed by phospholipid fatty acid analysis[J].Soil Biology and Biochemistry,1993,25(6): 723-730.
[9] Booth S C,Workentine M L,Wen J,et al.Differences in metabolism between the biofilm and planktonic response to metal stress[J].Journal of proteome research,2011,10(7): 3190-3199.
[10] Verron T,Sabatier R,Joffre R.Some theoretical properties of the O‐PLS method[J].Journal of chemometrics,2004,18(2):62-68.
[11] Stenlund H,Gorzsás A,Persson P,et al.Orthogonal projections to latent structures discriminant analysis modeling on in situ FT-IR spectral imaging of liver tissue for identifying sources of variability[J].Analytical chemistry,2008,80(18): 6898-6906.
[12] Chevallier S,Bertrand D,Kohler A,et al.Application of PLS‐DA in multivariate image analysis[J].Journal of Chemometrics,2006,20(5): 221-229.
[13] Qiu Y P,Cai G X,Su M M,et al.Serum metabolite profiling of human colorectal cancer using GC-TOFMS and UPLC-QTOFMS[J].Journal of proteome research,2009,8(10): 4844-4850.
[14] Tugizimana F,Piater L,Dubery I.Plant metabolomics: A new frontier in phytochemical analysis[J].South African Journal of Science,2013,109(5-6): 1-11..
[15] Cavigelli M A,Robertson G P,Klug M J,et al.Fatty acid methyl ester (FAME) profiles as measures of soil microbial community structure[J].Plant and soil,1995,170(1): 99-113.
[16] Kirk J L,Beaudette L A,Hart M,et al.Methods of studying soil microbial diversity[J].Journal of microbiological methods,2004,58(2): 169-188.
[17] 王 强,戴九兰,吴大千,等.微生物生态研究中基于 BIOLOG 方法的数据分析[J].生态学报,2010,30(3): 817-823.
[18] 邢德峰,任南琪.应用 DGGE 研究微生物群落时的常见问题分析[J].微生物学报,2006,46(2): 331-335.
[19] 辛 亮,武传东,曲 东.长期施肥对旱地土壤中氨氧化微生物丰度和分布的影响[J].西北农业学报,2012,21(6): 41-46.
[20] Pietikäinen J,Fritze H.Clear-cutting and prescribed burning in coniferous forest: comparison of effects on soil fungal and total microbial biomass,respiration activity and nitrification[J].Soil Biology and Biochemistry,1995,27(1): 101-109.
[21] Frostegård Å,Tunlid A,Bååth E,et al.Phospholipid fatty acid composition,biomass,and activity of microbial communities from two soil types experimentally exposed to different heavy metals[J].Applied and Environmental Microbiology,1993,59(11): 3605-3617.
[22] Weckwerth W.Metabolomics in systems biology[J].Annual review of plant biology,2003,54(1): 669-689.
[23] Bundy J G,Davey M P,Viant M R,et al.Environmental metabolomics: a critical review and future perspectives[J].Metabolomics,2009,5(1): 3-21.
[24] Xia J G,Mandal R,Sinelnikov I V,et al.MetaboAnalyst 2.0—a comprehensive server for metabolomic data analysis[J].Nucleic acids research,2012,40(W1): W127-W133.

