不同地理种源无患子的分子多态性分析
刘宝,范辉华,彭珠清,刘燕,陈存及
(1.福建农林大学 林学院,福建 福州 350002;2.福建省林业科学研究院,福建 福州 350012)
不同地理种源无患子的分子多态性分析
刘宝1,范辉华2,彭珠清1,刘燕1,陈存及1
(1.福建农林大学 林学院,福建 福州 350002;2.福建省林业科学研究院,福建 福州 350012)
利用筛选出的18对相关序列扩增多态性(SRAP)引物对12个来自印度和中国不同种源的无患子进行SRAP扩增,计算Dice遗传相似系数后用非加权组平均法(UPGMA)法进行聚类,分析其亲缘关系,以便为今后无患子Sapindus mukorossi优质种质资源的保存、选育和后续研究提供参考。试验结果表明:12个样本共扩增出253个带型,其中191条为多态性条带,多态性百分率为75.16%,样本的平均遗传相似系数为0.739,显示出无患子种内的相似度较高,遗传分化不明显。聚类分析表明:印度南的种源与其他种源产生较大的遗传分异,而印度北的种源由于地理纬度与中国国内种源较为相近,因此,和中国国内种源的遗传距离没有印度南种源相差那么大,各种源间的遗传距离与地理距离基本服从正相关的关系。图2表3参12
植物学;无患子;地理种源;相关序列扩增多态性(SRAP);亲缘关系;分子多态性
无患子Sapindus mukorossi主要分布于中国淮河以南各地,印度和日本也有分布,是一种经济价值很高的树种,其果可以提取皂苷制作肥皂等清洁用品,同时也能用作生产农药的原料;其树形优美,可吸收汽车尾气、二氧化硫等有害气体,是一种良好的行道树种;另外,它根系发达,在水土保持方面也有很大的利用价值。目前,国内未见通过分子手段揭示无患子不同种源之间遗传差异的研究,国外Mahar等[1]通过提取无患子DNA,采用随机扩增多态性DNA(RAPD)标记、DAMD(directed amplification of minisatellite region DNA)、简单重复间序列(inter-simple sequence repeat,ISSR)标记分析了印度5个不同种源地的遗传多样性,但在相关序列扩增多态性(sequence-related amplified polymorphism,SRAP)分子标记的运用上还存在着很大的空白。SRAP分子标记技术具有操作方便快捷、预先不用知道DNA序列的优点,并已在多种经济树种的种质资源研究中得到运用[2-4]。因此,本研究运用SRAP分子标记技术对国内的10个地理种源和印度的2个地理种源为对象进行多态性和亲缘关系的分析,为无患子优质种质资源的保存、亲缘关系的判断、育种方案的制定等提供参考依据。
1 材料和方法
1.1 供试材料
供试材料是采于福建省顺昌县无患子种源收集区的12个种源材料,取3株·种源-1,选择刚展开的幼叶,用硅胶干燥法保存带回实验室,置于-80℃冰箱备用提取DNA。种源涵盖了中国浙江省、江西省、广西省和福建省,及印度南北。
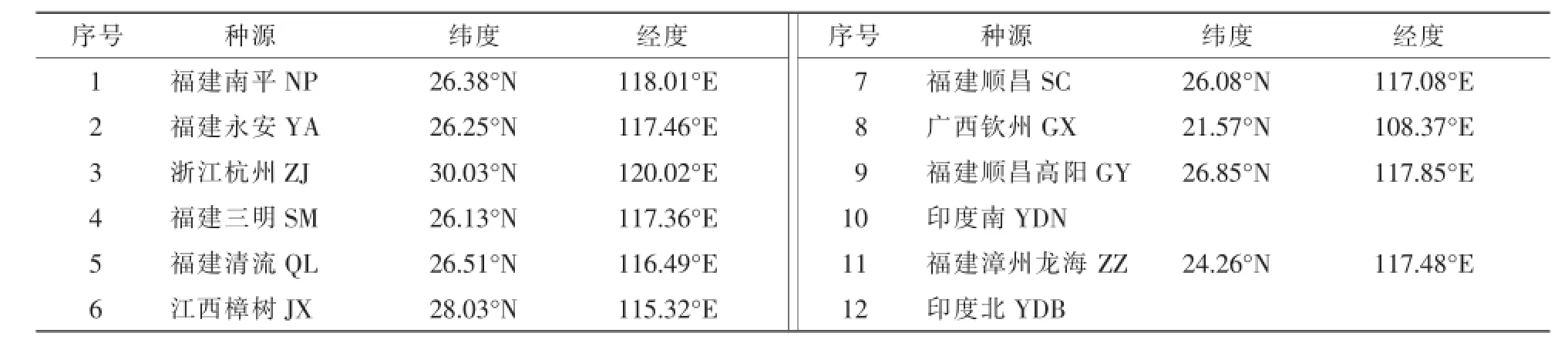
表1 无患子种源地理位置Table 1 Geographic positions of Sapindus mukorossi provenances
1.2 总DNA的提取与测定
采用改良的十六烷基三甲基溴化铵(CTAB)法提取DNA[5-6],用核酸蛋白分析仪测定提取的DNA浓度和纯度,用电泳法检测DNA的完整性,放入-20℃下存储备用。
1.3 PCR扩增、条件优化和检测
SRAP的引物购自上海生工生物工程技术服务有限公司。扩增反应在德国Eppendorf PCR基因扩增仪上进行。采用优化后的反应体系,20 μL体积中,含2.0 mmol·L-1Mg2+,0.2 mmol·L-1dNTPs,0.4 mmol·L-1primers,0.5×16.67 nkat TaqDNA酶,50 ng DNA。
SRAP-PCR扩增程序为:94℃预变性5 min;然后94℃变性1 min,35℃退火1 min,72℃延伸1 min共5个循环;再94℃变性1 min,50℃退火1 min,72℃延伸1 min,共35个循环;最后72℃后延伸10 min。PCR产物在质量分数为1%的琼脂糖凝胶上电泳分离后放入溴化乙锭(EB)中染色,然后用荧光/可见光数字成像分析系统(Alphalmager HP)拍照观察并保存。
1.4 引物筛选
以印度北和浙江杭州的DNA为模板链进行引物筛选,从88对SRAP引物中筛选出扩增条带清晰、带较多多态性较好的引物18对,用于12个无患子样品的SRAP分析。引物组合扩增情况见表2。
1.5 数据统计和分析
根据SRAP扩增产物相同位置清晰可辨的电泳带的有无为统计对象,有记为“1”,无记为“0”,在Excel表格上建立0,1矩阵图,用NTSYS-pc统计分析软件计算Dice遗传相似系数,并进行非加权组平均法(unweighted pair-group method with arithmetic means,UPGMA)聚类分析。相似性系数=(2×样品a和b共有的相同位置数)/(a样品的位置数+b样品的位置数)。

表2 引物组合扩增情况Table 2 Results of amplification
2 结果和分析
2.1 多态性分析
从88对SRAP引物中筛选出扩增条带清晰、带较多多态性较好的引物18对(表2),用于12个不同种源无患子样品的SRAP分析,共扩增出253条清晰可用的条带,分子量为100~3 000 bp,个别超过3 000 bp(图1)。其中多态性条带191条,占总条带的75.16%。每对引物扩增出7~18条多态性谱带,平均产生14.06条。条带统计结果表明:对于同一个种源(以印度南为例),不同的引物组合扩增出5(m6e10)~14(m7e5)条条带,条带数目不同,分子量不同,说明不同引物组合在DNA模板链上的结合位点不同扩增出的片段长短不一,显示了多态性。同一对引物(以m6e1为例)对不同种源扩增出的条带为7(印度南)~11(清流)条,且条带位点有很大差异,体现了各种源样本的遗传差别。此外,在所有引物组合显示的条带中,总有1~2个位点上有特别明亮的特征带型是大部分种源共有的,显示了种源间的相似性。

图1 m6e1为引物的扩增电泳图Figure 1 SRAP map of Sapindus mukorossiprovenance genome amplified with primer pair m6e1
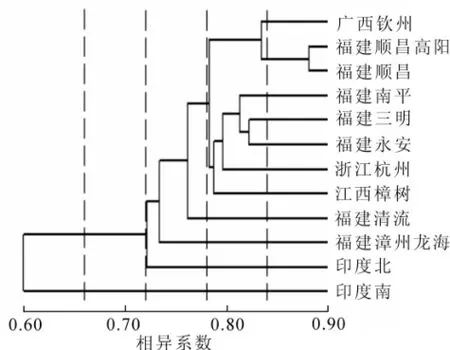
图2 无患子12个种源地的聚类图Figure 2 Dendrogram of 12 Sapindus mukorossi provenances
2.2 无患子地理种源间亲缘关系的聚类分析
根据SRAP结果计算出Dice遗传相似系数(表3),可以看出相似性最高的是福建顺昌和福建顺昌高阳的2个种源,为0.881;最低的是印度南和浙江杭州及印度南和永安,都为0.585。无患子种源间的平均相似系数为0.739,显示出无患子种源之间遗传距离较近。运用UPGMA法对12个种源进行聚类分析,得到聚类图(图2)。可以得出当相似系数以0.600为界时,印度南和其他种源地区分开;以0.720为界时,印度北和其他的种源地区分开;以0.735为界时,区分出福建漳州龙海和余下的种源;以0.760为界时又将福建清流种源分出来。而再往后,相似系数在0.780以后分为2类,第1组为广西钦州、福建顺昌高阳和福建顺昌,第2组为福建南平、福建永安、福建三明、浙江杭州、江西樟树。第1组中,距离最接近的福建顺昌和福建顺昌高阳聚为一类,与广西钦州区分开来。第2组中,地理位置最接近的福建三明和福建永安聚为一类后,与福建南平在0.810处相聚,然后此三者在0.795处和浙江杭州聚为一类,最终在0.788处和江西樟树聚为一类。从聚类结果不难看出,印度南种源有明显的遗传分异,而印度北因与国内的种源在纬度上较为接近,生长的光照、热量等条件较接近,因此,分化不太大。国内的种源间的亲缘关系也大致和地理距离呈正相关,但广西钦州、浙江杭州2处种源的亲缘关系规律就和以上趋势有较大出入。
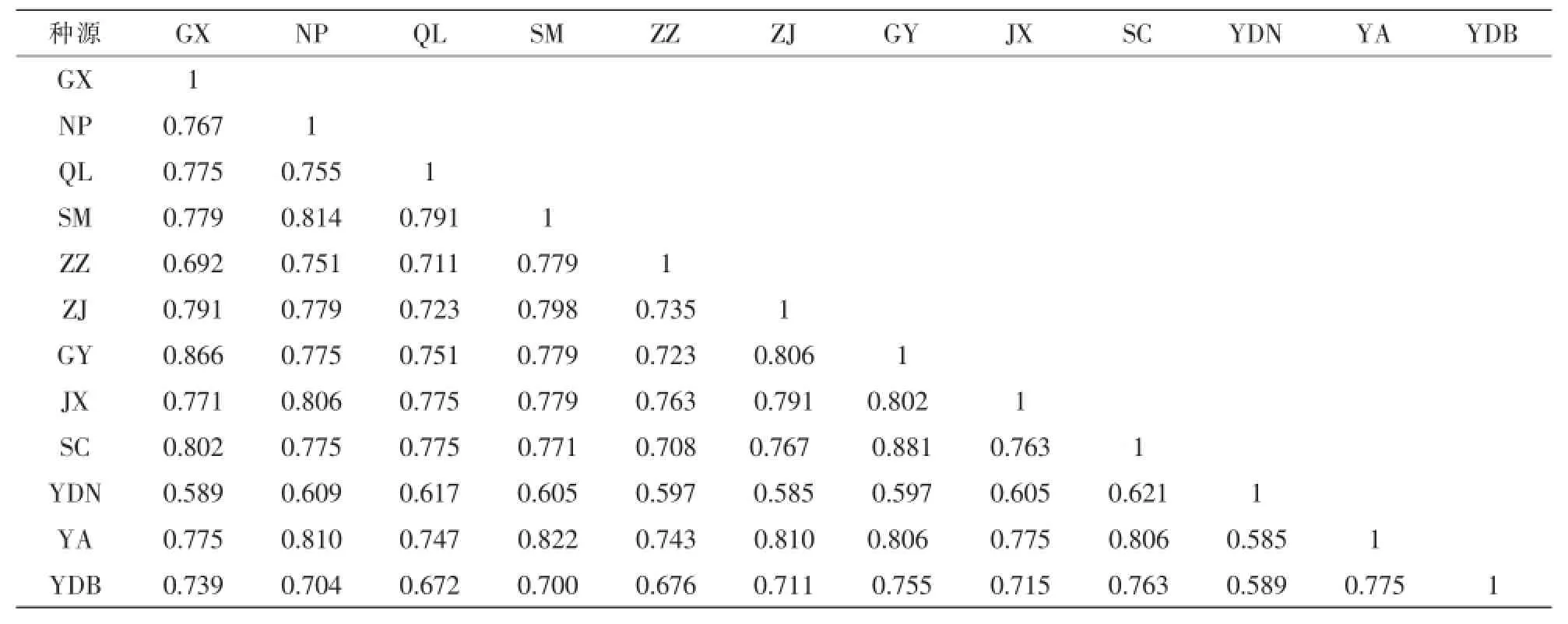
表3 各种源材料的相似系数Table 3 Similarity coefficient of 12 Sapindus mukorossi provenances
3 讨论
3.1 无患子地理种源的遗传多样性水平
一部分植物种[4,7-9]使用SRAP标记的材料出现了很高的多态性,多态条带占90%以上,验证了SRAP标记的高多态性;还有一部分如甘蓝Brassica oleraceavar.capitata[8],旱芹Apium graveolens[10]的多态性分别为51.7%,39.0%,则显示的多态性较低。而本试验所研究的无患子不同引物组合扩增结果的平均多态性为75.2%,多态性一般。由此可见,SRAP的扩增多态性在不同种之间是有差异的。另外,根据Wang等[10]对旱芹的研究,在简单重复序列(simple sequence repeat,SSR)和SRAP对比的情况下,SSR的平均多态性是49.2%,高于SRAP的39.0%也证明了SRAP标记不是在任何情况下都能呈现最大的多态性。
在本试验中,不同引物扩增出的多态率为50.00%~92.31%,显示了不同引物组合的多态性,说明无患子的遗传多样性在其DNA不同位置的片段上体现的程度有较大差异,这一点在Kamalesh等[1]对69个无患子材料所用的RAPD(不同引物的多态性为100%~46%),DAMD(100%~70%),ISSR(100%~60%)标记上也有体现。因此可以对无患子分子标记的引物选择做更深入的探究。
3.2 无患子地理种源的遗传相似性的聚类分析
种源间的平均相似系数为0.739,显示出无患子的种内变异很小,这与Kamalesh等[1]对印度的无患子种源进行多样性分析得到的结果相符。Kamalesh等推测原因是由于无患子树为雌雄同株,并主要靠虫媒传粉,当植株较大时,因为传粉昆虫(主要为蜜蜂Apis)会在同一植株体上来回停留,使得雌雄配子均来自同一植株,造成子代遗传分异小。
很多研究表明[10-12]遗传距离与地理距离有一定的正相关性。从聚类图来看,最初,种源地被很明显的分为2支,印度南的种源单独一支。因为印度南的种源是与所有种源地相隔最远的种源,与其他种源之间的异质性最大,因而产生了最大的遗传分异。而印度北的种源和印度南的种源相比,印度北种源和国内种源的遗传距离没有印度南种源大,除了树种本身的遗传特性外,还有可能是印度北种源地和国内种源地的纬度更为接近,如光照、温度这些对植物生长影响较大的生态因子差别不大。根据无患子种源地的聚类图,可将除印度南之外的种源全都归为一群,而群内的遗传距离与地理距离基本服从正相关的关系。个别特殊,如漳州龙海和永安的聚类结果可能是历史上曾发生过引种或地质变迁等,或者是种源相似系数太接近,以至于微小的试验误差就能引起聚类结果的不同。就后一种可能情况而言,在绘制聚类图时,可以将每对引物扩增得到的结果进行逐步累积,直到聚类关系基本稳定。但关于稳定的范围还有待进一步研究确定。
从研究结果看,无患子的种内分化较小,在进行无患子的种质资源保护和繁育时应注意聚集隔离度较高的不同种源地的种源进行繁殖,以获得更具遗传多样性的无患子后代。
[1]MAHAR K S,RANA T S,RANADE S A.Molecular analyses of genetic variability in soap nut(Sapindus mukorossi)[J].Ind Crops Prod,2011,34(1):1111-1118.
[2]乔燕春,林顺权,刘成明,等.SRAP分析体系的优化及在枇杷种质资源研究上的应用[J].果树学报,2008,25(3):348-352.
QIAO Yanchun,LIN Shunquan,LIU Chengming,et al.Optimization of SRAP analysis and its application in germplasm research of loquat(Eriobotrya japonica)[J].J Fruit Sci,2008,25(3):348-352.
[3]骈瑞琪,倪慧群,欧阳昆唏,等.SRAP体系优化及在胡枝子亲缘关系中的应用[J].华南农业大学学报,2011,32(3):63-67.
PIAN Ruiqi,NI Huiqun,OUYANG Kunxi,et al.Optimization of SRAP system and its preliminary application in genetic relationship analysis ofLespedeza[J].J South China Agric Univ,2011,32(3):63-67.
[4]李莉,彭建营,白瑞霞.中国枣属植物亲缘关系的SRAP分析[J].园艺学报,2009,36(4):475-480.
LI Li,PENG Jianying,BAI Ruixia.Studies on the phylogenetic relationships of ChineseZiziphusby SRAP technique[J].Acta Hortic Sin,2009,36(4):475-480.
[5]刘成明,梅曼彤.无患子科果树植物DNA的高效提取及RAPD分析体系的优化[C]//中国园艺学会.中国园艺学会第5届青年学术讨论会论文集:园艺学进展第5辑.广州:华南农业大学,2002:297-302.
[6]张铁,李洪超,刘伟.文山野生金线莲的RAPD鉴别[J].中草药,2009,32(3):347-348.
ZHANG Tie,LI hongchao,LIU Wei.Identification of wildAnoectochilus roxburghiiof Wenshan using RAPD[J].Chin Trad Herb Drugs,2009,32(3):347-348.
[7]昝逢刚,吴转娣,曾淇,等.荔枝种质遗传多样性的SRAP分析[J].分子植物育种,2009,7(3):562-568.
ZAN Fenggang,WU Zhuandi,ZENG Qi,et al.Genetic diversity analysis ofLitchi germplasmby SRAP markers[J].Mol Plant Breed,2009,7(3):562-568.
[8]缪体云,薄天岳,陈锦秀,等.甘蓝种质资源遗传多样性的SRAP分析[J].分子植物育种,2010,8(1):94-98.
MIAO Tiyun,BO Tianyue,CHEN Jinxiu,et al.Genetic diversity of cabbage germplasm revealed by SRAP analysis[J].Mol Plant Breed,2010,8(1):94-98.
[9]李严,张春庆.西瓜杂交种遗传多态性的SRAP标记分析[J].园艺学报,2005,32(4):643-647.
LI Yan,ZHANG Chunqing.Studies on genetic diversity with a molecular marker SARP of watermelon hybrids[J].Acta Hortic Sin,2005,32(4):643-647.
[10]WANG Shuai,YANG Wencai,SHEN Huolin.Genetic diversity inApium graveolensand related species revealed by SRAP and SSR markers[J].Sci Hortic,2011,129:1-8.
[11]王铁娟,杨持,马静,等.籽蒿的地理分布与遗传分化[J].植物生态学报,2005,29(1):122-127.
WANG Tiejuan,YANG Chi,MA Jing,et al.Geographic distribution and genetic differentiation ofArtemisia sphaerocephala[J].Acta Phytoecol Sin,2005,29(1):122-127.
[12]杨玉玲,马祥庆,张木清.不同地理种源杉木的分子多态性分析[J].热带亚热带植物学报,2009,17(2):183-189.
YANG Yuling,MA Xiangqing,ZHANG Muqing.Molecular polymorphic analysis for different geographic provenances of Chinese fir[J].J Tropic Subtropic Bot,2009,17(2):183-189.
Molecular polymorphic analysis with geographic provenances of Sapindus mukorossi
LIU Bao1,FAN Huihua2,PENG Zhuqing1,LIU Yan1,CHEN Cunji1
(Forestry College,Fujian Agriculture and Forestry University,Fuzhou 350002,Fujian,China;2.Fujian Research Institute of Forestry,Fuzhou 350012,Fujian,China)
To determine reference material for selective breeding ofSapindus mukorossi,genome DNA of 12 accessions,which were collected from different regions of India and China,were amplified with the sequencerelated amplified polymorphism(SRAP)method using 18 primer pairs.Then,the cumulative band data generated from SRAP was used to calculate the Dice genetic similarity coefficient which was analyzed by the Unweighted Pair Group Method with Arithmetic Mean(UPGMA)to detect their mutual relationships.Genetic diversity was determined with a cluster analysis.Results showed that 191 of the total 253 generated bands were polymorphic(75.16%)with a mean coefficient of 0.739.The cluster analysis showed that the provenance in southern India had greater genetic diversity than other provenances.Compared to the provenance of southern India,less genetic diversity was noted for provenances of northern India and Chinese provenances where geographic latitudes were similar.Positive relationships occurred between genetic distance and geographic distance in 12 geographic provenances ofS.mukorossi.Overall,the results showed a low level of diversity in this species.[Ch,2 fig.3 tab.12 ref.]
botany;Sapindus mukorossi;geographic provenances;sequence-related amplified polymorphism(SRAP);relationship;molecular polymorphism
S686;Q943
A
2095-0756(2014)01-0151-05
10.11833/j.issn.2095-0756.2014.01.023
2013-03-05;
2013-06-21
国家林业公益性行业科研专项(201104100)
刘宝,讲师,博士研究生,从事森林培育学研究。E-mail:40511779@qq.com

