溶藻弧菌Ⅲ型分泌系统分子伴侣护航蛋白VscO的基因克隆及其生物信息学分析
庞欢瑛周泽军丁燏黄郁葱吴灶和简纪常
(1.广东海洋大学水产学院,湛江 524088;2.广东省水产经济动物病原生物学及流行病学重点实验室,湛江 524088;3.广东省教育厅水产经济动物病害控制重点实验室,湛江 524088;4.仲恺农业工程学院,广州 510225)
溶藻弧菌Ⅲ型分泌系统分子伴侣护航蛋白VscO的基因克隆及其生物信息学分析
庞欢瑛1,2,3周泽军1,2,3丁燏1,2,3黄郁葱1,2,3吴灶和2,3,4简纪常1,2,3
(1.广东海洋大学水产学院,湛江 524088;2.广东省水产经济动物病原生物学及流行病学重点实验室,湛江 524088;3.广东省教育厅水产经济动物病害控制重点实验室,湛江 524088;4.仲恺农业工程学院,广州 510225)
克隆了溶藻弧菌(Vibrio alginolyticus)ZJ03株Ⅲ型分泌系统(T3SS)分子伴侣护航蛋白(Chaperone escort protein)vscO基因,并对其进行生物信息学分析。结果表明,vscO基因全长462 bp,编码153个氨基酸,理论分子量约为18.43 kD,pI值为9.22。细胞定位分析显示vscO基因位于外周质,不存在信号肽,无跨膜区。vscO基因具有转录起始区-35区和-10区,以及翻译识别信号SD序列(Shine-Dalgarno sequence)。该氨基酸序列含有多种活性位点,如蛋白激酶C磷酸化位点等。系统进化树发现溶藻弧菌的VscO蛋白与副溶血弧菌聚为同一亚族。VscO亚基三维结构模型显示其与副溶血弧菌T3SS 的YscO蛋白有相似构型。信号通路分析推测VscO位于T3SS的针状样结构上。蛋白网络互作图谱发现VscO与10种T3SS蛋白具有相邻关系。本研究结果将为溶藻弧菌Ⅲ型分泌系统护航机制的研究奠定基础。
溶藻弧菌 Ⅲ型分泌系统(T3SS) 分子伴侣护航 vscO基因
溶藻弧菌(Vibrio alginolyticus)是一种嗜盐嗜温性革兰氏阴性短杆菌,广泛存在于海水中,是水生动物弧菌病的主要病原菌之一,能引起鱼、虾、贝等多种水产动物爆发大规模弧菌传染病[1-3],给水产养殖业造成巨大经济损失。此外,该菌可导致人类的多种疾病,如腹泻、中耳炎以及败血症等[4,5],严重危害人类健康。
III型分泌系统(Type III secretion system,T3SS)是细菌保守存在的“针状样”毒力装置,与细菌的致病机制密切相关[6,7],是病原菌研究的热点之一。该装置通过输出通路(Export pathway)将效应蛋白注入宿主细胞,进一步诱导宿主细胞死亡[8,9]。研究发现某些细菌的T3SS输出通路中存在分子伴侣护航机制(Chaperone escort mechanism),即护航蛋白和分子伴侣蛋白将效应蛋白护送至胞外的机制,该机制在T3SS致病过程中起重要作用[10]。
本研究选取溶藻弧菌T3SS中分子伴侣护航蛋白VscO进行研究,对其进行基因克隆测序,并对其序列特征、信号通路、三维结构、蛋白网络互作等进行具体分析,以期为溶藻弧菌T3SS护航机制的研究奠定基础。
1 材料与方法
1.1 材料
1.1.1 菌株和载体 溶藻弧菌强毒株ZJ03,由本试验室从广东省湛江港海域患病红笛鲷鱼体中分离并保存[11];克隆载体pMD18-T购自TaKaRa公司。
1.1.2 主要试剂 ExTaqDNA聚合酶购自TaKaRa公司;细菌基因组DNA提取试剂盒、DNA 胶回收试剂盒购自天根公司;其余试剂均为进口或国产分析纯;引物合成和DNA测序均由上海生工生物技术服务有限公司完成。抗生素浓度为氨苄西林(Ap)100 μg/mL。
1.2 方法
1.2.1 溶藻弧菌ZJ03总DNA的提取 将溶藻弧菌接种于TSB(2% NaCl)培养基,28℃振荡培养18 h。取1 mL菌液于离心管中,10 000 r/min离心5 min收集菌体,参照试剂盒说明书提取细菌基因组DNA,-20℃保存备用。
1.2.2vscO基因的克隆 根据GenBank登录的溶藻弧菌全基因序列(Accession:NZ_AAPS00000000)设计一对引物。上游引物VscO1为:ATGATAGAACGTTTATTAGA,下游引物VscO2为:TTAGATAATGTCGACAGTG。以溶藻弧菌ZJ03总DNA为模板,PCR反应条件为:94℃预变性4 min;94℃ 30 s,50℃ 30 s,72℃ 30 s,共35个循环,72℃延伸 10 min。PCR产物经1%琼脂糖凝胶电泳检验后用DNA胶回收试剂盒切胶回收。
1.2.3 PCR产物的测序 按照说明书操作步骤,将PCR产物与pMD18-T载体连接,然后转化大肠杆菌DH5α感受态细胞,在含IPTG/X-Gal的Ap+/LB平板上进行筛选,挑取阳性克隆送上海生工测序。
1.2.4 溶藻弧菌ZJ03 VscO生物信息学分析 采用NCBI(http://blast.ncbi.nlm.nih.gov/Blast.cgi)进行序列同源性比对和相似性分析;使用DNAMAN进行氨基酸同源比对分析;运用ExPASy Proteomics Server(http://ca.expasy.org)推导氨基酸序列、确定开放阅读框(ORF)、分子量计算值(Mw)和理论等电点(pI)预测等;通过SignalP 4.0 Server(http://www.cbs.dtu.dk/services/SignalP)预测信号肽序列;应用TMHMM Server 2.0(http://www.cbs.dtu. dk/services/TMHMM)预测跨膜结构域;SoftBerry-Psite(http://linux1.softberry.com/berry.phtml? Topic = psite&group= programs & subgroup = proloc)预测氨基酸序列中的功能位点分布;使用Promoter 2.0软件分析启动子。PSORT II Prediction(http://psort.hgc. jp/ form2.html)预测亚细胞定位。使用Clastal 2.0及MEGA 5.0软件,以邻位相连法(neighbor-joining)构建系统进化树。利用SWISS-MODEL(http://www. swissmodel. expasy.org/)程序进行三维结构构建[12]。Kegg软件分析涉及的通路(http://www.genome. jp/kegg/)[13]。STRING数据库搜索蛋白网络互作(http://string.embl.de/)[14]。
2 结果
2.1vscO基因的全长克隆
PCR扩增得到一条约500 bp的特异条带(图1-A),将扩增产物连接到pMD18-T载体,菌落PCR扩增出约500 bp的条带(图1-B)。测序表明,vscO基因的开放阅读框(ORF)为462 bp,编
码153个氨基酸(图2),在GenBank上的登录号为KJ179947。

图1 vscO基因的克隆(A)和菌落PCR(B)
2.2 VscO的理化性质
利用ExPASy软件对溶藻弧菌ZJ03 VscO蛋白进行分析,结果显示其原子总数为2 631,分子结构式为C799H1334N248O249S1。理论分子量为18.430 kD,理论pI值为9.22。不稳定系数66.17,脂肪系数为81.05,总平均亲水性为-1.184,蛋白总体亲水性较高。该蛋白不含有甘氨酸(Gly)、脯氨酸(Pro)和半胱氨酸(Cys),在280 nm处的摩尔消光系数为9,970(mol·cm-1)。酸性氨基酸残基(Asp + Glu)总数为32个,碱性氨基酸(Arg + Lys)总数为36个,N末端是甲硫氨酸(Met)。在酵母和大肠杆菌中表达的半衰期分别大于20 h和10 h,在哺乳动物网织红细胞中体外培养表达的半衰期为30 h。
2.3 VscO的序列分析
应用Blast程序搜索发现,vscO基因在染色体上的相对排列顺序如图3所示,箭头表示转录方向。根据其他细菌的研究结果推测SycN是VopN的分子伴侣,TyeA是调节蛋白,VopN是T3SS的效应蛋白,VscN是T3SS中ATP酶,VscP是控制T3SS针筒长度的“尺子”,VscQ可以形成T3SS的内膜基座[15,16]。
利用SignalP 4.0 Server程序对VscO氨基酸序列进行N端信号肽结构的预测,发现其无明显的信号肽切割位点,不存在信号肽。TMHMM Server 2.0程序预测发现该蛋白无跨膜区。SoftBerry-Psite程序预测发现该氨基酸序列含有2个蛋白激酶C磷酸化位点(71-73 aa,93-95 aa),1个酪蛋白激酶II磷酸化位点(40-43 aa),1个酪氨酸激酶磷酸化位点(132-138 aa),1个内质网靶信号位点(139-142 aa),1个微体C末端靶信号位点(57-59 aa)(图2)。Promoter 2.0软件分析发现vscO基因有转录起始区-35区和-10区,以及翻译识别信号SD序列(Shine-Dalgarno sequence)(图2)。蛋白质亚细胞定位预测结果显示,VscO位于细胞核中可能性最大,为56.5%,其次为线粒体,可能性为21.7%,而位于线粒体和细胞支架的可能性分别为17.4%和4.3%。

图2 vscO基因核苷酸及其编码的氨基酸序列

图3 vscO基因在染色体相对排列顺序
2.4 VscO的同源性及进化分析
BLAST分析发现溶藻弧菌VscO与其他弧菌的VscO具有较高的同源性,其中与副溶血弧菌的VscO氨基酸序列的同源性最高达86%,与塔斯曼弧菌(Vibrio tasmaniensis)的同源性为72%,与塔式弧菌(Vibrio tubiashii)的同源性为71%,与Vibrio caribbenthicus的同源性为70%,与Vibrio azureus的
同源性最低为65%。氨基酸序列同源性比较(图4)表明VscO在弧菌中是相对保守的。利用Neighborjoining法,将推导的VscO氨基酸序列与其他微生物构建系统进化树(图5)发现,溶藻弧菌的VscO蛋白可明显地与副溶血弧菌聚为同一亚族,表明它们之间有较近亲缘关系,这与传统的形态学和生化特征分类结果一致。

图4 vscO基因推导的氨基酸序列与其他物种同源性比较

图5 VscO系统进化树
2.5 VscO的三维结构
将VscO氨基酸序列提交至SWISS-MODEL 程序,自动搜索同源蛋白作模板,得到VscO单亚基三级结构模型。结果见图6-A,与其同源蛋白副溶血弧菌 YscO非常相似(图 6-B),都具有两个α-螺旋,不存在β-折叠,在两个α-螺旋之间含有一个转角。两者的差别是YscO的第一个α-螺旋的长度比VscO略长。
2.6 VscO的信号通路分析
采用Kegg 软件对VscO的信号通路进行预测分析,以细菌T3SS的信号通路为模板绘制溶藻弧菌VscO信号通路,见图7。由图推测VscO位于Ⅲ型分泌系统的“针状样”结构上与VscP和VscX相邻。
2.7 VscO蛋白的网络互作分析
应用STRING数据库搜索VscO与其他T3SS蛋白的网络互作,结果见图8。目前已知vscO与vopD、vscP、vopB、vscS、vscN、vscT具有同时生成
(cooccurrence)、相邻(neighborhood)的关系,其他关系还有待研究(textmining),而与vscQ、vscR、vscX仅有相邻的关系,它们的其他关系还有待挖掘,下一步将对这些基因进行克隆和序列分析,初步了解它们的功能。

图6 溶藻弧菌ZJ03 VscO(A)与副溶血弧菌 YscO(B)三维结构比较

图7 VscO蛋白的信号通路图
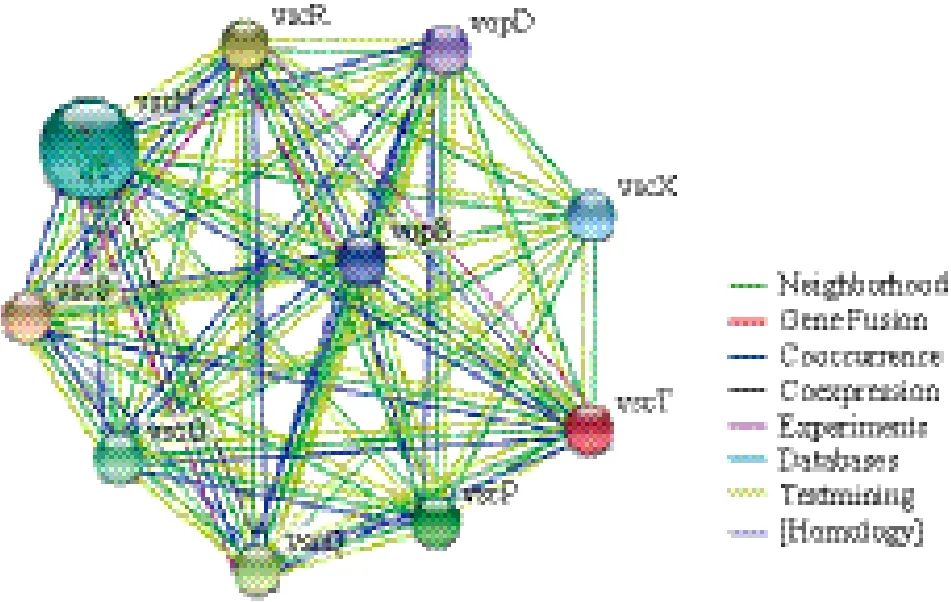
图8 VscO蛋白与其他T3SS蛋白的网络互作图
3 讨论
Ⅲ型分泌系统是溶藻弧菌重要的毒力因子之一,与细菌的致病性密切相关。病原菌黏附到宿主细胞表面后,通过T3SS直接将特异性效应蛋白注入宿主细胞内,改变宿主的各种生理功能,破坏宿主的组织结构,达到能在宿主体内生存和繁殖的目的[17]。
T3SS分子伴侣护航蛋白是最近鉴定的一种T3SS功能蛋白,其作用一方面是护航分子伴侣到相应的位置,使分子伴侣以正确的方式与效应蛋白结合;另一方面,在护航结束后,重新将分子伴侣释放出来以便下一轮的循环,从而保证分子伴侣的重复利用和效应蛋白的输出效率[18]。分子伴侣护航蛋白与分子伴侣结合,作用是稳定分子伴侣性质和重复利用分子伴侣[19]。
目前,对T3SS分子伴侣蛋白的研究不多,只报道了少数几种。例如:耶尔森氏菌的SycD[20],福氏志贺氏菌的IpgC[21]和Spa13[22],鼠伤寒沙门氏菌(Salmonella typhimurium)的FliJ和InvI,耶尔森菌的YscO均为此类蛋白[10]。小肠结肠炎耶尔森氏菌(Yersinia enterocolitica)的YscO是分子伴侣SycD的护航蛋白,其功能是护航SycD分子伴侣,帮助转位子蛋白YopB和YopD参与到宿主细胞膜上T3SS针孔的形成[23]。鼠伤寒沙门氏菌(Salmonella typhimurium)的FliJ,它是鞭毛分子伴侣护航蛋白,功能是将空载的分子伴侣转运给小纤维丝帽类亚基和鞭毛钩,使得鞭毛顺利组装[18]。InvI也是鼠伤寒沙门氏菌中分子伴侣SicA的护航蛋白。经过InvI的护航,分子伴侣SicA帮助其效应蛋白SipC通过T3SS的分泌,参与到宿主细胞膜上T3SS针孔的形成[10]。
目前,虽然溶藻弧菌T3SS护航蛋白已有少量研
究,但主要集中在其免疫功能上[24],有关其详细的生物信息学研究仍鲜见报道。本研究对T3SS系统中分子伴侣护航蛋白VscO进行克隆和生物信息学分析。结果表明,vscO基因全长462 bp,编码153个氨基酸,理论分子量为18.43 kD,理论pI值为9.22。BLAST分析发现溶藻弧菌VscO与副溶血弧菌(V.parahaemolyticus)VscO氨基酸序列的同源性最高达86%。信号通路分析推测VscO位于T3SS的“针状样”结构上,推测其主要功能是是分子伴侣的护航蛋白,但是其与分子伴侣的互作区域目前尚未清楚。我们下一步将对VscO进行基因敲除,然后对野生株和缺失株的生理生化特性进行分析,再应用蛋白质组学技术分析缺失株和野生株的胞外分泌蛋白,从中筛选VscO相关的分子伴侣和效应蛋白,并明确VscO与分子伴侣的互作区域,为阐明T3SS的护航机制奠定基础。
4 结论
本研究从溶藻弧菌ZJ03成功克隆T3SS系统中分子伴侣护航蛋白VscO的全基因序列。该基因全长462 bp,编码153个氨基酸,理论分子量为18.43 kD,理论pI值为9.22。VscO位于外周质,无信号肽和跨膜区。该氨基酸序列含有2个蛋白激酶C磷酸化位点,1个酪蛋白激酶II磷酸化位点,1个酪氨酸激酶磷酸化位点,1个内质网靶信号位点,1个微体C末端靶信号位点。vscO基因具-35和-10转录起始区,以及翻译识别信号SD序列(Shine-Dalgarno sequence)。同源性分析表明与副溶血弧菌亲缘关系较近。亚基三维结构模型显示其与副溶血弧菌YscO蛋白有相似构型。信号通路分析推测其位于T3SS的针状样结构上。蛋白网络互作图谱表明VscO与10种T3SS蛋白具有相邻关系。
[1]Sadok K, Mejdi S, Nourhen S, et al. Phenotypic characterization and RAPD fingerprinting ofVibrio parahaemolyticusandVibrio alginolyticusisolated during Tunisian fish farm outbreaks[J]. Folia Microbiologica(Praha), 2013, 58(1):17-26.
[2]Chang CC, Yeh MS, Lin HK, et al. The effect ofVibrio alginolyticusinfection on caspase-3 expression and activity in white shrimpLitopenaeus vannamei[J]. Fish and Shellfish Immunology, 2008, 25(5):672-678.
[3]Ben Kahla-Nakbi A, Chaieb K, Bakhrouf A. Investigation of several virulence properties amongVibrio alginolyticusstrains isolated from diseased cultured fish in Tunisia[J]. Diseases of Aquatic Organisms, 2009, 86(1):21-28.
[4]Campanelli A, Sanchez-Politta S, Saurat JH. Cutaneous ulceration after an octopus bite:infection due toVibrio alginolyticus, an emerging pathogen[J]. Ann Dermatol Venereol, 2008, 135(3):225-227.
[5]Sganga G, Cozza V, Spanu T, et al. Global climate change and wound care:case study of an off-seasonVibrio alginolyticusinfection in a healthy man[J].Ostomy Wound Manage, 2009, 55(4):60-62.
[6]Zhao Z, Zhang L, Ren C, et al. Autophagy is induced by the type III secretion system ofVibrio alginolyticusin several mammalian cell lines[J]. Archives of Microbiology, 2011, 193(1):53-61.
[7]Caron E, Crepin VF, Simpson N, et al. Subversion of actin dynamics by EPEC and EHEC[J]. Current Opinion in Microbiology, 2006, 9(1):40-45.
[8]Zhao Z, Chen C, Hu CQ, et al. The type III secretion system ofVibrio alginolyticusinduces rapid apoptosis, cell rounding and osmotic lysis of fish cells[J]. Microbiology, 2010, 156(9):2864-2872.
[9]Burdette DL, Seemann J, Orth K.VibrioVopQ induces PI3-kinaseindependent autophagy and antagonizes phagocytosis[J]. Molecular Microbiology, 2009, 73(4):639-649.
[10] Evans LD, Hughes C. Selective binding of virulence type III export chaperones by FliJ escort orthologues InvI and YscO[J]. FEMS Microbiology Letters, 2009, 293(2):292-297.
[11] Cai SH, Lu YS, Wu ZH, et al. Loop-mediated isothermal amplification method for rapid detection ofVibrio alginolyticus, the causative agent of vibriosis in mariculture fish[J]. Letters in Applied Microbiology, 2010, 50(5):480-485.
[12] Schwede T, Kopp J, Guex N, et al. SWISS-MODEL:an automated protein homology- modeling server[J]. Nucleic Acids Research, 2003, 31(13):3381-3385.
[13] Xia Z, Chen W, Chang C, et al. A novel network and sparsity constraint regression model for functional module identification in genomic data analysis[J]. International Journal of Data Mining and Bioinformatics, 2013, 8(3):311-325.
[14] Szklarczyk D, Franceschini A, Kuhn M, et al. The STRING
database in 2011:functional interaction networks of proteins, globally integrated and scored[J]. Nucleic Acids Research, 2011, 39(Database issue):561-568.
[15] Ghosh P. Process of protein transport by the type III secretion system[J]. Microbiology and Molecular Biology Reviews, 2004, 68(4):771-795.
[16] Francis M S. Type III secretion chaperones:a molecular toolkit for all occasions[J]. Handbook of Molecular Chaperones:Roles, Structures and Mechanisms, 2010, 79-148.
[17] Li W, Liu Y, Sheng X, et al. Structure and mechanism of a type III secretion protease, NleC[J]. Acta Crystallographica Section D, 2014, 70(1):40-47.
[18] Evans LD, Stafford GP, Ahmed S, et al. An escort mechanism for cycling of export chaperones during flagellum assembly[J]. Proceedings of the National Academy of Sciences, 2006, 103(46):17474-17479.
[19] Ibuki T, Uchida Y, Hironaka Y, et al. Interaction between FliJ and FlhA, components of the bacterial flagellar type III export apparatus[J]. Journal of Bacteriology, 2013, 195(3):466-473.
[20] Neyt C, Cornelis GR. Insertion of a Yop translocation pore into the macrophage plasma membrane byYersinia enterocolitica:requirement for translocators YopB and YopD, but not LcrG[J]. Molecular Microbiology, 2002, 33(5):971-981.
[21] Turbyfill KR, Hartman AB, Oaks EV. Isolation and characterization of aShigella flexneriinvasin complex subunit vaccine[J]. Infection and Immunity, 2000, 68(12):6624-6632.
[22] Cherradi Y, Hachani A, Allaoui A. Spa13 ofShigella flexnerihas a dual role:chaperone escort and export gate-activator switch of the type III secretion system[J]. Microbiology, 2014, 160(1):130-141.
[23] Diepold A, Wiesand U, Amstutz M, et al. Assembly of theYersinia injectisome:the missing pieces[J]. Molecular Microbiology, 2012, 85(5):878-892.
[24] Zhou Z, Pang H, Ding Y, et al. VscO, a putative T3SS chaperone escort ofVibrio alginolyticus, contributes to virulence in fish and is a target for vaccine development[J]. Fish and Shellfish Immunology, 2013, 35(5):1523-1531.
(责任编辑 李楠)
Molecular Cloning and Bioinformatics Analysis of T3SS Chaperone Escort Protein VscO from Vibrio alginolyticus
Pang Huanying1,2,3Zhou Zejun1,2,3Ding Yu1,2,3Huan Yucong1,2,3Wu Zaohe2,3,4Jian Jichang1,2,3
(1. Fisheries College of Guangdong Ocean University,Zhanjiang 524088;2. Guangdong Provincial Key Laboratory of Pathogenic Biology and Epidemiology for Aquatic Economic Animals,Zhanjiang 524088;3. Key Laboratory of Diseases Controlling for Aquatic Economic Animals of Guangdong Higher Education Institutions,Zhanjiang 524088;4. Zhongkai University of Agriculture and Engineering,Guangzhou 510225)
Primers for PCR cloning were designed according to the whole genome sequence of Vibrio alginolyticus published in GenBank. The type III secretion system(T3SS)chaperone escort protein vscO gene of V.alginolyticus strain ZJ03 was amplified by PCR and cloned into pMD18-T vector. Sequence analysis revealed that vscO gene is 462 bp and encodes a putative protein of 153 amino acids. The predicted molecular weight(MW)of VscO was 18.43 kD with an estimated pI of 9.22. Using SignalP 4.0 and TMHMM Server 2.0 software, it was predicted that the VscO protein was located in periplasm. It did not contain a signal peptide or a transmembranous region. The vscO gene with transcription initiation region at -35 and -10 region, and a translation recognition signal sequence(SD Shine-Dalgarno sequence). This protein had some active sites, such as phosphorylation sites. To further analyze the evolutionary relationship among VscO, a molecular phylogenetic tree was constructed using Mega 5.0 software. In this tree, the VscO protein showed high genetic relationship with Vibrio parahaemolyticus. The three-dimensional structure of VscO was determined using SWISS-MODEL work-space and it had a similar structure with YscO protein of V. parahaemolyticus. Signaling pathway analysis showed that VscO is located at the “needle” site of T3SS. Protein interaction map network found that the relation between VscO and 10 kinds of T3SS proteins was neighbourhood. These results can provide a basis for further studies on the chaperone escort machanism used by T3SS export pathway of Vibrio species.
Vibrio alginolyticus T3SS Chaperone escort vscO gene
2014-04-16
国家科技支撑计划(2012BAD17B02),广东省自然科学基金项目(S2013040014562)
庞欢瑛,女,博士,讲师,研究方向:水产经济动物病害;E-mail:phying1218@163.com
简纪常,男,博士,教授,研究方向:水产经济动物免疫学及病害控制;E-mail:jianjc@gmail.com

