基因组DNA甲基化的高效毛细管电泳定量检测
张敏 陈倩 张丽娟 权力
·论著·
基因组DNA甲基化的高效毛细管电泳定量检测
张敏 陈倩 张丽娟 权力★
目的建立快速准确的高效毛细管电泳(HPCE)定量基因组DNA甲基化的方法。方法以含有十二烷基磺酸钠(SDS)的碳酸氢钠溶液作为背景电解质溶液,优化运行电压、柱温,对DNA组成单元脱氧核苷酸进行分离定量。结果5种脱氧核糖核苷和5种核糖核苷在10 min内实现基线分离。脱氧胞苷(dC)的标准曲线为Y=172540.15X-6.19,相关系数(R)为0.999,线性范围从2× 10-3mol/L到2×10-1mol/L。5-甲基脱氧胞苷(5 mdC)的标准曲线为Y=170271.74X-46.19,相关系数(R)为0.995,线性范围从1×10-4mol/L到2×10-2mol/L。日内相对标准偏差(RSD%)<5%,日间相对标准偏差(RSD%)<9%。5 mdC和dC的检测限均为1×10-5mol/L。样品的甲基化程度以(5 mdC)含量占总脱氧胞苷(5 mdC+dC)的比率表示。应用本方法分析小牛胸腺DNA的甲基化水平,结果为6.18%,与本研究组所得液质联用(LC-MS/MS)的定量结果(6.77%)和文献报导中应用液相色谱-紫外检测器(HPLC-UV)的检测结果(6.26%)对比,一致性好。结论本研究建立的方法分析速度快,准确性高,稳定性好,灵敏度满足微量样本的检测需求,可以用于临床甲基化诊断。
DNA甲基化;高效毛细管电泳;定量分析
DNA甲基化是重要的表观遗传修饰方式之一,是DNA序列不发生改变的情况下产生的可遗传的表达改变。异常的DNA甲基化模式可能影响许多基因的表达,进而与多种疾病例如神经管畸形[1-2]、肿瘤[3]、系统性红斑狼疮[4]的发生和病情进展相关。定量检测基因组DNA甲基化水平对于肿瘤的诊断治疗和预后,以及对神经管畸形和系统性红斑狼疮等复杂疾病的发病机理和预防、治疗的研究有重要的意义。既往的研究多集中在特定基因的甲基化,对于基因组DNA甲基化的定量研究多采用高效液相色谱法[5-6]。与高效液相色谱相比,毛细管电泳分离度更高,特异性好,对样品量和样品纯度要求低,价格便宜,操作简便,更加适合临床研究。本研究优化了组成DNA和RNA的10种脱氧核糖核苷和核糖核苷的分离条件,建立了可以避免RNA干扰的基因组DNA甲基化的毛细管电泳定量分析方法并将其应用于小牛胸腺DNA的甲基化水平检测。
1 材料与方法
1.1试剂与标准品
5 mdC购自日本TCI公司,其他4种脱氧核糖核苷包括dC、脱氧鸟苷(dG)、脱氧腺苷(dA)、胸腺苷(T)以及5种核糖核苷5-甲基胞苷(5 mC)、胞苷(C)、鸟苷(G)、腺苷(A)、和尿苷(U)以及十二烷基磺酸钠(sodium dodecyl sulfate,SDS)、碳酸氢钠(NaHCO3)、小牛胸腺DNA和用于水解DNA样品的蛇毒磷酸二酯酶购自美国Sigma公司。核酸内切酶Benzonase购自德国Merck公司。碱性磷酸酶购自上海生物工程有限公司。
1.2标准溶液配置
根据各种核苷和脱氧核苷在水中溶解度的大小,dG和G储备液浓度为2×10-3mol/L,dA和A储备液浓度为1×10-2mol/L,其余六种核苷或脱氧核苷储备液均为1×10-1mol/L,所有储备液-20℃冻存,使用时超纯水稀释至所需浓度。配置含60 mmol/L SDS的NaHCO3(48 mmol/L)溶液作为分离缓冲液,用1 mol/L的NaOH调pH至9.6。溶液均以Milli Q过滤的超纯水配制,所有溶液使用前均通过0.22 μm滤膜过滤。
1.3仪器与装置
本方法应用Agilent7100高效毛细管电泳仪,二极管阵列检测器,选用Agilent G1600-61232毛细管柱进行分离(有效长度:56 cm)(美国Aglient公司)。所有的样品称量均在同一台梅特勒XS205天平上进行(瑞士METTLER TOLEDO公司),缓冲溶液PH的调定使用JENWAY 3520 PH计(英国Barloworld Scientific公司),Milli Q纯水净化装置来自法国Biopak公司。
1.4DNA水解
取单双链DNA都敏感的核酸内切酶Benzonase 250 U,蛇毒磷酸二酯酶300 mU,碱性磷酸酶200 U,加至5 mL包含20 mmol/L MgCl2,100 mmol/L NaCl的Tris-HCl缓冲溶液(pH=7.9,20 mmol/L)中,配制成DNA水解酶混合溶液。水解时,1 μg DNA添加50 μL的DNA水解酶混合溶液,37℃水浴培养6小时[7]。水解产物直接进样。
1.5电泳条件优化
分离缓冲体系选择含有60 mmol/L SDS的NaHCO3的溶液(48 mmol/L,pH=9.6),二极管阵列检测器检测波长设为254 nm。在毛细管柱温分别为20、22、25、28℃,电压分别为13、16、18、20、22 KV条件下,对组成DNA的5种脱氧核糖核苷以及包含RNA水解产物在内的10种核苷进行分离,进样方式选用压力50 mbar,时间5 s进样。
1.6校准曲线和灵敏度
配置一系列包含5种脱氧核苷的混合溶液,其中5 mdC的含量分别为1×10-4mol/L,5×10-4mol/L,1×10-3mol/L,5×10-3mol/L,1×10-2mol/L,dC的含量分别为2×10-3mol/L,1×10-2mol/L,2× 10-2mol/L,1×10-1mol/L,2×10-1mol/L。其他3种脱氧核苷dA,dG,T,的含量与dC相同。以采集到的dC和5 mdC峰面积为纵坐标,以浓度为横坐标,分别拟合二者的标准曲线。样品的甲基化水平(5 mdC%)通过计算[5 mdC]/([5 mdC]+[dC])×100%而得。灵敏度用检测限和定量限表示,通过不断减少dC和5 mdC的进样浓度,以信噪比≥3、≥10确定检测限和定量限。
1.7重现性及在小牛胸腺DNA中的应用
用已知5 mdC%为5%、10%、20%、40%的标准溶液考察方法的稳定性和重复性,以一天不同时间段内重复3次进样所测得值的相对标准偏差表示日内重现性,以一周内5天25次进样所测得值的相对标准偏差表示日间重现性。将建立好的方法应用于小牛胸腺DNA,将测得值与其他方法的测定值比对。
2 结果与分析
2.1核苷酸的分离
研究表明,分离缓冲体系为pH=9.6的48 mmol/L NaHCO3(含60 mmol/L SDS)溶液时,筛分介质的筛孔最适合用于分离核苷[8-9]。在该分离体系下,毛细管柱温和电压是影响待测物分离度的两个主要因素。柱温越高分析速度越快,但柱温高于28℃时,待测核苷不能实现基线分离。本文优化了柱温为20,22,25,28℃,电压为13,16,18,20,22 KV的条件下,待测核苷的分离情况。发现在温度22℃,电压为13 KV时,分离度最佳。组成DNA的5种脱氧核糖核苷(5 mdC,dC,dA,dG,T)在10分钟内实现完全的基线分离(图1),包含DNA,RNA组成单元的10种核苷(5 mdC,5 mC,dC,C,dA,A,dG,G,T,U)在13 min内分离完全(图2)。由于DNA纯化很难实现完全除去RNA及RNA的水解产物核糖核苷,因此实际DNA样品中通常都有RNA和核糖核苷存在,本研究中(图2)5 mC,5 mdC以及dC,C实现了完全基线分离,本方法对5 mdC%的定量不受样品中掺杂的RNA污染的影响。
2.2校准曲线及灵敏度
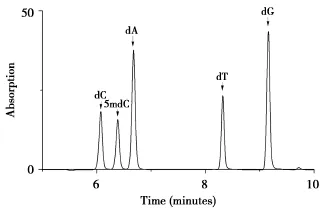
图1 5种脱氧核糖核苷的毛细管电泳分离图Figure 1The high performance capillary electrophoresis chromatogram(HPCE)of five deoxyribonucleic acids
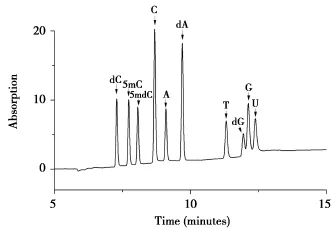
图2 10种核糖核苷的毛细管电泳分离图Figure2Thehighperformancecapillaryelectrophoresis chromatogram(HPCE)of ten ribonucleic acids

图3 dC校准曲线Figure 3The calibration curve of dC
dC的校准曲线为Y=172540.15X-6.29,相关系数(R)为0.999,线性范围是2×10-3mol/L-2× 10-1mol/L(图3)。5 mdC的校准曲线为Y=170271.74X-46.19,相关系数(R)为0.995,线性范围1×10-4mol/L-2×10-2mol/L(图4)。二者的检测限均为1×10-5mol/L,定量限均为2×10-5mol/L。
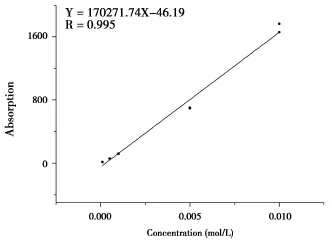
图45 mdC校准曲线Figure 4The calibration curve of 5 mdC
2.3重现性及在小牛胸腺DNA中的应用
日内和日间重现性分别用日内相对标准偏差(relative standard deviation,RSD%)和日间相对标准偏差(relative standard deviation,RSD%)来考量,本研究的日内相对标准偏差、日间相对标准偏差和准确度见表1。5 mdC%含量从5%到40%范围内,日内相对标准偏差<5%,准确度>80%;5 mdC%含量5%到10%范围内,日间相对标准偏差<10%,说明方法稳定性和准确性完全满足仪器分析定量要求。
应用本方法分析小牛胸腺DNA的甲基化水平,结果为6.18%,与本实验组前期应用液质联用(LC-MS/MS)的定量结果(6.77%)[10]和文献报导中应用液相色谱-紫外检测器(HPLC-UV)的检测结果(6.26%)有良好的一致性[6]。
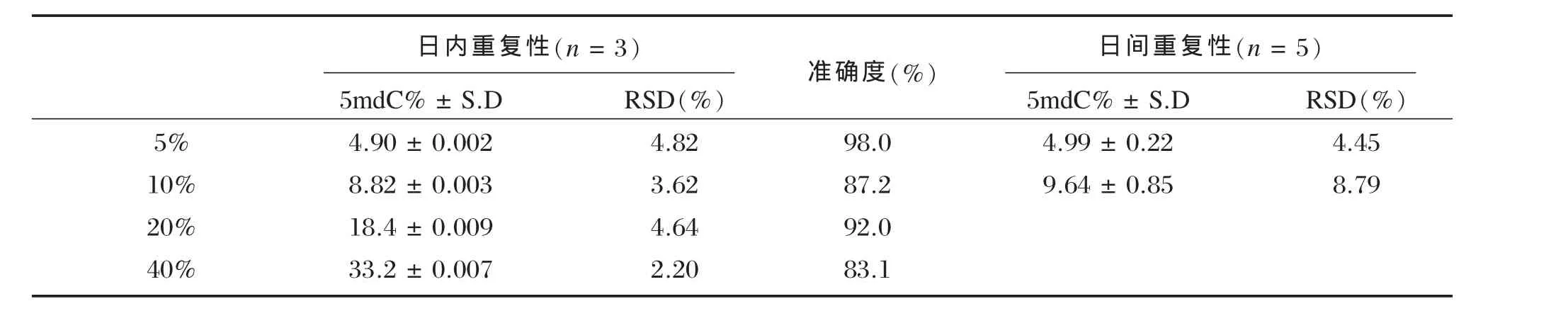
表1 高效毛细管电泳(HPCE)DNA甲基化定量方法的日间重复性和日内重复性Table 1Intra-day and inter-day reproducibility of the HPCE method
3 讨论
表观遗传学研究发现,甲基化修饰是基因表达调控的重要方式之一,启动子序列CpG岛区高甲基化引起基因表达抑制,因此甲基化也被认为是基因沉默的标识[11]。胚胎发育早期DNA甲基化修饰水平的动态变化,DNA甲基化修饰与基因印记[12],以及DNA甲基化修饰与许多慢性疾病如糖尿病[13]、高血压[14]、癌症[3]的关系,这一系列现象都是表观遗传学研究的热点。
DNA甲基化水平的定量是目前表观遗传学研究必不可少的数据基础。所有针对DNA甲基化对转录和表达影响的研究,以及营养变化对甲基化水平改变进而影响生命过程的表观遗传学研究都无可避免的需要测定DNA的甲基化水平——包括基因中特定位点的甲基化状态、特定基因的甲基化水平和整体基因组的甲基化水平。这些研究中涉及的DNA甲基化定量检测通常样品数较多而样品量较小,其中某些研究的样品采集时间跨度也较大。这些样品特点对检测方法的准确性,灵敏度,检测速度和批间差异的控制要求较高,也是目前DNA甲基化定量检测的难点所在。
目前广泛采用的定量检测方法包括免疫沉淀后[15]或重亚硫酸盐处理后[16]测序以及色谱直接分离定量。其中免疫沉淀的主要问题为限制性内切酶的选择困难。重亚硫酸盐处理应用亚硫酸氢盐使单链DNA中的胞嘧啶脱氨变成尿嘧啶,而5-甲基脱氧胞嘧啶无变化的特点对特定位点的甲基化情况进行检测,其主要的缺点为准确度依赖于脱氨反应的彻底性控制,并且该反应不能区分5-甲基脱氧胞嘧啶和5-羟甲基脱氧胞嘧啶,甲基化定量值是二者之和。这两类方法的优点在于能对特定位点的甲基化状态进行描述,方法容易以试剂盒的形式商品化。所以重亚硫酸盐处理测序是目前对特定片段的甲基化研究采用的最广泛的方法。色谱分离定量利用酶切将DNA彻底水解成单个核苷状态,然后利用色谱的高分离能力,对5种脱氧单核苷分离,并对色谱峰积分定量,从而计算出DNA的甲基化水平。这种方法无法对具体基因进行定量,因此主要用于基因组甲基化水平检测。其优点是直接定量5-甲基脱氧胞嘧啶和脱氧胞嘧啶的浓度,预处理过程简单,样品需求量低,但实际定量过程中,为了避免RNA污染的干扰,通常需要比较长的分析时间来实现5 mdC和dC的基线分离,或者需要选择步骤复杂,操作要求严苛的预处理过程去除RNA。这种高要求的实验操作在样品回收率和试验时间上都很难满足研究的要求。液质联用技术用于甲基化定量之后,借助质谱的定性能力,对分离要求大为降低,分析时间大大缩短,RNA的干扰也可以较容易的克服,因此成为目前整体甲基化定量的标准参考方法。但是液质联用的超高价格,严苛的操作技术要求和更先进的实验室环境需求使得这种方法只能在大型实验室进行,具有很大的局限性。
本研究基于色谱分离定量的思路,利用HPCE低廉的分析成本,更低的进样量和更高的分离能力的特点,对HPCE测定基因组整体甲基化的方法进行了优化并对各项性能指标进行了评价。其结果显示:组成DNA的5种脱氧核糖核苷和组成RNA的5种核糖核苷在13min内良好分离,与既往研究相比[8,9,17],本研究对DNA甲基化的定量不受样品中RNA污染的干扰。样品预处理应用混合酶溶液一步水解,6小时即可酶解完全,与既往HPCE定量DNA甲基化的研究中耗时18小时的多步水解方法相比[8,9,17],极大的节约了时间和人力,更加简便快捷稳定,容易实现高通量处理。5 mdC和dC的检测限均为1×10-5mol/L,dC的线性范围从2×10-3mol/L到2×10-1mol/L,5mdC的线性范围1×10-4mol/L到2×10-2mol/L。此检测限和线性范围配合HPCE超低的进样量完全满足检测对象超低样品量的要求。方法的稳定性和准确性也经过标准的方法学质控,完全满足仪器分析定量检测的要求;并且通过实际样品检测的对比,其结果(6.18%)的准确性与液质联用(LC-MS/MS)的定量结果(6.77%)[10]和文献报导中应用液相色谱-紫外检测器(HPLC-UV)的检测结果(6.26%)[6]有良好的一致性。HPCE的方法相对于液相色谱更加快速简便,达到了液质联用类似的结果,同时其仪器价格、实验成本、实验室环境要求和人员水平要求都远低于液质联用和液相色谱,适用于一般医院和研究机构,仪器自动化程度较好,利于在基层推广。
4 结论
本方法对5mdC%的检测可以较好的克服RNA污染的影响,准确性高,稳定性好,灵敏度能满足微量样本的检测需求。且与高效液相色谱方法相比,仪器试剂成本低廉且标本操作简便,可以做为标准方法用于临床诊断和研究。
[1]Chen X,Guo J,Lei Y,et al.Global DNA hypomethylation is associated with NTD-affected pregnancy:A casecontrol study[J].Birth Defects Research Part A:Clinical and Molecular Teratology,2010,88(7):575-581.
[2]Wang L,Wang F,Guan J,et al.Relation between hypomethylation of long interspersed nucleotide elements and risk of neural tube defects[J].The American Journal of Clinical Nutrition,2010,91(5):1359-1367.
[3]Sandoval J,Mendez-Gonzalez J,Nadal E,et al.A prognostic DNA methylation signature for stage I nonsmall-cell lung cancer[J].Journal of Clinical Oncology, 2013,31(32):4140-4147.
[4]Zhang Y,Zhao M,Sawalha A H,et al.Impaired DNA methylation and its mechanisms in CD4(+)T cells of systemic lupus erythematosus[J].Journal of Autoimmunity,2013,41:92-99.
[5]Song L,James S R,Kazim L.Karpf AR specific method for the determination of genomic DNA methylation by liquid chromatography-electrospray ionization tandem mass spectrometry[J].Analytical Chemistry, 2004,77(2):504-510.
[6]Sandhu J,Kaur B,Armstrong C,et al.Determination of 5-methyl-2′-deoxycytidine in genomic DNA using highperformanceliquidchromatography-ultraviolet detection[J].Journal of Chromatography B,2009,877 (20-21):1957-1961.
[7]Quinlivan E P,Gregory J F.DNA methylation determination by liquid chromatography-tandem mass spectrometry using novel biosynthetic[U-15N]deoxycytidine and[U-15N]methyldeoxycytidine internal stan-dards[J].Nucleic Acids Research,2008,36(18):e119.
[8]Fraga M F,Rodríguez R,Cañal M J.Rapid quantification of DNA methylation by high performance capillary electrophoresis[J].Electophoresis,2000,21(14):2990-2994.
[9]Fraga M F,Uriol E,Diego L B,et al.Highperformance capillary electrophoretic method for the quantification of 5-methyl 2′-deoxycytidine in genomic DNA:Application to plant,animal and human cancer tissues[J].Electophoresis,2002,23(11):1677-1681.
[10]Zhang M,Quan L,Lu X,et al.Hypomethylation of fetal brain genomic DNA in neural tube defects determined by a new liquid chromatography-electrospray ionization tandem mass spectrometry(LC-MS/MS)method[J].Analytical Methods,2012,4(8):2515-2521.
[11]Jones P A.Functions of DNA methylation:islands, start sites,gene bodies and beyond[J].Nat Rev Genet, 2012,13(7):484-492.
[12]Smith Z D,Meissner A.DNA methylation:roles in mammalian development[J].Nat Rev Genet,2013,14 (3):204-220.
[13]Rakyan V K,Beyan H,Down T A.Identification of type 1 diabetes-associated DNA methylation variable positions that precede disease diagnosis[J].PLoS Genet, 2011,7(9):e1002300.
[14]Smolarek I,Wyszko E,Barciszewska A M,et al.Global DNA methylation changes in blood of patients with essential hypertension[J].Med Sci Monit,2010, 16(3):149-155.
[15]Ley T J,Ding L,Walter M J,et al.DNMT3A mutations in acute myeloid leukemia[J].New England Journal of Medicine,2010,363(25):2424-2433.
[16]Huang Y,Pastor W A,Shen Y.The behaviour of 5-hydroxymethylcytosine in bisulfite sequencing[J].PLoS One,2010,5(1):e8888.
[17]Li M,Hu S L,Shen Z J,et al.High-performance capillary electrophoretic method for the quantification of global DNA methylation:application to methotrexateresistant cells[J].Anal Biochem,2009,387(1):71-75.
Quantification of genomic DNA methylation by high performance capillary electrophoresis
ZHANG Min,CHEN Qian,ZHANG Lijuan,QUAN Li★
(Beijing Municipal Key Laboratory of Child Development and Nutriomics,Capital Institute of Pediatrics, Beijing,China,100020)
ObjectiveTo establish a high-throughput and accurate quantitative method by high performance capillary electrophoresis(HPCE)for DNA methylation analysis.MethodsNaHCO3buffer containing sodium dodecyl sulfonate(SDS)was used as the running buffer.The operating voltage,column temperature and injection mode were optimized for baseline separation and quantification of nucleosides.Results5 deoxynucleosides and 5 nucleosides from RNA interference were totally separated in 10min.The calibration equation for deoxycytidine(dC)was Y=172540.15X-6.19 with correlation coefficient R of 0.999 and the linear range was from 2×10-3mol/L to 2×10-1mol/L.The calibration equation for 5-methyl deoxycytidine(5 mdC)was Y=170271.74X-46.19 with correlation coefficient R of 0.995 and the linear range was from 1×10-4mol/L to 2×10-2mol/L.The intra-day relative standard deviation(RSD%) was less than 5%and the inter-day relative standard deviation(RSD%)was less than 9%.The detection limit for dC and 5 mdC were both 1×10-5mol/L.Methylation value was calculated as the ratio of 5mdC to the total dC:[5 mdC]/([5 mdC]+[dC])×100%.The methylation level of calf thymus DNA measured using this method was 6.18%,which was consistent with the value observed on high performance liquid chromatography-tandem mass spectrometry(LC-MS/MS)and the value reported before on high performance liquid chromatography-UV detector(HPLC-UV).ConclusionThe method this study established is fast,high accuracy,good stability and good sensitivity.It could meet the requirement of determination of trace samples and be used in clinical methylation diagnosis.
DNA methylation;High performance capillary electrophoresis;Quantitative analysis
国家自然科学基金青年基金(NSFC20905051);北京市科技新星计划B类(2009B36)
首都儿科研究所,儿童发育营养组学北京市重点实验室,北京100020
★通讯作者:权力,E-mail:quanli.ql@gmail.com

