赤水丹霞山土壤来源抗菌活性链霉菌的分离及放线菌素产生链霉菌Streptomycessp.CSDX001发酵产物分析
岳昌武,李园园,黄兵,罗显涛,邵美云,王苗,保玉心,黄英
(1.遵义医学院贵州省微生物资源及药物开发特色重点实验室,贵州遵义563003;2.中国科学院微生物研究所微生物资源前期开发国家重点实验室,北京100101)
赤水丹霞山土壤来源抗菌活性链霉菌的分离及放线菌素产生链霉菌Streptomycessp.CSDX001发酵产物分析
岳昌武1,2,李园园1,黄兵2,罗显涛1,邵美云1,王苗1,保玉心1,黄英2
(1.遵义医学院贵州省微生物资源及药物开发特色重点实验室,贵州遵义563003;2.中国科学院微生物研究所微生物资源前期开发国家重点实验室,北京100101)
分离、鉴定赤水丹霞地区土壤来源的抗临床耐药菌活性的链霉菌。采集赤水丹霞山区不同海拔的土样并利用多种培养基分离、纯化菌株。热酚法提取菌株基因组DNA,PCR扩增16S rRNA基因并测序后在线比对以确定其系统分类学地位。利用多种测试菌测试抗菌活性,通过HPLC和LC/MS分析菌株发酵抽提物并与数据库比较。分离得到菌株600株,其中59株链霉菌中有41株具有1种以上的抗测试菌活性。Streptomycetessp.CSDX001具有抗耐药耐甲氧西林金黄色葡萄球菌、抗耐万古霉素金黄色葡萄球菌、抗多药耐药鲍曼不动杆菌、抗枯草芽孢杆菌、抗藤黄微球菌、抗白色念球菌的活性,具有抗多种微生物的活性和产生多种新结构次级代谢产物潜力,可能是抗微生物新天然活性产物的重要来源。
链霉菌;抗菌活性;16S rRNA;次级代谢产物
放线菌是抗生素的主要产生菌,来源于放线菌的抗生素类天然产物超过10 000种,这其中3/4以上的天然产物来自链霉菌,是微生物来源抗生素类物质的主要产生菌[1-3]。从特殊环境和典型生态环境中分离得到具有开发价值的新菌株和新的天然产物的可能性远远高于受人类活动影响较大的普通生态环境[4-7],如孙承航等[8-9]在塔克拉玛干沙漠南麓土壤及罗布泊地区沙生植物根际分离得到大量的可能产生新活性产物的抗菌活性放线菌。黄英[10]从南海深海海水分离出的海洋链霉菌Streptomyces olivaceusFXJ 8.012分离抗病原性Klebsiella pneumoniae新的化合物Tetroazolemycins A and B。
贵州很多地区保留了其独特的原生态环境,这些原生态环境中可能孕育着新的特殊的微生物资源,具有从中开发微生物抗菌药物的巨大潜力,值得进一步研究[11]。本试验通过对发酵产物进行高效液相色谱(high performance liquidchromatography,HPLC)和液质联用(chromatography/massspectrometry,LC/MS)分析并与已知数据库比较,从赤水丹霞地区土壤中分离41株有抗菌活性的的链霉菌分离菌株,其中1株产放线菌素C2/放线菌素D的链霉菌Streptomycetessp.CSDX001,该菌除了产放线菌素外,还具有很强的抗耐甲氧西林金黄色葡萄球菌临床分离株、抗耐万古霉素金黄色葡萄球菌、抗多药耐药鲍曼不动杆菌、抗枯草芽孢杆菌、抗藤黄微球菌、抗白色念珠菌活性,并可产生多种新结构次级代谢产物,是值得进一步开发利用的菌株资源。
1 材料与方法
1.1材料与试剂
土样采集:300份试验土样采集于贵州省赤水丹霞山区海拔600 m左右的除地表腐殖质的10~20 cm土层,分装于无菌样品采集袋中常温保存运输。
分离培养基:菌种分离主要利用高氏1号培养基(可溶性淀粉20 g,KNO31 g,NaCl 0.5 g,K2HPO40.5 g,MgSO40.5 g,FeSO40.01 g,琼脂20 g,加双蒸水定容至1 L)、自来水酵母粉琼脂培养基(酵母浸汁0.25 g,K2HPO40.5 g,琼脂18 g,加双蒸水定容至1 L)和GYM(glucose yeast extract maltextract)固体培养基(葡萄糖4 g,CaCO32 g,麦芽抽提物10 g,酵母抽提物4 g,琼脂粉15 g,加双蒸水定容至1 L)。
抗菌测试菌:多药耐药耐甲氧西林金黄色葡萄球菌(耐药谱:甲氧西林、氨苄西林、红霉素、林可霉素、青霉素、四环素)、多药耐药鲍曼不动杆菌临床分离株(耐药谱:氨苄西林、左氧氟沙星、氨苄新林/舒巴坦、哌拉西林/他唑巴坦、头孢哌酮/舒巴坦、氨曲南、头孢曲松、头孢他啶、头孢唑啉、环丙沙星、头孢吡肟、头孢替坦、呋喃妥因、庆大霉素、泰能霉素、妥布霉素)为遵义医学院附属医院检验科微生物检验实验室陈泽慧教授提供。枯草芽孢杆菌(Bacillus subtilis)、藤黄微球菌(MicrococcusluteusCGMCC1.2156)、耐万古霉素金黄色葡萄球菌、白色念珠菌等菌株均由微生物资源前期开发国家重点实验室提供。
主要试剂:抗菌药物及抗生素(抗菌剂储液:制霉菌素20 mg/L,重铬酸钾50 mg/L,萘啶酮酸20 mg/L,放线菌酮50 mg/L):Sigma公司;酚/氯仿等DNA提取试剂:北京索莱宝公司;常规生化试剂(分析纯):成都科龙化工试剂厂;分子生物学试剂及酶类:大连宝生物公司;1 000×微量元素预混液(ZnSO4·7H2O 2 g,FeSO4·7H2O 2 g,MnCl2·4H2O 2 g,CuSO4·5H2O 2 g,Na2B4O7·10H2O 2 g,(NH4)6Mo7O24· 4H2O 2 g加水定容至1 L);培养基:碧迪生物上海公司。
1.2仪器与设备
Hitachi SU8010冷场发射扫描电子显微镜:日本日立公司;Agilent 1220LC System VL高效液相色谱系统:美国安捷伦公司;1200HPLC/6520QTOFMS液质联用仪:美国安捷伦公司;HC-518R高速冷冻离心机:安徽中科中佳科学仪器有限公司;DZ-900英培摇床:太仓市实验设备厂;R-210旋转蒸发仪:瑞士步其公司;BSP-400培养箱:上海博讯设备仪器厂;E002092超级洁净工作台:苏州市金净净化设备有限公司。
1.3方法
1.3.1菌株分离及形态观察
分别称取每份样品2 g于无菌研钵中,加入0.2 g(土样重量的1/10)CaCO3,用无菌杵研磨成细粉,装入50 mL无菌离心管,28℃风干14 d,加入30 mL无菌水,在28℃摇床充分振荡1 h充分悬浮,55℃静置处理20 min,促使放线菌孢子萌发。采用梯度稀释法将土壤预混液分别梯度稀释成10-1、10-2和10-3等3个浓度梯度,振荡混匀后及时吸取200 μL土样悬浊液分别均匀涂布于固体分离培养基平板,28℃静置培养,随时观察培养基上的菌落状态,挑取疑似放线菌形态的目的菌株单菌落进行纯化培养[1]。电镜观察在中科院微生物研究所大型仪器中心进行,主要步骤:2.5%戊二醛固定过夜,双蒸水漂洗3次,每次8 min;依次用体积分数为50%-70%-85%-95%的乙醇梯度脱水15 min,然后用体积分数100%的乙醇脱水三次,每次15 min;然后利用BAL-TEC CPD030进行二氧化碳临界点干燥;BAL-TEC SCD005离子溅射仪喷金后利用FEI QUANTA 200扫描电镜观察。
1.3.2次级代谢产物发酵
接种环挑取一环活性菌株接种于装有100 mL GYM发酵液体培养基(ISP2+1 mL微量元素预混液,加入去离子水定容至1 L溶液)的500 mL锥形瓶中,28℃、150 r/min振荡培养215 h,菌体连同培养基加入100 mL乙酸乙酯,150 r/min、28℃振摇30 min充分萃取,静置10 min,取上层乙酸乙酯层于旋转蒸发仪中,38℃蒸干,加入1 mL甲醇重溶抽提物,用于活性测试和化合物结构初步分析。
1.3.3发酵产物活性测试
将多药耐药金葡菌、多药耐药铜绿假单孢菌、耐药肠杆菌、枯草芽孢杆菌、藤黄微球菌分别接种LB液体培养基,28℃摇床200 r/min培养6~8 h,白色念球菌接种GYM液体培养基,37℃摇床200 r/min培养6~8 h。取30 mL新鲜菌液与70 mL将凝固(约45℃)的相应固体培养基分别混匀后迅速倒板,待培养基凝固后用无菌打孔器打直径为5 mm孔,分别吸取50 μL抽提产物于孔中。多药耐药金葡菌、多药耐药铜绿假单孢菌、枯草芽孢杆菌、藤黄微球菌等测试平板置于37℃过夜培养。根据抑菌圈的有无及大小判定抗菌活性的强弱。
1.3.416S rRNA基因系统发育分析
将筛选得到的活性菌株利用经典溶菌酶/SDS结合酚/氯仿法提取基因组总DNA为模板,利用细菌16S rRNA PCR扩增通用引物27F/1492R进行PCR扩增,通用引物序列27F(5'-AGAGTTTGATCCTGGCTCAG-3')和1492R(5'-TACGGCTACCTTGTTACGACTT-3'),PCR反应条件为:50 μL反应体系94℃,预变性5 min;PCR热循环程序为94℃变性0.5 min,56℃退火0.5 min,72℃延伸1.5 min,共扩增30个循环后72℃总延伸5 min。PCR产物用1%琼脂糖凝胶电泳进行检测。16SrRNA的PCR扩增产物测序由上海立菲生物公司完成。根据测序结果,将16S rRNA序列用NCBI数据库中的Blast程序[9]在线进行序列相似性比对搜索,从GenBank数据库中获得相似性较高且是有效描述的典型菌株的16S rRNA序列作为参比对象,采用MEGA5.0软件进行聚类分析和构建系统发育树[12-15]。
1.3.5发酵产物的HPLC及LC/MS分析
色谱条件:InertsilODS-3液相色谱柱(4.6mm×150mm,5 μm),进样量:20 μL,流速1 mL/min,甲醇∶水(80∶20)梯度洗脱。粗产物经RP-HPLC(ShimadzuSPD-M20A,色谱柱:ODS,10×150 mm)进一步纯化,通过HRESI-MS(Waters Xevo G2 QTof)来鉴定化合物。
2 结果与分析
2.1活性菌株的分离

图1 Streptomycetessp.CSDX001发酵产物抗临床多药耐药菌活性分析Fig.1 Activity analysis ofStreptomycetessp.CSDX001 fermentation products against multi-drug resistance bacteria

表1 分离菌株抗菌活性Table 1 Antibacterial activity of isolated strains
采集了43份土壤样品,从中分离到600株菌,根据形态初步排重后选取其中59株链霉菌形态的菌株进行16S rRNA基因测序及序列分析,将其获发酵液乙酸乙酯萃取浓缩物进行抗菌活性筛选,发现有41株链霉菌分离菌株有至少1种以上的抗菌活性,活性菌株占测活菌株总数的69.5%。其中Streptomycetessp.CSDX001表现出广谱的抗测试菌的活力(见表1)。临床分离菌株测活结果表明Streptomycetessp.CSDX001具有很强的抗多药耐药耐甲氧西林金黄色葡萄球菌(抑菌圈约直径1cm)、多药耐药铜绿假单孢菌临床分离菌株的活性(抑菌圈直径>3cm)(见图1)。
2.2链霉菌Streptomycetessp.CSDX001形态特征
28℃分纯培养的Streptomycetessp.CSDX001菌落质地紧密,气丝发达,呈粉状,营养菌丝与培养基紧密结合,菌落产生黄色色素,背面呈褐色,成熟时产生灰色孢子(见图2A)。5 000倍电镜下观察Streptomycetessp.CSDX0010气生菌丝发达,孢子较少,基内菌丝有分支,菌丝无横隔,不断裂,孢子丝直或柔曲,孢子表面有疣,具备典型的链霉菌电镜形态特征(见图2B)[1]。
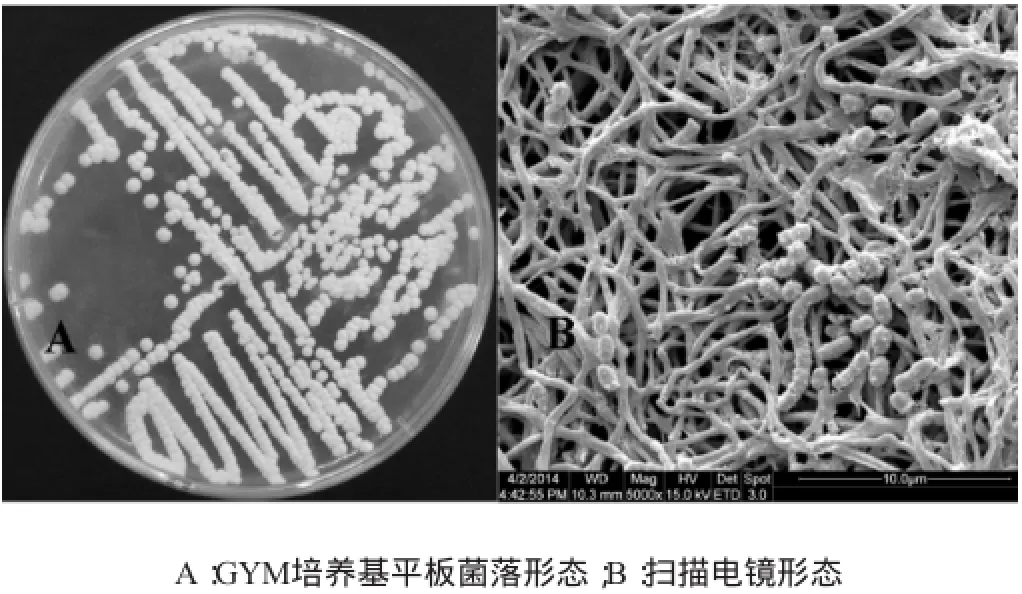
图2 Streptomycetessp.CSDX001形态特征Fig.2 Morphological characters ofStreptomycetessp.CSDX001
2.3Streptomycetessp.CSDX001 16S rRNA分子分类
利用细菌16S rRNA通用引物27F/1492R扩增得到约1 450 bp的PCR产物,PCR产物过柱纯化后利用该引物测序,测序结果拼接后得到1 390 bp核酸序列,利用该序列在线Blast,结果表明,Streptomycetessp.CSDX001 16S rRNA与链霉菌(Streptomyces griseoluteus)P510的16S rRNA同源性达97%,提示该菌可能属于Streptomyces griseoluteus类群中的一个比较新的种(见图3)。
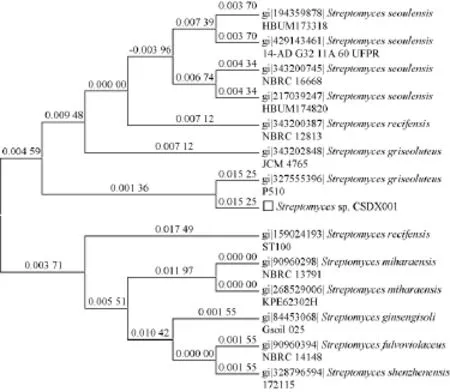
图3 Streptomycetessp.CSDX001 16S rRNA同源树Fig.3 Phylogenetic analysis ofStreptomycetes sp.CSDX001 16S rRNA
2.4链霉菌Streptomycetessp.CSDX001次级代谢潜力分析
对链霉菌Streptomycetessp.CSDX001进行小量发酵,乙酸乙酯抽提产物利用HPLC分析结果表明,该菌具有丰富的次级代谢产物,LC/MS分析结果表明,该菌产生放线菌素C2和放线菌素D(见图4),此外该菌的还产生天然产物数据库(http://db.sgst.cn/intersdb;http://scdb/default.asp;http://naturalprod.ucsd.edu/;www.pharmdata.ac.cn/China TACW.asp;microbial natural products database,MNPD等)里没有的紫外吸收特征的分子式可能分别为C23H36O7、C24H38O7和C23H36O6的3个化合物,其相应的紫外光谱及质谱图(见图5)提示这三个化合物可能是同系物。

图4 链霉菌Streptomycetessp.CSDX001产放线菌素LC/MS分析Fig.4 LC/MS analysis of actinomycins producted byStreptomycetessp.CSDX001

图5 链霉菌Streptomycetessp.CSDX001产疑似新化合物LC/MS分析Fig.5 LC/MS analysis of suspected new compounds produced byStreptomycetessp.CSDX001
3 讨论
临床上对耐药金黄色葡萄球菌、多药耐药铜绿假单孢菌、耐药肠杆菌属及白色念珠菌等感染缺乏有效的抗生素药物治疗[16-20],从特殊环境中分离新的来源的链霉菌可为解决这些难题提供物质基础[21-24]。贵州省境内蕴藏着丰富的微生物资源,具有很大的开发潜力。本研究从贵州省赤水丹霞地区土壤中采集的分离获得1株能产生放线菌素C2/放线菌素D的链霉菌Streptomycetessp.CSDX001,该菌除了能产生放线菌素外,还具有很强的抗耐药金黄葡萄球菌、多药耐药铜绿假单孢菌、抗枯草芽孢杆菌、抗藤黄微球菌及抗白色念球菌活性。此外,该菌可产生多种新结构次级代谢产物,值得进一步开发利用。后期对于该菌的研究主要集中在疑似新化合物的发酵条件的优化、化合物纯化及结构解析、生物活性的确定,期望能为工、农业生产及新药的开发提供新的天然产物来源。
[1]阮继生,黄英.放线菌快速鉴定与系统分类[M].北京:科学出版社,2011.
[2]徐丽华,李文军,刘志恒,等.放线菌系统学:原理、方法及实践[M].北京:科学出版社,2007.
[3]BÉRDY J.Thoughts and facts about antibiotics:Where we are now and where we are heading[J].J Antibiot,2012,65(8):385-395.
[4]BHATTACHARJEE K,BANERJEE K,JOSHI S R.Diversity ofStreptomycesspp.in Eastern Himalayan region computational RNomics approach to phylogeny[J].Biomed Inform,2012,8(12):548-554.
[5]LO C W,LAI N S,CHEAH H Y,et al.Actinomycetes isolated from soil samples from the Crocker Range Sabah[C].ASEAN Review of Biodiversity and Environmental Conservation,2002,1-7.
[6]WAKSMAN S A,WOODRUFF H B.Actinomyces antibioticus,a new soil organism antagonistic to pathogenic and nonpathogenic[J].J Bacteriol,1941,42(2):231-249.
[7]BOWEN D,GOLDMAN I D.The relationship among transport,intracellular binding,and inhibition of RNA synthesis by actinomycin D in Ehrlich ascites tumor cellsin vitro[J].Cancer Res,1975,35(11):3054-3060.
[8]董艳萍,郭琳,旭格拉·哈布丁,等.塔克拉玛干沙漠南麓土壤放线菌资源勘探及抗菌活性筛选[J].中国抗生素杂志,2013,38(4):241-247.
[9]庹利,旭格拉·哈布丁,郭琳,等.罗布泊地区沙生植物根际放线菌多样性及生物活性的研究[J].中国抗生素杂志,2012,37(1):21-26.
[10]LIU N,SHANG F,XI L,et al.Tetroazolemycins A and B,two new oxazole-thiazole siderophores from deep-seaStreptomyces olivaceusFXJ 8.012[J].Marine Drugs,2013,11(5):1524-1533.
[11]李园园,保玉心,吕玉红,等.梵净山保护区土壤放线菌分离及其次级代谢产物初步研究[J].长江大学学报:自然科学版,2013,10(23):60-65.
[12]ALTSCHUL S F,MADDEN T L,SCHAFFER A A,et al.Gapped BLAST and PSI-BLAST:a new generation of protein database search programs[J].Nucleic Acids Res,1997,25(17):3389-3402.
[13]SAITOU N,NEI M.The neighbor-joining method:a new method for reconstructingphylogenetic trees[J].Molecular biology and evolution,1987,4(4):406-425.
[14]KUMAR S,TAMURA K,NEI M,et al.MEGA:Molecular evolutionary genetics analysis software for microcomputers[J].CABIOS,1994, 10(2):189-191.
[15]EVANS J,SHENEMAN L,FOSTER J,et al.Relaxed neighbor joining:A fast distance-based phylogenetic tree construction method[J].J Mol Evol,2006,62:785-792.
[16]KUMARASAMY KK,TOLEMAN MA,Walsh TR,et al.Emergence of a new antibiotic resistance mechanism in India,Pakistan,and the UK:a molecular,biological,and epidemiological study[J].Lancet Infect Dis,2010,10(9):597-602.
[17]TAUBES G.The bacteria fight back[J].Science,2008,321(5887): 356-361.
[18]WANG LD,WANG Y,JIN SG,et al.Emergence and control of infectious diseases in China[J].Lancet,2008,372(9649):1598-1605.
[19]AMINOV RI.The role of antibiotics and antibiotic resistance in nature [J].Environ Microbiol,2009,11(12):2970-2988.
[20]Race against time to develop new antibiotics.Bulletin of the World Health Organization,2011.
[21]CHRONÁKOVÁ A,KRISTU°FEK V,TICHY′M,et al.Biodiversity of streptomycetes isolated from a succession sequence at a post-mining site and their evidence in Miocene lacustrine sediment[J].Microbiol Res,2010,165(7):594-608.
[22]RASSI H,GHASEMI SF.Production of novel chemotherapeutic drugs byStreptomycetes[J].Lik Sprava,2008(7-8):40-47.
[23]MANIVASAGAN P,VENKATESAN J,SIVAKUMAR K,et al.Marine actinobacterial metabolites:current status and future perspectives[J]. Microbiol Res,2013,168(6):311-32
[24]DEB ROY A,GR?SCHOW S,CAIRNS N,et al.Gene expression enabling synthetic diversification of natural products:chemogenetic generation of pacidamycin analogs[J].J Am Chem Soc,2010,132(35): 12243-5.
Isolation of antimicrobialStreptomycetesspp.from Chishui Danxia soil and fermentation product analysis of actinomycin producingStreptomycessp.CSDX001
YUE Changwu1,2,LI Yuanyuan1,HUANG Bing2,LUO Xiantao1,SHAO Meiyun1,WANG Miao1,BAO Yuxin1,HUANG Ying2
(1.Guizhou Key Laboratory of Characteristic Microbial Research&Drug Development,Zunyi Medical University,Zunyi 563003,China; 2.State Key Laboratory of Microbial Resources,Institute of Microbiology,Chinese Academy of Sciences,Beijing 100101,China)
In order to isolate and identify theStreptomycetesspp.with antimicrobial activity from Chishui Danxia region,the soil samples from different altitudes was collected,and then isolated and purified by a variety of medium.The strain gene DNA was extracted by hot phenol method,16S rRNA PCR products was sequenced and blasted online for determine the phylogeny of isolated strains.Antimicrobial activities were detected with multiple test bacteria,and the fermentation extracts were analyzed by HPLC and LC/MS,and the results were compared with database.Results showed that 600 strains were isolated and 41 out of 59Streptomycetesstrains had more than one antimicrobial activity.Streptomycessp.CSDX001 showed resistance activity to methicillin-resistantStaphylococcusaureus,vancomycin-resistant Staphylococcus aureus,multi drug resistantAcinetobacter baumannii,Bacillus subtilis,Micrococcus luteus,Candida albicans;it showed antimicrobial activities and secondary metabolites-producing potential,which might be a potential bacterial resource for active natural products research and development.
Stretomycetesspp.;antimicrobial activity;16S rRNA;secondary metabolite
R9
A
0254-5071(2014)11-0041-06
10.11882/j.issn.0254-5071.2014.11.009
2014-06-12
国家自然科学基金(31160004);贵州省教育厅特色重点实验室建设项目(黔教合KY字[2012]011号);贵州省科技学技术基金项目(黔科合J字[2010]2156号,黔科合J字[2012]2348号,黔科合SY字[2013]3013号)
岳昌武(1975-),男,副研究员,博士,研究方向为微生物天然产物发现及生物合成。
李园园(1987-),硕士研究生,研究方向为微生物天然产物发现及活性机制分析。
注:以上两人为并列第一作者。

