适于SSR分析的茶树高质量基因组DNA提取方法
王 磊 ,陈 琪 ,杨 华 ,王家琴 ,韦朝领 ,宛晓春 *
(1.安徽农业大学农业部茶叶生物化学和生物技术重点实验室,合肥230036;2.云南省红河州屏边县农业局茶果站,红河661400)
简单重复序列(simple sequence repeat,SSR)又称微卫星(microsatellite DNA),是一类由几个核苷酸(一般2~6个)为重复单位而成的长度可达几十个核苷酸的串联重复序列[1]。Powell等[2]对SSR的优点做了很好的综述,如共显性、多态性相对丰富,基因组覆盖较多等。由于该技术具有简便、快速、稳定性高和等位基因多样性高等特点,在基因组研究中作为一种主要的分子标记技术,已经广泛地应用于小麦、大豆、水稻、茶树等植物的遗传图谱的构建、遗传多样性分析和系统学研究等方面[3~6],而提取高质量的茶树基因组DNA则是茶树SSR分析的基础。
茶树[Camellia sinensis(L.)O.Kuntze]为多年生常绿木本植物,起源于中国的西南地区[7~10],然后从起源中心向其他地域自然传播并从中国向世界其他地区人为传播。由于茶树异花授粉的特性与人为的变异选择,发生了从形态水平到细胞水平、生物化学水平、再到分子水平的一系列连续的、渐进的变化[11],进而形成了今天丰富的种质资源。不同的茶树品种,由于其演化过程中环境和栽培方式的不同造成了其主要生化成分的差异,如野生茶树的茶多酚、儿茶素、总糖、寡糖和铁、铜、锰等微量元素含量较栽培种茶树高,而其他一些内含物则低于栽培种[12,13]。茶树叶片中富含的这些次生代谢物质给茶树DNA的提取带来很大的影响,而且不同品种茶树样品中次生代谢物含量差异较大,因此,一种适用于不同品种茶树的优质高效的DNA提取方法对于茶树SSR研究尤为重要,我们在一种广泛地适用于茶树以及其他植物基因组DNA提取的CTAB(十六烷基三甲基溴化铵)法[14,15]的基础上进行了改良,旨在建立起适合不同资源类型茶树SSR分析的基因组DNA提取方法。
1 材料与方法
1.1 材料
本实验中供试茶树材料分为3类:野生型、过渡型和栽培型,分别采自云南、贵州、安徽、福建等省份,共计10份,采摘新鲜幼嫩枝条保湿条件下放入冰盒带回,置于-80℃冰箱保存。
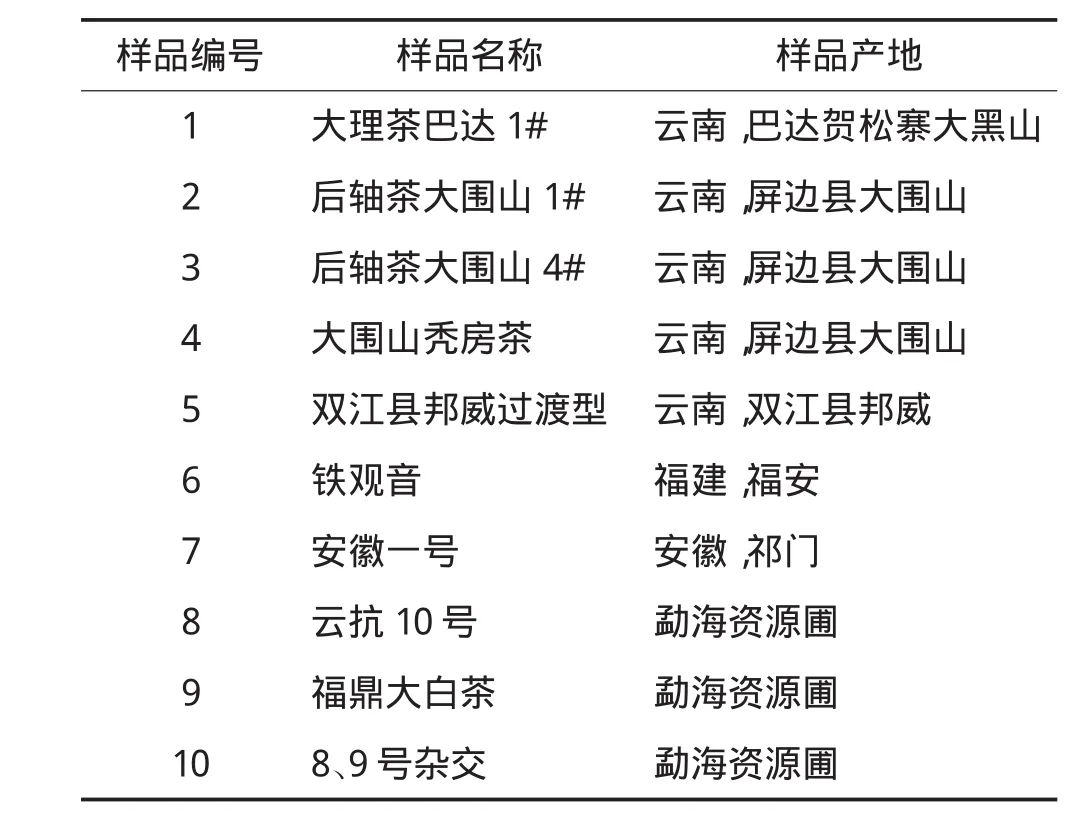
表1 材料及产地
1.2 实验设备
美国产Beckman Allegra 64R高速冷冻离心机,Thermo ND-1000核酸定量仪,SSR-PCR反应在Eppendorf PCR仪上进行,琼脂糖电泳和聚丙烯酰胺凝胶电泳分别在Bio-Rad Power Pac Basic和Bio-Rad Power Pac HV电泳仪上进行,用Bio-Rad凝胶成像仪拍照。
1.3 主要试剂
所用试剂均为分析纯,德国Sigma生产PVP40(PolyVinylPyrrilodone,聚乙烯吡咯烷酮),CTAB、Tris平衡酚和RNase-A为上海生工生产,rTaq DNA聚合酶、dNTP购自TaKaRa。SSR扩增引物根据Gen-Bank公布的茶树ESTs数据库和本实验室测序获得的基因组数据设计,由上海生工合成。
1.4 茶树总DNA的提取
(1)称取茶树鲜叶0.2g放入预冷的研钵中,加入等量的PVPP经液氮研磨均匀后装入灭菌的2ml离心管中,加入1mL pH 8.0的提取缓冲液(250mmol/L NaCl,250mmol/L Tris-HCl,50mmol/L EDTA)充分涡旋,12000rpm,室温离心5min。
(2)弃上清后加入1mL 65℃下预热的DNA抽提缓冲液(100mmol/LTris-HCl,pH8.0,1.4mol/LNa-Cl,20mmol/LEDTA,2%(w/v)CTAB,2%(w/v)PVP40)以及2%(v/v)β-巯基乙醇(现用现加),用灭菌枪头搅匀后置于65℃下水浴45~60min左右,期间大约每10min轻微翻转下离心管。水浴后12000rpm,室温离心15min。
(3)取上清于新的灭菌离心管中,加入0.1%RNAase-A(10mg/mL),37℃水浴 60min,每 10min轻微翻转下离心管。
(4)加等体积的酚:氯仿:异戊醇(25:24:1,v/v),温和翻转至充分混合均匀后12000rpm,25℃,离心15min。
(5)吸取上清于新的灭菌离心管,加入等体积的氯仿:异戊醇(24:1,v/v),按上一步骤再抽提1次。
(6)吸取上清于新的灭菌离心管,加入与上清等体积的预冷异丙醇,温和混匀后静置于-20℃1h以上。
(7)挑取每个样品中的絮状沉淀于新的灭菌离心管,加入70%的乙醇和无水乙醇各洗一次,弃酒精风干沉淀,加入50μL 1×TE溶解待用。
1.5 提取DNA质量检测
取2μLDNA样品,用Thermo核酸定量仪测定DNA样品在230nm、260nm和280nm波长处的紫外吸光度值,可以自动计算出A260/A280和A260/A230的比值,并结合0.8%琼脂糖凝胶电泳判断DNA的质量。
其中1~7号样采用本法提取,8~10号样采用天根DNA提取试剂盒提取。
1.6 SSR-PCR反应
扩增反应在Eppendorf公司扩增仪上进行,反应总体积为20μL,依次加入10×PCR Buffer(含20mmol/LMg2+)2μL,dNTP(2.5mmol/L)2μL,正反向引物(10μmol/L)各 1.5μL,rTaq酶(5U/μL)0.2 μL,20 ng/μL的DNA模板1μL,加ddH2O补足。
扩增反应程序采用热启动的方式,在加入Taq酶前对模板和引物用95℃预变性5min,降至78℃后加酶,然后进入33个循环,95℃变性1min,55~65℃(根据引物Tm值不同选择)退火1min,72℃延伸50s,33个循环后再72℃延伸5min。少数SSR引物中上下游引物退火温度相差较大的,可用Touchdown反应程序(郑景生和吕蓓,2003)[16]。采用前10个循环退火温度用58℃,后23个循环退火温度用50℃,这样对于上下游引物的退火温度相差较大的SSR标记也能扩增出清晰的条带。扩增后产物用6%SDS-PAGE电泳检测。
2 结果与分析
2.1 核酸定量仪检测
使用Thermo核酸定量仪检测DNA样品的纯度和浓度。一般说来,A260/A280和A260/A230的比值可以进行核酸样品纯度评估,A260/A280<1.8说明样品中蛋白(芳香族)或者酚类物质的污染含量较高,在1.8~2.0之间说明样品DNA纯度较高,若大于2.0说明样品中RNA含量较高。A260/A230比值若小于2.0表明样品被碳水化合物(糖类)、盐类或有机溶剂污染,需要纯化。结果如表2所示,本方法提取的DNA样品A260/A280绝大多数数值在1.8~2.0之间,而A260/A230的比值也基本上大于2.0,说明DNA样品的纯度高,少有蛋白质、RNA或其它的的污染。从表中比较还可以看出,采用本方法提取的样品浓度相较于试剂盒提取的DNA浓度大多数高了一个数量级,说明本法提取的DNA在保证质量的同时能够有很高的得率,这对于后续实验有着非常重要的意义,大大提高了珍贵植物材料的利用效率。
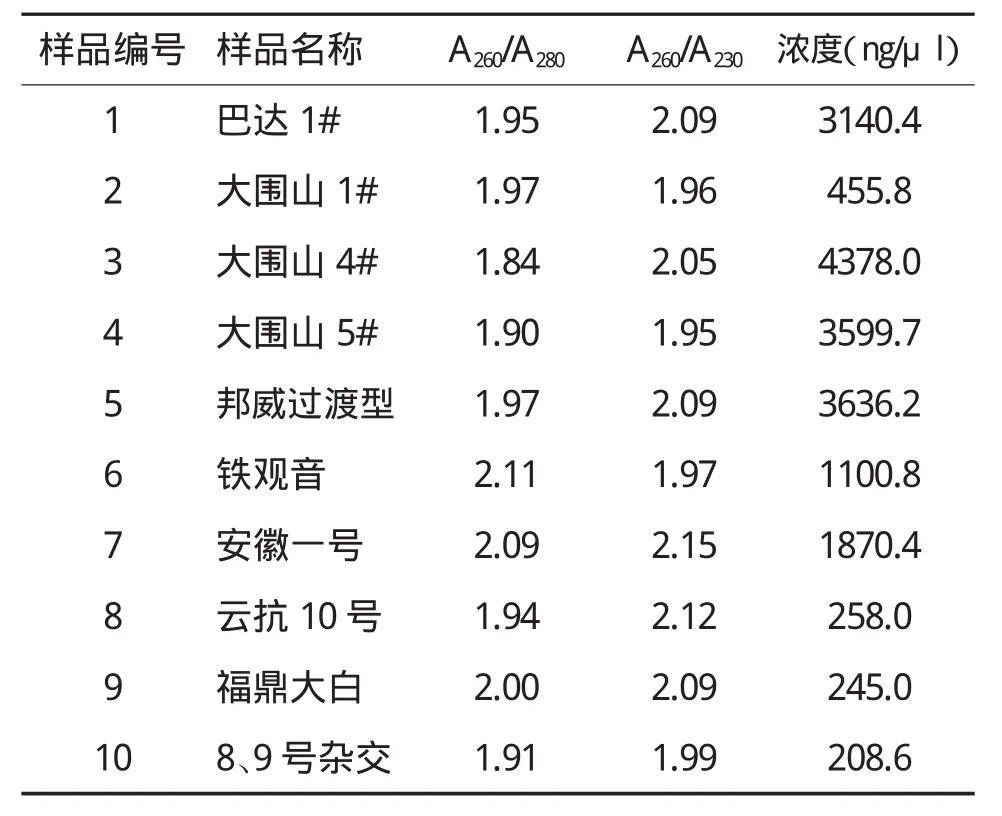
表2 提取DNA的纯度和浓度测定值
2.2 琼脂糖电泳检测

从图1可以看出,DNA样品条带清晰,亮度高,且蛋白污染少,没有拖带和弥散,说明本方法提取的DNA具有良好的完整性。点样孔附近滞留物少且没有RNA污染。
2.3 SSR-PCR分析
应用SSR技术时必需针对每个SSR设计引物,本课题组从已测序的基因组数据中选择一批SSR设计引物,从中筛选具有多态性的引物,从图2可以看到,PCR产物进行琼脂糖电泳图片的条带明亮清晰,进一步进行聚丙烯酰胺凝胶电泳分析后可以看出扩增出的多态性片段(见图3),说明本方法提取的DNA可以满足SSR分析的要求。
3 讨论
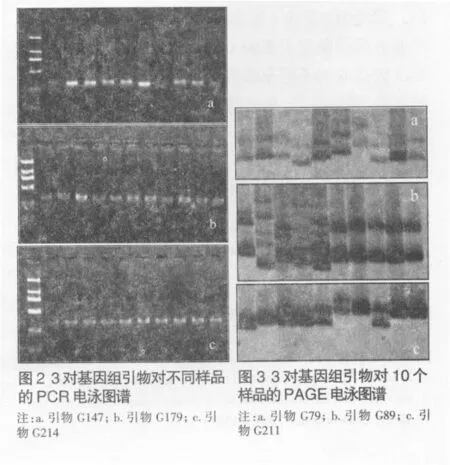
SSR分子标记是一种新型的分子标记技术,其在茶树上的应用有着广阔的前景[3,11,17],近年来,我国茶叶科研人员利用EST-SSR分子标记对我国丰富的茶树地方品种的遗传多样性进行了很多的分析[18~20]。本实验室从进行基因组SSR引物开发的需求出发,为了满足不同来源茶树品种基因组DNA提取的需要进行了提取高质量的DNA的技术改良。与其他植物相比,茶树嫩叶中含有丰富的多糖和多酚等次生代谢物质,会对提取的DNA质量产生很大的影响,本实验在前人CTAB法[21]的基础上进行改进,其不同之处在于:首先在加入CTAB提取液之前先加入普通提取缓冲液快速抽提,能够在一定程度上预先去除部分可溶性多糖、多酚和生物碱类物质,有利于不同样品的均匀化同时便于后续提取过程中细胞内含物的释放;其次是把RNA酶解步骤提前,在弃除CTAB提取液后直接用RNase-A去除RNA,然后再用有机溶剂去除蛋白,这样可以利用后续酚/氯仿/异戊醇去除蛋白质的同时除去RNase-A酶蛋白,避免酚/氯仿/异戊醇反复抽提过程对DNA链可能造成的断裂及影响DNA得率。实验结果表明,本方法提取的不同来源的茶树基因组DNA纯度高、浓度高、完整性好,能够满足SSR分析的要求。

表3 部分基因组SSR引物
[1]陆德如,陈永青.基因工程 [M].北京:北京化学工业出版社,2002:30~31.
[2]PowellW.,Machray G.C.,Provan J.Polymorphism revealed by simple sequence repeats[J].TrendsPlantSci,1996,1:5221~5222.
[2]陈宏.基因工程原理与应用[M].北京:中国农业出版社,2004,328~329.
[3]陈盛相,齐桂年,李建华.微卫星DNA分子标记技术及其在茶树研究中的应用[J].福建茶业,2008,1:36~37.
[4]Li-Ping Zhao,Zhen Liu,LiangChen.Generation and characterization of 24 novel EST derivedmicrosatellites from tea plant(Camellia sinensis)and cross-species amplification in its closely related species and varieties[J].ConservGenet,2008,9:1327~1331.
[5]Xu Y,Ma RC,Xie H,Liu JT,Cao M Q.Developmentof SSRmarkers for the phylogenetic analysis of almond trees from China and the Mediterranean region[J].Genome,2004,47:1091~1104.
[6]Wu X-L,He C-Y,Chen S-Y,et al.Phylogenetic analysis of interspecies in genusglycine through SSRmarkers[J].Acta GenetSin,2001,28(4):359~366.
[7]虞富莲.论茶树原产地和起源中心[J].茶叶科学,1986,6(1):1~8.
[8]Hashimoto M,Takasi S.Morphological studieson the origin of tea plant V:A proposal of one place of origin by cluster analysis[J].Japanese Journalof TropicalAgriculture,1978,21(2):93~101.
[9]HashimotoM.Theorigin of the tea plant[J].Japan AgriculturalResearch Quarterly,1985,19(1):40~43.
[10]Takeo T,You XQ,Wang H F,etal.One speculation on theorigin and dispersion of tea plant in China--One speculation based on the chemotaxonomy by using the contest-ration of terpen-alcohols found in the tea aroma composition[J].Journal of Tea Science,1992,12(2):81~86.
[11]陈亮,虞富莲,杨亚军.茶树种质资源与遗传改良[M].北京:中国农业科学技术出版社,2006:11~40.
[12]陈继伟,梁名志,王立波,等.古茶园与台地茶园鲜叶常量成分及成茶品质比较研究[J].中国农学通报,2011,27(4):339~344.
[13]梁名志,夏丽飞,张俊,等.老树茶与台地茶品质比较研究[J].云南农业大学学报,2006,21(4):493~497.
[14]袁长春,施苏华,叶创兴.从富含酚类的茶类植物叶中提取纯净的总DNA[J].中山大学学报论丛,2001,21(3):1~4.
[15]高俊,蔡新,许明辉,等.云南茶树DNA提取和PAPD分子标记和探讨[J].云南农业大学学报,2000,12(2):126~128.
[16]郑景生,吕蓓.PCR技术及实用方法[J],分子植物育种,2003,1(3):381~394.
[17]周炎花,姚明哲,陈亮,等.茶树遗传演化研究进展及SSR在茶树遗传演化研究中的应用前景[J].中国农学通报,2009,25(15):9~15.
[18]金基强,崔海瑞,陈文岳,等.茶树EST-SSR的信息分析与标记建立[J].茶叶科学,2006,26(1):17~23.
[19]王丽鸳,姜燕华,段云裳,等.利用SSR分子标记分析茶树地方品种的遗传多样性[J].作物学报,2010,36(12):2191~2195.
[20]Ming-Zhe Yao,Chun-LeiMa,Ting-TingQiao,etal.Diversity distribution and population structureof teagermplasms in China revealed by EST-SSR markers[J].Tree Genetics&Genomes,DOI 10.1007/s11295-011-0433-z.Publish online:24September2011.
[21]高洪晓,杨凯,刘建斌.3种植物DNA提取法中多糖类物质去除效果的研究[J].北京农学院学报,2011,26(1):70~72.

