腹膜间皮瘤组织体细胞突变的分析
陈 宾,马建婷,陈利玲,洪旭涛,唐晓婧,陈枢青
(1.华中科技大学同济医学院,湖北武汉 430030;2.余姚人民医院,浙江余姚 315400;3.浙江大学浙江加州国际纳米技术研究院,浙江杭州 310058;4.浙江大学药学院,浙江 杭州 310058)
由于第二代全基因组测序技术的发展,测序成本迅速下降,全基因组测序分析成为常用的研究手段。2010年,《Nature》发表了2个重要研究成果[1-2],一个是对属于同一个体的肺癌细胞和皮肤细胞进行了全基因组序列测定,发现肺癌细胞存在22910个体细胞突变;另一个发现黑色素瘤细胞与来自同一个体的淋巴细胞存在33345个体细胞突变。之后,脑瘤、白血病、卵巢癌、嗜铬细胞瘤和副神经节瘤、肝癌、肾癌、肺癌[3-9]等都进行了类似研究。由于体细胞突变是造成肿瘤、糖尿病、高血压等许多难治性疾病的主要因素,准确测定体细胞突变有重要的理论和实际意义。那么,目前的检测方法是否可以准确判定体细胞突变呢?本研究选取了1例手术切除的间皮瘤和癌旁组织,分别进行了全基因组测序,将测序结果与人类基因组数据库的参考序列进行比较,对肿瘤细胞的体细胞突变进行分析鉴定,结果发现正常细胞存在的体细胞突变会干扰肿瘤体细胞突变的鉴定,因此建议,尽早为个体进行全基因组测序或者保存一份人生早期的DNA样本以便对照。
1 材料与方法
1.1 材料 临床标本来源于1例68岁确诊为高度分化的乳头状腹膜间皮瘤的女性患者,其临床指标为:CK5/6(+++),CK7(+++),钙结合蛋白(++),D2-40(+),vim(++),CK(++),ER(-),PR(-),CEA(-),Ki67(<10%+)。样本为肿瘤手术切除的肿瘤组织及其癌旁组织,本研究经过浙江大学医学院伦理委员会的批准及患者的知情同意。
1.2 DNA提取、文库构建及测序 临床标本迅速放入液氮中冻存备用。DNA提取前,切除含血管的肿瘤组织。肿瘤组织及癌旁组织的基因组DNA用QIAGEN DNeasy血液组织试剂盒提取。根据Illumina TruSeq DNA SamplePrep v2说明书,用3μg提取的基因组DNA建立测序用DNA文库,再用DNA LabChip 1000试剂盒和Agilent bioanalyzer芯片分析系统进行质量检测。测序由Illumina Hiseq2000完成,碱基识别由 CASAVA 1.8.2 完成。
1.3 生物信息学分析 利用Burrows-Wheeler Aligner(BWA,http://bio-bwa.sourceforge.net/bwa.shtml),将过滤后的数据定位到参考基因组 hg18、hg19(http://hgdownload.cse.ucsc.edu/downloads. html#human),用 Samtools(http://samtools.sourceforge.net/)去除由 PCR反应产生的冗余读数,用GATK(http://www.broadinstitute.org/gatk/)检测 SNV。之后利用dbSNP135(http://www.ncbi.nlm.nih.gov/projects/SNP/)和 refGene(http://refgene.com)的基因组信息,用软件 ANNOVAR(http://www.openbioinformatics.org/annovar/)对 SNV进行注释和归类。
2 结果
2.1 DNA文库的质量 图1显示DNA文库的平均插入片段长度为350 bp。
2.2 测序数据概况 应用双末端测序技术分别从肿瘤组织和癌旁正常组织获得了149 M和150 M的测序数据(表1)。去除污染数据后,分别从肿瘤组织和癌旁组织获得了12.5 Gbp和12.6 Gbp的纯净数据。
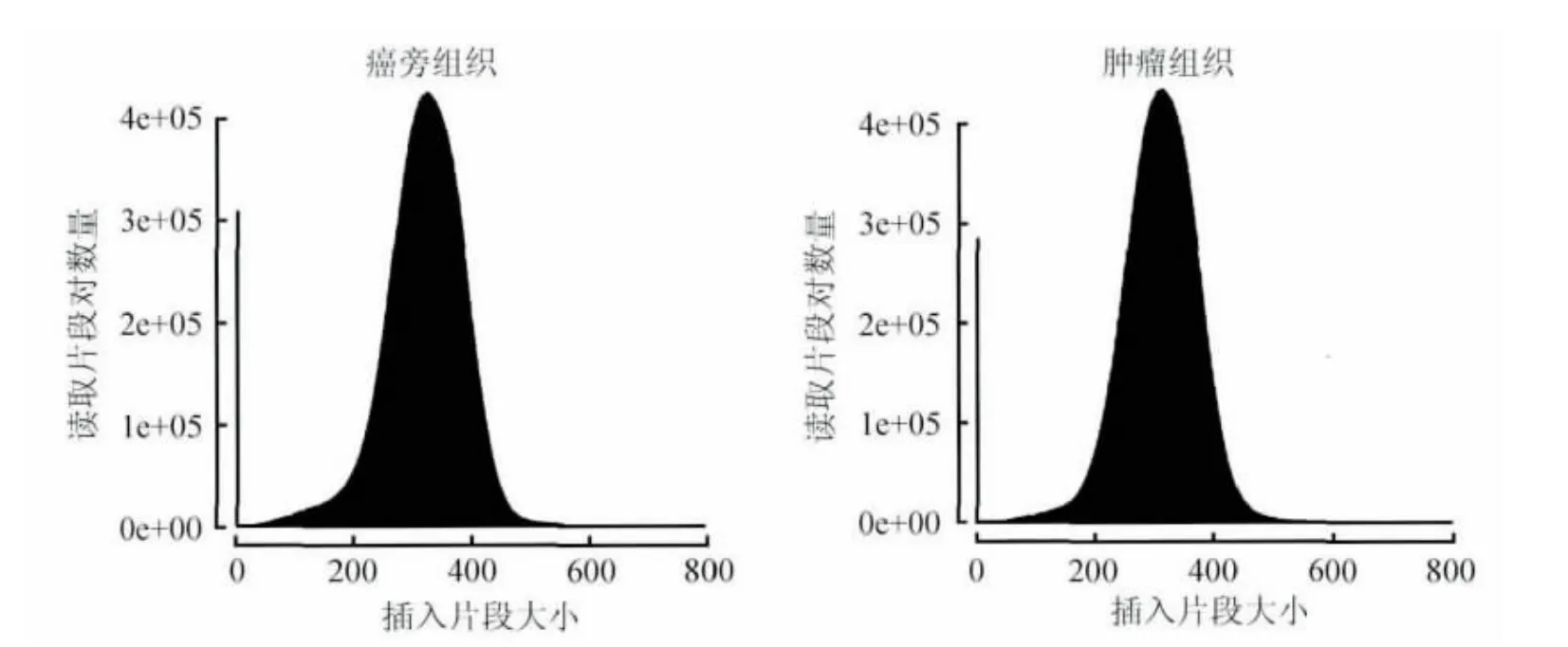
图1 肿瘤组织和癌旁组织样本建立的DNA测序文库中插入片段的长度分布Fig.1 Distrabution of fragment sizes of DNA library built by tumor and paraneoplastic tissues
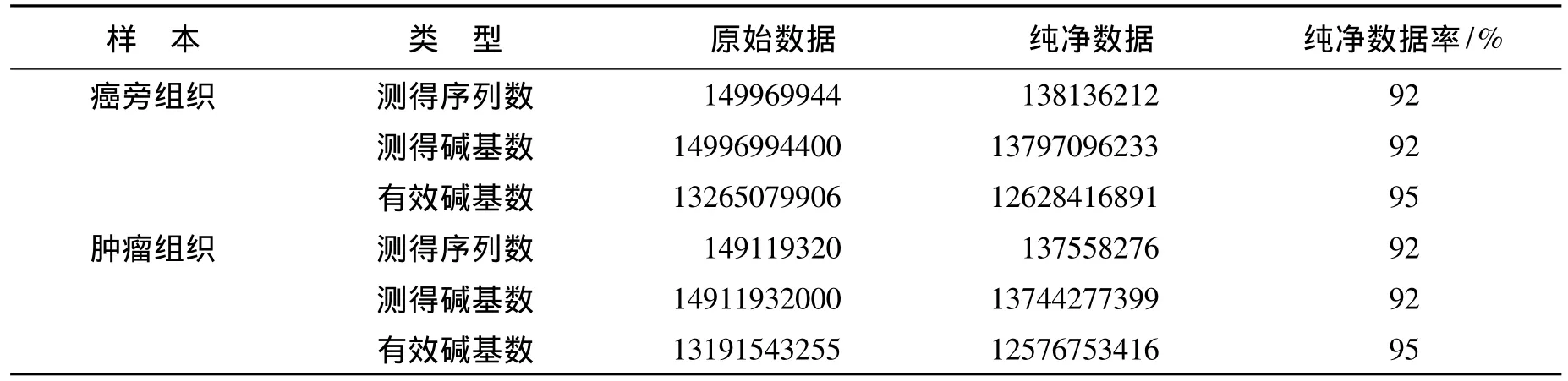
表1 肿瘤组织和癌旁组织分别测序获得的数据量对照Table 1 Sequencing data from tumor and paraneoplastic tissues
2.3 生物信息学分析获得的SNV 根据上述方法获得的数据,本研究比较了2个样本间高可信度的 SNV、位于外显子的高可信度的SNV、包含非同义突变的基因、包含插入/缺失的基因。所得数据如表2。

表2 肿瘤组织、癌旁组织及人类基因组参考序列相互比对结果Table 2 Variation numbers of nucleotides among tumor,paraneoplastic tissues and human genome reference sequence
3 讨论
体细胞突变是区别于生殖细胞突变的一种突变类型,生殖细胞突变会将突变的后果传递到下一代个体,并对下一代的表型产生影响,对自身是几乎没有影响的。而体细胞突变只在同一个体随细胞分裂传递突变的后果,影响的是其本人。人一生长期与环境互相作用,有些DNA首先发生体细胞突变,这个突变位点会在其分裂生成的子代细胞中得到遗传,称为体细胞遗传。在遗传信息传递过程中,体细胞突变会逐步积累。环境越恶劣,接触时间越长,积累的体细胞突变越多。2011年,Gaisa等[10]对膀胱上皮细胞进行了研究,发现其拥有多个单克隆族群。每一个族群内部拥有相同的基因序列,而族群之间在某些基因上可发现独特的体细胞突变特征。肇事突变(driver mutation)或肇事突变组合(driver mutation pattern)的形成是细胞生长过程中的一个偶然事件。一旦发生就会让细胞发生异化,异化了的细胞如果能够遵循体内“细胞社会”的规则,一般会选择凋亡以保证个体组织正常发展。但有时异化的细胞没有选择凋亡并逃脱体内免疫监视,则形成异常细胞群,进而影响组织、器官,发生疾病。很多疾病如肿瘤、糖尿病、高血压和阿尔茨海默病等都有这样的一个发生发展的过程。由于体细胞突变是一件随机事件,其发生的位点是极其个性化的,导致肿瘤、高血压、糖尿病等疾病发生的个性化特点,因此造成一种药物不能治疗所有患者的苦恼。个性化医疗不仅要关注患者的个性化,如年龄、性别、有否吸烟等外在因素,以及靶标基因多态性和代谢分布基因多态性等内在因素,还要同时关注疾病的个性化,如体细胞突变的个性化等。目前已经有许多研究阐述了体细胞突变在诊断和治疗难治性疾病中的重要作用[11-16]。
本研究对一例腹膜间皮瘤进行了肿瘤组织及癌旁组织的全基因组测序,试图研究第二代测序技术在检测体细胞突变中的临床适应性。图1显示两个样本的测序DNA文库建立是符合要求的,两者基本一致。表1的结果显示测得的数据量也基本符合要求。表2的结果是最值得深入分析的。首先假设N不存在任何体细胞突变,那么,无论N=R或者N≠R,只要是N≠T都是肿瘤存在的体细胞突变,本研究中就是 R=N≠T、R=T≠N、R≠T≠N 之和 22710个体细胞突变。但是实际上N要么是癌旁组织,要么是血液中白细胞或者是来源便利的其他组织样本。根据广泛接受的理论,在人与环境长期接触过程中,环境物质对DNA攻击造成体细胞突变,对于同一个体中的不同组织来说概率几乎相等,除非有特殊的职业接触或者非正常事故。对于普通个体,一般体细胞的存在数量与年龄大小和环境的恶劣程度成正相关。因此,可以粗略认为正常细胞和肿瘤细胞存在几乎数量相等的体细胞突变,只是肿瘤细胞中的体细胞突变发生在关键位点,而正常细胞中的体细胞突变不是在关键位点。以此推理,我们可以粗略判断肿瘤存在的体细胞突变应该是22710的一半。可是,从诊断和治疗的角度来说,数量不是最重要的,具体的肿瘤体细胞突变位点才是最重要的。那么,哪些位点是只属于肿瘤细胞的体细胞突变呢?R=N≠T是肿瘤体细胞突变的概率较大,但也存在R的那个位点就是一个体细胞突变的可能性,因为R也是来源于某一个DNA的测序结果。R=T≠N看似最大概率是N的体细胞突变,但是,也不排除N是一个SNP,而T突变后正好与R一样。R≠T≠N是突变发生的热点(hotspots),根本无法推测到底是哪一个细胞发生了突变,这种位点数量虽少,却往往起很重要的作用。
临床研究选择对照是一个非常重要的问题,由于这类疾病都是长期与环境接触造成的,现实中这样的对照组织是不存在的。唯一的办法是利用多种组织样本,进行反复测序,再用计算机推导演算,才可能获得那些可用于诊断和治疗的体细胞突变位点。这样就会给将来基因科技的应用带来很多问题,如驱动突变和肿瘤发生发展的理论性问题,以及靶向治疗、免疫治疗和基因治疗的个体化诊疗手段中的具体位点选择的实际性问题。如果能够为每一个新生婴儿进行全基因组测序或者保存一份当时的DNA,对于将来利用基因科技发展带来的个体化诊疗技术的应用,将是一个卓有远见的考虑。杨焕明等[17-18]曾经提出为新生儿进行全基因组测序,由于测序成本与近期获益不成比例,响应者寥寥无几。测序方法仍然是目前研究的热点,可以预见基因测序成本将继续下降,但是,我们认为更明智的方法是保存一份可以为每个个体一生作对照的DNA。
[1]PLEASANCE E D,STEPHENS P J,O'MEARA S,et al.A small-cell lung cancer genome with complex signatures of tobacco exposure[J].Nature,2010,463(7278):184-190.
[2]PLEASANCE E D,CHEETHAM R K,STEPHENS P J,et al.A comprehensive catalogue of somatic mutations from a human cancer genome [J].Nature,2010,463(7278):191-196.
[3]CLARK M J,HOMER N,O'CONNOR B D,et al.U87MG decoded:the genomic sequence of a cytogenetically aberrant human cancer cell line[J].PLoS Genet,2010,6(1):e1000832.
[4]LINK D C,SCHUETTPELZ L G,SHEN D,et al.Identification of a novel TP53 cancer susceptibility mutation through whole-genome sequencing of a patient with therapy-related AML [J].JAMA,2011,305(15):1568-1576.
[5]BELL D,BERCHUCK A,BIRRER M,Integrated genomic analyses of ovarian carcinoma [J].Nature,2011,474(7353):609-615.
[6]BURNICHON N,VESCOVO L,AMAR L,et al.Integrative genomic analysis reveals somatic mutations in pheochromocytoma and paraganglioma[J].Hum Mol Genet,2011,20(20):3974-3985.
[7]TAO Y,RUAN J,YEH S H,et al.Rapid growth of a hepatocellular carcinoma and the driving mutations revealed by cell-population genetic analysis of whole-genome data[J].Proc Natl Acad Sci USA,2011,108(29):12042-12047.
[8]DALGLIESH G L,FURGE K,GREENMAN C,et al.Systematic sequencing of renal carcinoma reveals inactivation of histone modifying genes [J].Nature,2010,463(7279):360-363.
[9]GREULICH H. The genomics of lungadenocarcinoma:opportunities for targeted therapies[J].Genes Cancer,2010,1(12):1200-1210.
[10]GAISA N T,GRAHAM T A,MCDONALD S A,et al.The human urothelium consists of multiple clonal units,each maintained by a stem cell[J].J Pathol,2011,225(2):163-171.
[11]OCANA A,PANDIELLA A.Personalized therapies in the cancer"omics"era [J].Mol Cancer,2010,9:202.
[12]PICKER A,JACKSON D B.Genetic determinants of anticancer drug activity:towards a global approach to personalized cancer medicine [J].Expert Rev Mol Diagn,2011,11(6):567-577.
[13]SWORDS R T,DEZUBE B J,MEDEIROS B C.Personalized medicine for acute myelogenous leukemia--at the entrance gate [J].Am J Hematol,2011,86(8):631-632.
[14]DIENSTMANN R,MARTINEZ P,FELIP E.Personalizing therapy with targeted agents in nonsmall cell lung cancer[J].Oncotarget,2011,2(3):165-177.
[15]MULLER F L,COLLA S,AQUILANTI E,et al.Passenger deletions generate therapeutic vulnerabilities in cancer[J].Nature,2012,488(7411):337-342.
[16]IYER G,HANRAHAN A J,MILOWSKY M I,et al.Genome sequencing identifies a basis for everolimus sensitivity [J].Science,2012,338(6104):221.
[17]YANG Huanming(杨焕明).Personal Genomics-New Challenge of Life Ethics in New Era of Biomedicine [J].Medicine and Philosophy:Humanistic& Social Medicine Edition(医学与哲学:人文社会医学版),2009,30(10):1-4.(in Chinese)
[18]CAPLAN J,DELL K,DORFMAN A,et al.TIME's Best Inventions of 2008.Time Magazine.2008-10-29. http://www. time. com/time/specials/packages/0,28757,1852747,00.html

