耐氟康唑光滑念珠菌耐药机制研究
张 炜, 应春妹
(上海交通大学医学院附属仁济医院检验科,上海200127)
侵袭性念珠菌引起的深部真菌感染的上升,已经成为一个严重的公共健康问题[1]。随着免疫抑制剂在临床的广泛使用,免疫缺陷人群的增多尤其是获得性免疫缺陷综合征患者的增加,目前白念珠菌在临床的分离率有所下降,而非白念珠菌,尤其是光滑念珠菌等引起的感染明显增加[2]。由于光滑念珠菌对常用抗真菌药物包括两性霉素B的敏感性低,且对三唑类抗真菌药物的最低抑菌浓度值偏高,可对三唑类药物原发耐药,也可出现继发耐药。耐药的发生是一个多水平、多因素参与的复杂过程。念珠菌对唑类药物耐药机制的研究已经成为当今医学研究的热点,但国内目前在念珠菌耐药性尤其是光滑念珠菌耐药性方面的研究仍然较少。因此,我们采用实时荧光定量逆转录聚合酶链反应(polymerase chain reaction,PCR)对光滑念珠菌临床分离株ERG11、CDR1和CDR2基因表达的mRNA进行相对定量,从而探讨基因表达与耐药之间的关系。
材料和方法
一、材料
1.试验菌株 光滑念珠菌临床菌株共22株,其中对氟康唑耐药11株(编号分别为NSG-1、DF-1、DF-2、DF-3、DF-5、DF-7、DF-8、DF-10、DF-17、DF-18、DF-20)、剂量依赖性敏感 3 株(EME-1、SUR-2、SIC-1)、敏感 8 株(NSG-7、NSG-3、NSG-4、SUR-1、RES-2、WFF-1、SIC-2、OST-1),均由仁济医院检验科鉴定并保存。试验菌株药物敏感性分析方法及结果判定参考文献[3]。质控菌株为近平滑念珠菌(ATCC 22019)和克柔念珠菌(ATCC 6258),由瑞金医院卢湾分院检验科惠赠。
2.主要试剂与仪器 Trizol试剂 (美国Invitrogen公司);DEPC(北京鼎国昌盛公司);氯仿、异丙醇、无水乙醇(上海化学试剂公司);反转录试剂 (美国 Fermentas公司);Realtime试剂(瑞士Roche公司);RNA free水 (日本Takara公司);荧光定量PCR仪 (美国ABI公司);核酸蛋白分析仪 (德国Eppendorf公司)。
3.引物 扩增光滑念珠菌 CDR1、CDR2、ERG11基因以及actin编码基因(ACT1)的引物均使用Primer 5.0软件进行设计。所有引物均由Invitrogen公司合成。具体引物序列见表1。

表1 荧光定量PCR引物
二、方法
1.试验菌株总RNA提取 首先挑取活化后的单个菌落接种于酵母浸出粉胨葡萄糖培养基中30℃振荡培养过夜,用细胞计数板调整菌液浓度至(5~10)×106个/mL;再取1mL菌液离心收集细胞,用焦碳酸二乙酯水洗涤2次;然后按照Trizol试剂说明提取试验菌株总RNA,最后加入适当体积30~50μL RNA free水,充分溶解。取适量样本1.2%琼脂糖凝胶电泳检测RNA完整性,并分别取1μL样本测定吸光度(A)260nm/A280nm值,其余分装,-70℃保存。
2.逆转录PCR 在冰上配制逆转录反应液,先采用11μL总体积(10×缓冲液1μL;脱氧核糖核酸酶1μL;RNA样本4μg;RNA free水补足至11μL),37℃ 30 min;然后加入终止缓冲液(Stop solution)和胸腺嘧啶寡聚核苷酸Oligo(dt)各1μL,75℃ 5 min,冰上退火5 min,离心2~3 s;最后加入5*Moloney小鼠白血病病毒缓冲液5μL,dNTP5μL,核糖核酸酶抑制剂1μL,Moloney小鼠白血病病毒1μL,总体积25μL,42℃1h,70℃ 10min,冰上退火得cDNA。
3.实时PCR PCR反应体系(25μL):SYBR Green PCR预混液12.5μL、引物(10μmol/L)各2.5μL、cDNA 模板 2.5 μL、dH2O5.0μL。反应条件:50℃2min,95℃10min;95℃15s,60℃60s,共40个循环。
三、统计学方法
每一样本重复3次,求其平均Ct值(即每个反应管内的荧光信号达到设定的阈值时所经历的循环数)。依据Livak等[4]设计的一种比较阈值法来测定目的基因的相对表达量,目的基因的量表示的是试验组目的基因的表达相对与对照组的变化倍数。
通过R(2.15.2)软件进行数据处理和分析,数据用中位数(M)和四分位数(P25~P75)表示,用W:lcoxon轶和检验进行显著性检验,P<0.05为差异有统计学意义。
结 果
一、逆转录PCR产物的确定分析
紫外分光光度计测定RNA在260和280 nm波长A值,A260nm/A280nm均在1.8~2.0,且RNA电泳结果显示 RNA完整性好,说明所提取真菌RNA质量较好。ERG11、CDR1、CDR2以及 ACT1基因均可见扩增曲线呈现典型的S型荧光定量动力学曲线,溶解曲线为单一波峰、形状锐利,说明试验中未出现污染、引物二聚体和非特异性扩增。
二、耐药组与敏感组多个耐药基因mRNA表达水平差异
将22株临床光滑念珠菌分为耐药组、剂量依赖性敏感组和敏感组,不同组 CDR1、CDR2和ERG11基因mRNA表达差异见表2和图1~3。CDR1基因在耐药组与敏感组以及剂量依赖性敏感组与敏感组之间差异均有统计学意义(W=88、24,P均<0.01);CDR2基因在耐药组与敏感组以及剂量依赖性敏感组与敏感组之间差异存在统计学意义(W=71、24,P均<0.05);ERG11 基因在耐药组与敏感组以及剂量依赖性敏感组与敏感组之间差异也有统计学意义(W=82、24,P均<0.05)。

表2 不同组CDR1、CDR2和ERG11基因mRNA表达的差异 [M(P25~P75)]
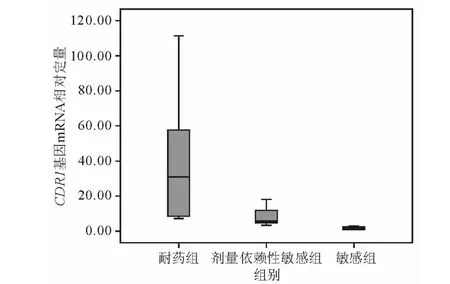
图1 光滑念珠菌耐药组、剂量依赖性敏感组及敏感组CDR1基因表达量比较

图2 光滑念珠菌耐药组、剂量依赖性敏感组及敏感组CDR2基因表达量比较

图3 光滑念珠菌耐药组、剂量依赖性敏感组及敏感组ERG11基因表达量比较
讨 论
由于获得性免疫缺陷综合征发病率增高,免疫抑制剂、广谱抗菌药物和糖皮质激素的大量应用,器官移植术、外科侵袭性操作及肿瘤放化疗的不断发展,使得深部真菌感染发病率不断升高。目前光滑念珠菌已成为仅次于白念珠菌之后居于第2位的常见真菌感染病原体,且由于光滑念珠菌对唑类抗真菌药物的最低抑菌浓度值趋于更高,且有些光滑念珠菌对抗真菌药物存在固有的低敏感率,包括两性霉素B[5],造成感染后治疗相当困难。Borst等[6]研究表明即使事先未使用唑类药物治疗,光滑念珠菌也会由于ATP结合转运蛋白的表达增高而导致耐药。
导致临床光滑念珠菌对唑类抗真菌药物耐药的机制主要有:(1)唑类药物作用的靶酶即羊毛甾醇14α-去甲基酶增加,导致三唑类药物在光滑念珠菌细胞内必须有更高的浓度才能发挥其阻断靶酶合成的作用;(2)药物与羊毛甾醇14α-去甲基酶亲和力下降;(3)真菌细胞内药物外排能力增强而导致细胞体内无法积聚药物,而与光滑念珠菌耐药密切相关的ATP结合转运蛋白是Cdr1p和Cdr2p,分别由CDR1和CDR2编码;(4)因固醇Δ5,6-脱氢酶失活造成麦角固醇生物合成途径失活。
部分试验研究结果提示,CDR1、CDR2及ERG11的过度表达与光滑念珠菌耐药性形成有关。本研究对该3种基因mRNA表达水平进行相对定量,发现耐药菌株 CDR1、CDR2及ERG11基因的mRNA表达量均高于敏感株,且随着对氟康唑耐药程度增加而增加,提示试验菌株CDR1、CDR2及ERG11基因表达上调可能是本研究中光滑念珠菌临床分离株对氟康唑耐药的主要分子机制。这与多数文献报道结果一致[7]。除此之外,我们还发现CDR1的表达水平明显高于CDR2,提示不同基因在耐药性形成中地位不同,CDR2基因在本研究所用菌株耐药性形成中的作用较CDR1基因小,这与国外的研究结果一致。Sanglard等[8]研究发现敲除光滑念珠菌CDR2基因未导致其对唑类药物敏感性的增加,而同时敲除CDR1和CDR2可使光滑念珠菌对唑类药物敏感性上升50%并且不会出现耐氟康唑的突变株。因此,CDR2可能是依赖于CDR1发挥作用的,但本试验中并未发现CDR1基因表达而CDR2不表达的情况。
综上所述,光滑念珠菌CDR1、CDR2及ERG11基因的过表达是临床分离菌株对氟康唑耐药的主要分子机制,但光滑念珠菌耐药的形成是一个多因素作用、多水平调节的复杂过程,单个耐药基因或单一耐药机制均不能完全解释光滑念珠菌的所有耐药现象,尚待在后续试验中进一步研究。
[1]龚晓霖,储怡星,范基农,等.真菌感染及其药敏试验方法[J].检验医学,2005,20(1):81-83.
[2]Pfaller MA,Messer SA,Boyken L,etal.Geographic variation in the susceptibilities of invasive isolates of Candida glabrata to seven systemically active antifungal agents:a global assessment from the ARTEMIS Antifungal Surveillance Program conducted in 2001 and 2002[J].J Clin Microbiol,2004,42(7):3142-3146.
[3]郑 冰,姚冬婷,应春妹,等.医院感染光滑念珠菌耐药性及流行病学分析[J].检验医学,2012,27(6):461-466.
[4]Livak KJ,Schmittgen TD.Analysis of relative gene expression data using real-time quantitative PCR and the 2(-Delta Delta C(T))method[J].Methods,2001,25(4):402-408.
[5]Lockhart SR,Joly S,Pujol C,etal.Development and verification of fingerprinting probes for Candida glabrata[J].Microbiology,1997,14(Pt 12):3733-3746.
[6]Borst A,Raimer MT,Warnock DW,etal.Rapid acquisition of stable azole resistance by Candida glabrata isolates obtained before the clinical introduction of fluconazole[J].Antimicrob Agents Chemother,2005,49(2):783-787.
[7]Niimi M,Nagai Y,Niimi K,etal.Identification of two proteins induced by exposure of the pathogenic fungus Candida glabrata to fluconazole[J]. J Chromatogr B Analyt Technol Biomed Life Sci,2002,782(1-2):245-252.
[8]Sanglard D,Ischer F,Bille J.Role of ATP-bindingcassette transporter genes in high-frequency acquisition of resistance to azole antifungals in Candida glabrata[J].Antimicrob Agents Chemother,2001,45(4):1174-1183.

