酵母菌诱导和阻遏分子机理的研究进展*
池振明,马再超
(中国海洋大学海洋生命学院,山东 青岛,266003)
在微生物中诱导和阻遏是非常普遍的代谢调控现象。在细菌中尤其在大肠杆菌(Escherichia coli)中诱导和阻遏分子机理已经比较清楚[1]。但是在酵母菌中诱导和阻遏的分子机理明显不同于细菌,许多详细的分子机理,特别是与信号传导有关的机理还很不清楚。酿酒酵母(Saccharomyces cerevisiae)和其他许多酵母菌可以在各种碳源中生长繁殖,但是葡萄糖和果糖是它们生长的最适碳源。当培养基存在其中之一碳源时,用于分解其他碳源所需的酶合成速率就会降低或完成不能合成,这种现象就叫做碳代谢物阻遏作用(Carbon catabolite repression)。但是至今还未发现能起阻遏作用的“非葡萄糖碳代谢物”,所以更多情况下用葡萄糖阻遏作用(Glucose repression)这个名词来代替碳代谢物阻遏作用这个说法[2]。除了葡萄糖阻遏作用外,酵母菌还存在氮代谢物阻遏作用,当培养基缺少富氮源如谷氨酰氨、天冬氨酸或氨时,酿酒酵母可以利用不良氮源(精氨酸、尿素、脯氨酸等)作为氮源。这时细胞中的氮阻遏作用可以在转录水平上得到解除,把这种现象叫做氮代谢物阻遏作用(Nitrogen catabolite repression),简称NCR[3]。许多丝状真菌和酵母菌生长在含铁培养基中时,铁载体合成和有关基因的表达也会受到阻遏。当酿酒酵母生长在半乳糖和麦芽糖培养基中时,与半乳糖和麦芽糖代谢有关的基因表达和酶活力便可以得到诱导;同样当产淀粉酶的酵母菌生长在淀粉、糊精和麦芽糖培养基中时,淀粉酶基因表达和产生的淀粉酶活力可以得到诱导;当产乳糖酶的酵母菌生长在乳糖和半乳糖培养基中时,乳糖酶基因和产生的乳糖酶活力可以得到诱导。现在一般认为阻遏作用是一种影响整个细胞全局的代谢调控现象,也就是说一旦细胞得到葡萄糖解阻遏或氮解阻遏,许多酶的基因表达和酶活力,甚至许多代谢途径都可以得到解阻遏,而诱导作用具有特异性,也就是说受某种物质诱导后的细胞只能与该物质代谢有关的基因表达和酶活力受到诱导,而与其他物质代谢有关的基因表达和酶活力不会受到影响。这样诱导的机理要比阻遏机理复杂的多,所以酵母菌的许多诱导机理至今仍很不清楚。
1 葡萄糖阻遏的分子机理
当培养基存在葡萄糖或果糖时,酵母菌代谢其他糖的酶基因表达和酶合成会受到阻遏。现在一般认为这种阻遏作用主要发生受葡萄糖阻遏的各种基因转录水平上。参与葡萄糖阻遏的蛋白质有2类,一类叫做Mig1(Multicopy Inhibitor of Galactose,半乳糖多拷贝抑制蛋白),另一类叫做 Snf1(sucrose non-fermentable,蔗糖非发酵蛋白)复合物,实际上Snf1复合物是一类激酶,它能催化Mig1发生磷酸化反应,也有人认为还有一类蛋白属于连Snf1复合物激酶激酶,就是能催化Snf1复合物激酶发生磷酸化反应的酶,但是目前这种Snf1复合物激酶激酶还未知。Mig1这种蛋白质是C2H2锌指蛋白,它们能结合在由葡萄糖阻遏的各种基因启动子上,结合时启动子上必须有GC盒,含有共有序列(G/C)(C/T)GGGG,但是在与 GC盒连接的DNA 5′-端也应含有1个富含AT的区域。在酒精酵母细胞中具有Mig1和Mig2两种蛋白。现有证据表明Mig1蛋白具有2个锌指结构,锌指结构1能辨认G(G/A)G三联密码子,而锌指结构2能辨认(G/C)(C/T)G三联密码子。Snf1复合物是一种蛋白质激酶,在培养基中葡萄糖浓度很低或无葡萄糖情况下,在Snf复合物激酶激酶催化下,Snf1复合物受到磷酸化作用,这时它可以催化Mig1蛋白质发生磷酸化反应,这时磷酸化的Mig1蛋白质便不能与受葡萄糖阻遏的基因启动子结合,并离开细胞核到细胞质中,这样有关的基因便得到了解阻遏,并进行基因表达,所以Snf1复合物在酵母菌葡萄糖阻遏作用过程中由葡萄糖引起的信号传导途径中起着非常重要的作用。在培养基葡萄糖浓度很高的情况下,Snf1复合物去磷酸化作用,这时Snf1复合物是没有活性的,Mig1蛋白质由于不能受到磷酸化作用,便对有关基因,如SUC2(蔗糖酶基因)基因产生阻遏作用[2,4],如图1所示。
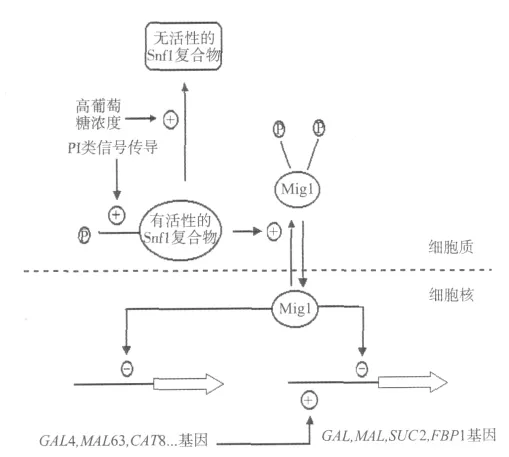
图1 Mig1和Snf1作用的模式图Fig.1 The schema chart of Mig1and Snf1
在这个过程中细胞应该能感受培养基中的葡萄糖浓度,所以无疑还存在由葡萄糖引起的信号传导作用,通过这种信号传导作用使Snf1复合物激酶发生磷酸化作用还是去磷酸化作用。现在一般认为引起葡萄糖阻遏的信号就是细胞内的葡萄糖浓度,而不是葡萄糖流入细胞的速度,所以葡萄糖本身就是1个信号分子[2]。
2 磷脂酰肌醇类信号传导与葡萄糖阻遏的可能关系
在酵母菌细胞中的磷脂主要由磷脂酸(PA)、磷脂酰胆碱(PC)、磷脂酰乙醇胺(PE)、磷脂酰肌醇(PI)、磷脂酰丝氨酸(PS)和心磷脂CL)组成的。其中的磷脂酰肌醇(PI)的化学结构如图2所示。
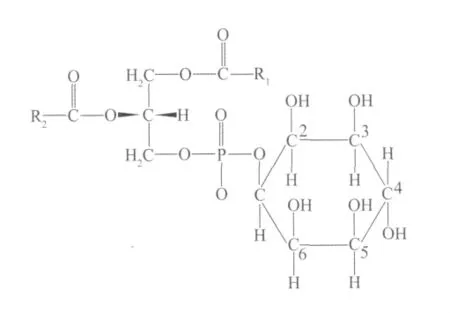
图2 磷脂酰肌醇的化学结构Fig.2 The chemical structure of PI
本实验室大量研究结果表明在酿酒酵母中蔗糖酶分泌导致的葡萄糖解阻遏作用与细胞中PI含量有某种关系,同时作者发现酿酒酵母中蔗糖酶分泌的解阻遏作用是发生在转录水平上,因为酿酒酵母分泌的蔗糖酶活力与编码胞外蔗糖酶的mRNA量和PI含量存在有正相关性。与在酿酒酵母中的情况一样,当粟酒裂殖酵母含有较高PI含量时,蔗糖酶分泌、细胞生长和编码蔗糖酶的基因表达在某种程度上得到了葡萄糖解阻遏作用,并且这种葡萄糖解阻遏作用认为与细胞中较高浓度的PI含量有密切的关系。粟酒裂殖酵母的麦芽糖酶是胞内酶,研究结果发现该酵母菌的磷脂酰肌醇含量与粟酒裂殖酵母麦芽糖酶基因的表达和麦芽糖酶活力也有某种关系。当酒裂殖酵母细胞PI含量增加时,能够解除高浓度葡萄糖对麦芽糖酶活力和麦芽糖酶基因表达的阻遏作用[4-6]。
在酵母菌细胞中,PI在激酶催化下通过依赖ATP的磷酸化作用(ATP-dependent phosphorylation)可分别在肌醇分子的第4位和第5位上发生磷酸化反应,形成4,5-二磷酸磷脂酰肌醇(PIP2)。当细胞外的某些刺激物与细胞质膜表面的受体结合后,可以活化对磷脂酰肌醇特异的磷脂酶C(Phosphatidylinoside-specific phospholipase C,简称PLC),在细胞质膜上的PLC把PIP2裂解成具有第二信使功能的1,4,5-三磷酸肌醇(IP3)和CDP-二酰基甘油(CDP-DAG)。由于IP3是可溶性的,它便从细胞质膜上进入细胞质中,这时便引起细胞内的钙离子库,如内质网的钙离子库得到释放,细胞质中钙离子浓度的增加可以活化细胞中许多酶,如蛋白质激酶(Protein kinase C,简称PKC)的转位和激活,但是PKC的完全激活还需要DAG和磷脂酰丝氨酸(PS)。PKC激活后会引起pH和细胞膜的一系列变化[4]。根据这种PI类信号传导的原理,把PI类信号传导与上述的PI含量与葡萄糖解阻遏的关系联系在一起,在培养基中添加肌醇或酵母细胞合成更多的肌醇,引起酵母菌细胞中的PI含量增加,这时即使酵母细胞生长在高浓度葡萄糖培养基中,酵母细胞中PI含量的增加通过PI类的信号传导作用还可以激活Snf1复合物激酶激酶,使Snf1复合物发生磷酸化反应,并同时导致Mig1发生磷酸化反应而被转移到细胞质中,这样便引起蔗糖酶和麦芽糖基因表达和蔗糖酶和麦芽糖分泌进而发生葡萄糖解阻遏作用。
3 葡萄糖解阻遏与酵母菌酶的合成和调控
扣囊复膜酵母菌(Saccharomycopsis fibuligera)是子囊菌属的1个种,在营养生长过程中可以出现丝状细胞和酵母状细胞,是一种二形态酵母菌,它可以利用淀粉积累大量的海藻糖,并能分泌大量的淀粉酶、酸性蛋白酶、β-葡糖苷酶和其它酶。所以扣囊复膜酵母在发酵行业、医药行业和生物能源工业有着巨大的潜在应用价值[4]。在有葡萄糖存在情况下,该酵母菌许多酶的合成和基因表达都会受到阻遏。为了了解Mig1蛋白在该酵母菌葡萄糖阻遏中的作用,从扣囊复膜酵母A11菌株的基因组DNA中克隆出了MIG1基因。克隆的MIG1基因(NCBI的登录号:HM450676)全长1 152bp,编码由384个氨基酸组成的蛋白质,该蛋白质的氨基酸序列与其它真菌的Mig1s非常类似,有高度保守的2个锌指结构,这2个锌指结构可以与受葡萄糖阻遏的基因启动子结合。在该Mig1蛋白的N-末端,有1个KPTLK的氨基酸序列,该序列决定了该蛋白可以在细胞核中定位[8]。另外该蛋白第二锌指结构中还有1个KRFS氨基酸序列,它是依赖环化AMP的蛋白激酶催化的磷酸化位点。该蛋白也有1个含有多个谷氨酰氨的序列,所有这些特征都是Mig1蛋白必须具备的。获得该酵母MIG1基因后,就敲除该菌株的MIG1基因,获得了敲除菌株A11-c,它能在含有2-脱氧-D葡萄糖的平板上生长,而原始菌株A11则都不能,说明敲除菌株的葡萄糖阻遏得到了解除。与菌株A11相比,敲除菌株A11-c在72h内产生的α-淀粉酶、葡萄糖淀粉酶、酸性蛋白酶和β-葡糖苷酶比活力达到了89.11,17.42,2.12 和0.20U/mg菌体干重,而野生型A11菌株在同样条件下在72h内产生的α-淀粉酶、葡萄糖淀粉酶、酸性蛋白酶和β-葡糖苷酶比活力仅达到了41.26,9.76,1.63和0.16U/mg菌体干重。同时编码α-淀粉酶、葡糖淀粉酶、酸性蛋白酶和β-葡糖苷酶基因的表达量也有极大提高,由野生型A11菌株的100%α-淀粉酶、葡糖淀粉酶、酸性蛋白酶和β-葡糖苷酶基因的表达量提高到敲除菌株A11-c的271.2%,308.6%,190.7%和468.7%α-淀粉酶、葡糖淀粉酶、酸性蛋白酶和β-葡糖苷酶基因的表达量。同时发现敲除菌株A11-c在生长过程中多数细胞呈酵母状细胞,丝状细胞非常少,说明MIG1基因的敲除还可以改变该酵母细胞的形态,其中的原理还不清楚。这就证明,扣囊复膜酵母中的Mig1蛋白确实能阻遏某些基因的转录,它对一些基因的表达和一些胞外酶的生产起负调控作用[9]。
Kluyveromyces marxianus是1种能进行同宗接合的半子囊酵母菌,在亲缘关系方面与酿酒酵母关系比较近,它是 Kluyveromyces lactis 的姐妹种[10-11]。该酵母最重要的特征是能够同化乳糖和菊糖,并能利用这2种糖作为碳源[12]。由于K.marxianus能同化乳糖和菊糖、生长速率非常快、代时短、具有抗热性和具有很强的分泌蛋白质的能力,所以被广泛应用在生物技术领域。该酵母菌被公认为是最安全的微生物,所以有关该酵母菌的产品在实际应用中没有任何限制,这样该酵母菌在生物技术领域中就更加广泛了。该酵母产生的乳糖酶和纤维二糖酶是胞内酶,产生的菊糖酶是与细胞壁结合。现在发现该酵母的乳糖酶、菊糖酶和其他酶合成和分泌也受到葡萄糖阻遏,并且发现转录阻遏蛋白(Mig1)在葡萄糖阻遏作用中起非常重要的作用[12]。所以利用含有潮霉素抗性 HPT基因的ORF框替换了工业酵母菌K.marxianus KM 的MIG1基因ORF框,获得的敲除突变株(mig1突变株)KM-15能够分别生长在含有潮霉素和2-脱氧-D-葡萄糖培养基上。与K.marxianus KM相比,突变株的β-半乳糖苷酶,菊糖酶和纤维二糖酶合成和这些酶基因的表达得到很大提高。敲除突变株KM-15产生的β-半乳糖苷酶,菊糖酶和纤维二糖酶比活力在48h内达到了7.2,5.7和0.06U/mg菌体干重,而K.marxianus KM在同样条件下β-半乳糖苷酶,菊糖酶和纤维二糖酶比活力在48h内达到了3.4、2.5和0.01U/mg菌体干重。β-半乳糖苷酶,菊糖酶和纤维二糖酶基因的表达量由K.marxianus KM的100%增加到敲除突变株 KM-15的205.5%,298%和699.6%[13]。这些研究结果表明Mig1蛋白可以调节工业酵母菌胞内酶和细胞壁结合酶的合成和它们基因的表达,MIG1基因的敲除可以解除这些酶的合成和基因表达,大大提高酶的产量。同时发现该酵母β-半乳糖苷酶,菊糖酶和纤维二糖酶的合成分别受到了乳糖,菊糖和纤维二糖的诱导[13]。上述2个例子说明在产工业用酶酵母菌中敲除MIG1基因是提高酶产量的重要方法之一。
4 在铁载体合成过程中阻遏机理的研究
铁在所有生命过程中具有非常重要的作用。但是细胞中过量的铁也是有害的,所以生物体内的铁浓度必须进行严格的调节。铁在自然界中是1种非常丰富的元素,但是生物可利用的铁量却非常有限,浓度不超过10~18mol/L。在铁浓度非常低的情况下,大多数真菌可以分泌一种特异络合铁的小分子化合物,即铁载体(Siderophore),从而使环境中的铁以可溶性的形式存在。铁载体可以分为3类:(1)儿茶酚类(Catechols);(2)羧酸类(Carboxylates)和(3)羟肟酸类(Hydroxamates)。但是真菌一般仅产生羟肟酸类铁载体[14]。当铁载体络合三价铁离子后,便被真菌吸收,在细胞内三价铁离子被还原成二价铁离子,然后二价铁离子被细胞利用。所有真菌的铁载体合成都是从L-鸟氨酸(L-ornithine)开始,第一步反应是L-鸟氨酸的N5发生羟基化形成 N5-羟基-L-鸟氨酸(N5-hydroxyornithine),参与催化的酶是 L-鸟氨酸-N5-羟化酶 (L-orinithine-N5-xoygenase),从某些丝状真菌中,如曲霉中已经克隆到编码该酶的基因,称为SidA或Sid1,发现在霉菌中这些基因的表达受到铁离子的严重阻遏。第二步反应是N5-羟基-L-鸟氨酸发生烷基化反应,形成 N5-烷基-N5-羟基-L-鸟氨酸,N5-烷基-N5-羟基-L-鸟氨酸是羟肟酸铁载体合成的基本单位。多个N5-烷基-N5-羟基-L-鸟氨酸基本单位通过非核糖体肽键合成酶(NRPSs)的缩合作用形成含有多个肽键的链状或环状化合物,如玫红酵母酸(Rhodotorulic acid)等,如图3所示。
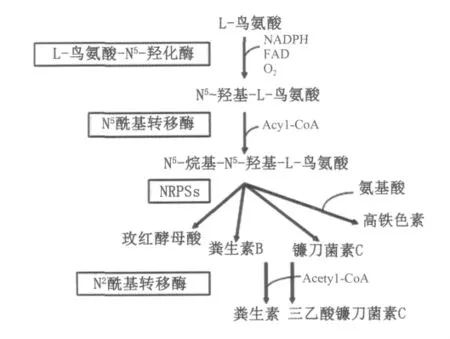
图3 真菌铁载体合成的基本过程Fig.3 The biosynthesis of siderophore in fungi
在真菌中参与由铁引起的阻遏作用的蛋白是转录阻遏蛋白,叫做GATA蛋白,这些蛋白含有GATA类型的锌指结构,从而可以与铁载体合成和铁吸收有关基因的启动子结合。至今已经发现的Ustilago maydis URBS1,N.crassa SRE,P.chrysogenumSREP,A.nidulans SREA 和 Schizosaccharomyces pombe GAF2p都被鉴定为GATA蛋白,这类蛋白在大多数真菌中参与了铁载体合成和运输的负调控作用,它们都有一对锌指结构域,以便与有关基因启动子中含有HGATAR序列的顺式片段(cis-elements)结合(其中H代表碱基A,T或C,R代表任何一个嘌呤)。比如在S.pombe中Fep1可以结合在该酵母所有已知编码铁吸收蛋白基因,包括frp1(还原酶),fio1(铁氧化还原酶),fip1(渗透酶),和铁受体基因str1-3的5′-非编码区5′-(A/T)GATAA-3′序列上,如图4所示。但是这些GATA蛋白如何与细胞中铁结合后才能结合DNA或蛋白质上或铁离子的存在如何影响到这些GATA蛋白与有关DNA结合现在还不清楚。有实验结果表明纯化的重组Fep1蛋白在有铁络合物存在情况下不能结合在含有HGATAR序列的顺式片段上,所以说Fep1与DNA结合是依赖于铁离子的存在。也有人认为这类蛋白的锌指结构能直接与铁结合或通过铁-硫簇(Iron-sulphur cluster)与铁结合。根据上述5种阻遏蛋白(fep1,SRE,SREA,Urbs1和Sfu1)共有的氨基酸序列G(S/T)CPG(D/G)GXCNTGG,通过进一步分析发现它们的核心序列是GSCPGDGLCNGTGG,这是这些依赖铁进行转录阻遏的蛋白特有的保守序列。在某些情况下,这些蛋白必须与共阻遏蛋白,如图4所示的Tup11或Tup12一起才能发挥功能,所以这些GATA蛋白是作为多蛋白复合物的一部分发挥功能,如图4所示。
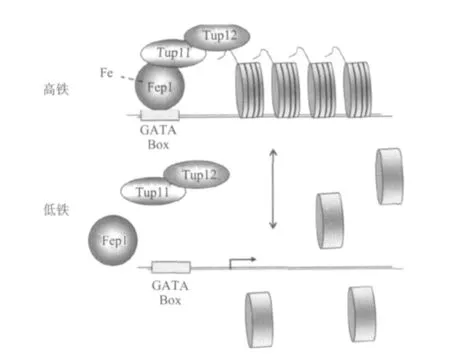
图4 通过GATA蛋白Fep1进行转录调控的模式图Fig.4 Schema chart of the transcriptional regulation through GATA protein Fep1
Fep1是裂殖酵母菌中许多基因的1种转录共阻遏蛋白,负责铁吸收的调控作用。这类蛋白能通过一对锌指结构域特异地与顺式片段(GATA Box)结合,这种结合作用是在铁过量情况下进行的,结合后与铁载体合成和铁运输有关的基因转录和蛋白合成就受到阻遏。当培养基缺铁时,Fep1离开GATA Box,与铁载体合成和铁运输有关的基因转录和蛋白合成就开始合成。
本实验室从海洋酵母菌种库的300多株海洋酵母中利用高氯酸铁检测的方法筛选得到14株产铁载体的酵母,经过复筛得到一株铁载体产量较高的海洋酵母菌菌株HN6.2,该菌株分离自青岛东风盐场近海表层海水。通过鉴定,发现该株海洋酵母菌属于普鲁兰类酵母(Aureobasidium pullulans)。在最佳条件下培养,HN6.2菌株的铁载体最高产量可达到1.1mg/mL。在产铁载体培养基中加入L-鸟氨酸后铁载体的产量明显增加,而加入Fe3+后,其产量明显减少。并且,从分子水平上检测到了L-鸟氨酸-N-羟基化酶基因在加有L-鸟氨酸培养基中表达量上升,而加有Fe3+情况下该基因表达量非常低。该菌株HN6.2产生的铁载体对海洋鳗弧菌V.P、海洋副溶血弧菌 W-1和枯草杆菌具有明显的抑菌活性,并且对海洋鳗弧菌V.P的抑菌活性最大。同时,发现Fe3+对铁载体的抑菌作用具有抑制作用。该铁载体的热稳定性很强,对铁载体进行加热处理后,仍具有同样的抑菌活性[15]。经过纯化、红外光谱和质谱法分析,发现该铁载体的结构组分是异羟肟酸类Fusigen,如图5所示[16]。纯化的铁载体仍具有抑菌活性。
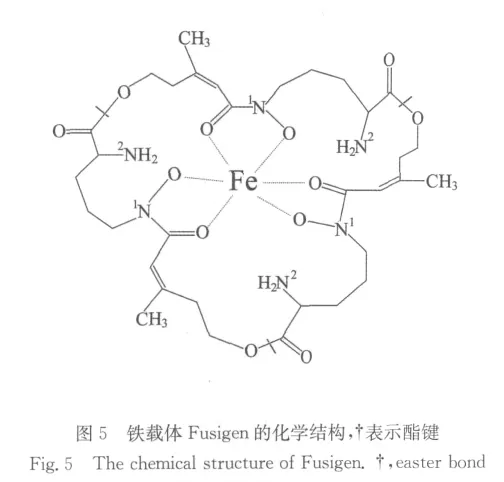
从图5的化学结构可以看出Fusigen合成的第一步反应也是L-鸟氨酸的5位N原子发生羟基化形成N5-羟基-L-鸟氨酸,但是在该铁载体合成过程中 N5-羟基-L-鸟氨酸不发生烷基化反应,而是 N5-羟基-L-鸟氨酸直接作为铁载体合成的基本单位。3个N5-羟基-L-鸟氨酸基本单位通过非核糖体肽键合成酶(NRPSs)的缩合作用形成含有3个酯键的环状化合物(见图5)。目前该L-鸟氨酸-N-羟基化酶基因包括上游的非编码区和下游的终止序列已经得到了克隆,命名为SidA基因,登录号为FJ769160。该基因的ORF框大小为1 461bp,编码1个由486个氨基酸组成的蛋白质(等电点7.79),推测的分子量为55.4kD。该基因(无内含子)启动子位于-1~-824bp,并且具有3个HGATAR boxes和1个CATA box,推测前者就是阻遏蛋白(GATA蛋白)结合的结构域,所以就出现了上述的由铁离子引起的铁载体合成和L-鸟氨酸-N-羟基化酶基因表达的阻遏现象。把该基因敲除后获得的敲除菌株S6(ΔsidA)不能合成胞内和胞外的铁载体,所以培养的上清液不能抑制病原菌Vibrio anguillarum 和Vibrio parahaemolyticus的生长。敲除菌株S6在完全缺铁的培养基上和海水中因为细胞出芽停止不能生长,但是在培养基添加上10μmol/L Fe3+和Fe2+后就可以生长。与野生型菌株相比,培养基中的H2O2对敲除菌株的毒性要大得多,因为在细胞中没有铁载体来储存铁,这样过多的铁会导致过量过氧化合物的形成。所以推测该酵母产生的铁载体fusigen在络合、吸收和浓缩铁方面能发挥独特的作用,以维持细胞某些特殊的生理功能。另外该酵母能分泌具有抑制其他微生物生长的铁载体,这样可以排除竞争者,在海洋环境中能更好地获得有限的营养物[17]。
5 氮阻遏机理的研究现状
在酿酒酵母中所有与氮代谢(包括谷氨酰氨、谷氨酸、脯氨酸、尿素、精氨酸、含氮的γ-氨基丁酸(GABA)和尿囊素合成)有关的已知途径受到了4个调节蛋白Gln3,Gat1,Dal80和Deh1的调控,这些蛋白可以结合到受调控基因启动子的共有序列5′-GATAA-3′上。Gln3和Gat1结合有利于有关基因的表达,所以它们是转录激活蛋白,而Dal80和Deh1的结合不利于有关基因的表达,所以它们是转录阻遏蛋白。另外氨基酸渗透酶和氨渗透酶基因的表达也受到这4个蛋白的调控。该酵母蛋白酶基因的表达同样也受到Gln3,Gat1,Dal80和Deh1的调节,因为这些基因编码的蛋白酶可以把蛋白质降解成氨基酸,从而给细胞提供氮源,这样培养基存在这些氨基酸同样也会产生氮阻遏和解阻遏作用。谷氨酸和谷氨酰氨是细胞合成所有氨基酸的前体物。当培养基中不存在谷氨酰氨、天冬氨酸或氨时,编码合成脯氨酸、尿素、精氨酸、含氮的γ-氨基丁酸(GABA)和尿囊素合成的基因表达可以在转录水平上得到解阻遏,因为细胞无法直接从谷氨酰氨、天冬氨酸或氨合成其他的氨基酸。Gln3含有730氨基酸,在306~330氨基酸区域中有1个Cys2/Cys2锌指结构,它能结合在含有5′-GATA/TA-3′Gln3DNA 序列中,把这段序列叫做上游激活序列UASNTR。在含有谷氨酰氨、天冬氨酸或氨的阻遏培养基中,Gln3的功能受到阻遏,其中谷氨酰氨是引起阻遏的信号分子,当天冬氨酸或氨被转化成谷氨酰氨时能起同样的作用。Gat1是能引起氮阻遏途径基因解阻遏的第二个激活蛋白,像Gln3蛋白一样,Gat1也含有1个与DNA结合的锌指结构,但是当培养基中谷氨酸浓度增加时Gat1失去活力,而氨浓度增加时Gat1具有活力。像Gln3蛋白一样,作为1种阻遏蛋白Dal80能结合在启动子含有5′-GATAA-3′的DNA序列中;这些DNA序列被命名为上游阻遏序列URSGATA。编码Dal80的基因DAL80含有807个碱基,编码269个氨基酸。如果氮代谢途径的基因启动子含有URSGATA序列,Dal80能够阻遏这些基因表达。如果启动子中的2个5′-GATAA-3′序列是尾对尾或头对头相连,那么 Dal80结合在URSGATA上最为牢固。基因DAL80的表达依赖于Gln3,在含有脯氨酸解阻遏培养基中它会阻遏它自己的表达。在含有谷氨酰氨、天冬氨酸和氨的阻遏培养基中它不会影响GLN3,GAT1或DEH1基因的表达,但是在解阻遏培养基中会影响GLN3,GAT1或DEH1基因的表达。与Dal80一样,在含有谷氨酰氨、天冬氨酸和氨的丰富培养基(阻遏培养基)中产生的Deh1是一种阻遏蛋白,能阻遏与氮代谢过程中的许多基因表达。它含有551个氨基酸,与Dal80有很高的同源性:C-末端有1个假定的亮氨酸拉链结构(Putative leucine zipper)和锌指结构,这些锌指结构与其他真菌和人类的Gln3,Gat1,Dal80和各种GATA因子有很高的同源性。与Gln3和Gat1所不同的是,Deh1和Dal80没有酸性区域,所以不能参与转录激活作用[3]。
上述能与GATA结合的蛋白质是氮代谢物途径中的4种主要调节蛋白,所有这些蛋白具有很高的同源序列,都能辨认启动子中的5′-GATAA-3′的序列。Dal80和 Deh1与启动子中的5′-GATAA-3′序列结合后,能拮抗Gln3和Gat1的结合,所以阻遏了许多基因的转录,所有这些调节蛋白都存在于阻遏和解阻遏培养基中,但是在解阻遏培养基中Gln3和Dal80是主要的调节蛋白,而在阻遏培养基中Gat1和Deh1是主要的调节蛋白。通过这种方式完成完全阻遏(含有谷氨酰氨)到完全解阻遏(含有脯氨酸)过程[3]。
6 诱导机理的研究现状
与阻遏现象一样,酵母菌许多酶的合成和代谢途径都会受到底物的诱导。现在认为在酵母菌诱导过程中,转录激活蛋白(Transcriptional activator)起着非常关键的作用,转录激活蛋白最重要的特征是含有锌指结构,能与许多基因的启动子结合,起转录激活作用。这类蛋白还有一个很重要的特征就是有特异性,就是一种转录激活蛋白只能调控一种物质的代谢。它们可以分为3类,第一类是C2H2蛋白,这类蛋白共有的氨基酸序列是 Cys-X2-4-Cys-X12-His-X3-5-His;第二类是C4蛋白,这类蛋白共有的氨基酸序列如下:Cys-X2-Cys-Xn-Cys-X2-Cys-Xn-Cys-X2-Cys-Xn-Cys-X2-Cys;第三类是C6蛋白,这类蛋白共有的氨基酸序列如下:Cys-X2-Cys-X6-Cys-X5-12-Cys-X2-Cys-X6-8-Cys, 这类锌指蛋白含有6个半胱氨酸和2个锌原子,所以通常被称作为Zn(II)2Cys6或者(Zn2C6),它们是真菌中特有的转录激活蛋白。转录激活蛋白二级结构变化非常大,并且功能多样。指状结构实际上是由1个α-螺旋和1对反平行β-折叠链组成的。通常情况下,有1个或多个锌原子与氨基酸Cys或His结合,这种结合有利于结构域的稳定性,并且对该类蛋白有合适的结构和功能具有重要的作用[18]。
转录激活蛋白除了具有富含半胱氨酸的DNA结合结构域(DNA binding domains,DBD)外,还有调节和激活结构域,如图6所示。转录激活蛋白的功能结构域包括锌指(Zinc finger)、接头(Linker)、二聚体区域(Dimerization)、调节结构域(Regulatory domain)和酸性区域(激活结构域)(Aciddic region),其中锌指、接头和二聚体区域3部分统称为DNA结合结构域DBD。锌指一般位于N-末端,负责与被诱导基因启动子的结合,它所辨认的启动子一般含有CGG三联密码子,当然该三联密码子附近的核苷酸也会在某种程度上影响DBD与DNA结合的亲和力,另外该三联密码子的方向性和三联密码子之间的空间对于DBD与DNA结合也有影响。接头决定了DBD与被诱导基因启动子结合的特异性。二聚体区域决定了2个转录激活蛋白同时结合到被诱导基因启动子上,并且对蛋白质-蛋白质相互作用起某种作用。调节结构域在调节转录激活蛋白的转录活力方面起某种作用。酸性区域通常位于C-末端,主要的功能是作为激活结构域,它们不是很保守的结构域,并且结构功能多样[18]。
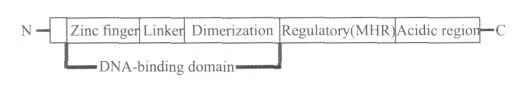
图6 转录激活蛋白的功能结构域Fig.6 The functional domain of transcriptional activator
在有诱导物存在情况下,酵母菌转录激活蛋白如何激活有关基因的表达现在还不完全清楚。这方面研究最多的是酒精酵母中与半乳糖代谢有关的Gal4p这种转录激活蛋白。这种激活蛋白可以激活与半乳糖和蜜二糖代谢有关的一系列基因表达,如GAL1,GAL2,GAL7,GAL10和MEL1。在没有DNA存在情况下,Gal4p是以单体的形式存在,当它的锌指与DNA含有CCG三联密码子结合时是以二聚体的形式存在。Gal4p可以与调节蛋白Gal80形成一个复合物,这种复合物的形成需要Gal4p蛋白C-末端存在的30个氨基酸[2,18]。
在培养基没有半乳糖存在情况下,这种复合物可以与受诱导基因的上游激活序列UASGAL结合,但是这时不能激活转录作用(见图7A)[2,18]。
当培养基中存在半乳糖时,一种类似于半乳糖激酶的调控蛋白Gal3p首先与半乳糖结合,然后再与Gal4p-Gal80复合物结合,从而解除了Gal80对Gal4p活力的抑制作用,有关的基因转录便开始(见图7B)。Gal3p-半乳糖复合物能影响GAL基因的表达是由于这种复合物能使Gal4p-Gal80p复合物发生构象变化,从而使Gal4p的激活结构域能和普通的转录因子发生相互作用。在没有半乳糖存在情况下,Gal80可以通过改变激活结构域而抑制 Gal4p活力[2,18]。
Gal4p蛋白上的许多氨基酸可以得到磷酸化反应。在不受诱导情况下,磷酸化和去磷酸化的Gal4p形式都可以存在于细胞中。在有诱导物半乳糖存在情况下,Gal4p其中之一的氨基酸(Ser-699)出现磷酸化后,这时Gal4p具有激活有关基因的转录作用,Ser-699氨基酸发生磷酸化作用对于Gal4p发生最大的转录激活作用能起关键的作用(见图7C)。RNA Pol II全酶中的细胞周期蛋白依赖性激酶(The cyclin-dependent protein kinases of the RNA Pol II holoenzyme)参与了这种磷酸化作用。在上述磷酸化过程中,还有一个蛋白还参与了作用,那就是Srb10p,在Srb10p有活力情况下,Gal4p的Ser-699才能发生磷酸化作用,从而使Gal4p-Gal80p复合物处在有活力的构象,如果Srb10p能使Gal4p发生磷酸化作用,那么Srb10p就可以加快GAL基因的诱导作用(见图7C)[2,18]。现在的问题是半乳糖的存在如何使Gal4p的Ser-699发生磷酸化反应,其中有可能发生信号传导作用。
当培养基中存在葡萄糖时,这时培养基中即使有半乳糖存在,与半乳糖代谢有关的GAL基因转录也会受到阻遏。葡萄糖可以在不同水平上作用于Gal4p,通过Mig1蛋白质阻遏它的合成,如上述、阻止它与上游激活序列UASGAL的结合、干扰Gal4p的激活功能。另外还会干扰半乳糖-Gal3-Gal4p-Gal80复合物抑制作用的解除。另外一种可能是由于在有Gal80存在情况下,葡萄糖可以抑制Gal4p发生磷酸化作用[2,18]。
在非诱导情况下(没有加半乳糖情况下),转录激活蛋白Gal4p活力受到负调控蛋白Gal80p的抑制(A)。当加入半乳糖后(B),半乳糖与Gal3p结合成复合物,该复合物与Gal80p相互作用导致Gal4p构象发生瞬间变化,使得Gal4p转录激活蛋白具有激活转录作用。在与RNA Pol II全酶作用过程中(C),转录激活蛋白Gal4p的Ser-699受到Srb10p的磷酸化作用,这种磷酸化作用使得受Gal3p-半乳糖诱导的Gal4p-Gal80p构象得到激活,这样激活有关基因的表达。转录激活蛋白Gal4p中的Ser-699受到Srb10p的磷酸化作用是受到单独的环境信号调[2,18]。
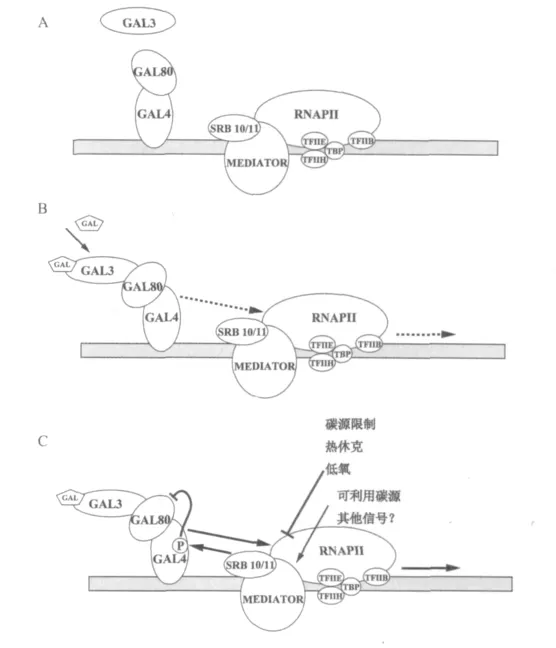
图7 酿酒酵母半乳糖酶的诱导机理Fig.7 The induction mechanism of GAL in S.cerevisae
7 展望
从上述情况来看,目前仅对酿酒酵母诱导机理有所了解。同时发现参与诱导作用的关键组分转录激活蛋白的基因表达也受到葡萄糖阻遏。转录阻遏蛋白Mig1可以调控细胞中许多受葡萄糖阻遏的基因表达,但是转录激活蛋白对调控的基因具有特异性,比如说Gal4p只能调控酿酒酵母半乳糖和蜜二糖的代谢,对其他物质的代谢没有调控作用。目前许多酵母菌糖代谢和活性物质代谢也受到了诱导调控,如上述的扣囊复膜酵母菌淀粉酶合成、纤维二糖酶合成和酸性蛋白酶合成分别受到淀粉、糊精、麦芽糖、纤维二糖和蛋白质的诱导、马克斯克鲁氏酵母菌乳糖酶合成、菊糖酶合成和纤维二糖酶合成受到乳糖、半乳糖、菊糖、蔗糖、纤维二糖的诱导、另有一株普鲁兰酵母铁载体合成受鸟氨酸诱导。这些酵母的诱导机理是否与酒精酵母的Gal4p调控作用一样,现在还一无所知。这就需要人们在这些酵母菌克隆更多的转录激活蛋白基因,并通过基因敲除实验验证转录激活蛋白基因与诱导的关系。这些转录激活蛋白与基因启动子的结合是否需要淀粉、糊精、麦芽糖、蛋白质、纤维二糖、乳糖、半乳糖、菊糖、蔗糖和鸟氨酸的参与;在培养基中的淀粉、糊精、麦芽糖、蛋白质、纤维二糖、乳糖、半乳糖、菊糖、蔗糖和鸟氨酸与转录激活蛋白的磷酸化和有关基因的表达存在何种关系;转录激活蛋白基因的表达与葡萄糖阻遏之间的关系如何;在不同酵母菌中和不同物质引起的诱导机理是否一样;在不同转录激活蛋白中哪一种对有关的基因表达诱导最有效;过量表达这种转录激活蛋白是否可以增加有关酶的产量,所有这些问题都是将来需要进行深入的研究。同样许多阻遏机理也完全未知,如上述在铁载体合成过程中由GATA蛋白参与的阻遏作用是否需要铁离子存在,铁离子如何与GATA蛋白结合,然后发挥阻遏有关基因表达的作用。
[1] 沈萍,陈向东.微生物学 [M].北京:高等教育出版社,2006:238-243.
[2] Gancedo J M.Yeast carbon catabolite repression[J].Microbiology and Molecular Biology Reviews,1998,62:334-361.
[3] Hofman-Bang J.Nitrogen catabolite repression in Saccharomyces cerevisiae[J].Molecular Biotechnology,1999,12:35-73.
[4] Coffman J A,Rai R,Cunningham T,et al.Gat1p,a GATA family protein whose production is sensitive to nitrogen catabolite repression,participates in transcriptional activation of nitrogencatabolicgenes in Saccharomyces cerevisiae [J].Molecular and Cellular Biology,1996,16:847-858.
[5] Coffman J A,Rai R,Loprete D M,et al.Cross regulation of four GATA factors that control nitrogen catabolic gene expression in Saccharomyces cerevisiae [J].Journal of Bacteriology,1997,179:3416-3429.
[6] Cooper T G.Transmitting the signal of excess nitrogen in Saccharomyces cerevisiae from the Tor proteins to the GATA factors:connecting the dots[J].FEMS Microbiology Reviews,2002,26:223-238.
[7] Courchesne W E,Magasanik B.Regulation of nitrogen assimilation in Saccharomyces cerevisiae:roles of the URE2and GLN3 genes[J].Journal of Bacteriology,1988,170:708-713.
[8] Cox K H,Rai R,Distler M,et al.Saccharomyces cerevisiae GATA sequences function as TATA elements during nitrogen catabolite repression and when Gln3p is excluded from the nucleus by overproduction of Ure2p[J].Journal of Biological Chemistry,2000,275:17611-17618.
[9] Cunningham T S,Andhare R,Cooper T G.Nitrogen catabolite repression of DAL80expression depends on the relative levels of Gat1p and Ure2p production in Saccharomyces cerevisiae[J].Journal of Biological Chemistry,2000,275:14408-14414.
[10] Cunningham T S,Cooper T G.Expression of the DAL80gene,whose product is homologous to the GATA factors and is a negative regulator of multiple nitrogen catabolic genes in Saccharomyces cerevisiae,is sensitive to nitrogen catabolite repression [J].Molecular and Cellular Biology,1991,11:6205-6215.
[11] Cunningham T S,Cooper T G.The Saccharomyces cerevisiae DAL80repressor protein binds to multiple copies of GATAA-containing sequences(URSGATA)[J].Journal of Bacteriology,1993,175:5851-5861.
[12] Cunningham T S,Dorrington R A,Cooper T G.The UGA4 UASNTRsite required for GLN3-dependent transcriptional activation also mediates DAL80-responsive regulation and DAL80protein binding in Saccharomyces cerevisiae [J].Journal of Bacteriology,1994,176:4718-4725.
[13] Cunningham T S,Rai R,Cooper T G.The level of DAL80expression down-regulates GATA factor-mediated transcription in Saccharomyces cerevisiae [J].Journal of Bacteriology,2000,182:6584-6591.
[14] Deberardinis R J,Lum J J,Hatzivassiliou G,et al.The biology of cancer:metabolic reprogramming fuels cell growth and proliferation[J].Cell Metabolism,2008,7:11-20.
[15] Feller A,Boeckstaens M,Marini A M,et al.Transduction of the nitrogen signal activating Gln3-mediated transcription is independent of Npr1kinase and Rsp5-Bul1/2ubiquitin ligase in Saccharomyces cerevisiae [J].Journal of Biological Chemistry,2006,281:28546-28554.
[16] Chi Z M,Li J F,Wang X H,et al.Inositol and phosphatidylinositol mediated glucose derepression,gene expression and invertase secretion in yeasts[J].Acta Biochimica et Biophysica Sinica,2004,36:443 449.
[17] Yao S,Chi Z M,He S.Studies on inositol-mediated expression of MAL gene encoding maltase and phospholipid biosynthesis in Schizosaccharomyces pombe[J].Journal of Industrial Microbiology and Biotechnology,2006,33:417-422.
[18] Yao S,Chi Z M,He S.Regulation of MAL1+gene expression encoding maltase in Schizosaccharomyces pombe by added inositol[J].Indian Journal of Biochemistry and Biophysics,2006,43:143-147
[19] Chi Z,Ma L,Gao L,et al.Added inositol regulates invertase secretion and glucose-repressed SUC2gene expression in Saccharomyces sp.W4[J].Indian Journal of Biochemistry and Biophysics,2007,44(3):106-113.
[20] Chi Z M,Ni X,Yao S.Cloning and overexpression of a maltase gene from Schizosaccharomyces pombe in Escherichia coli and characterization of the recombinant maltase [J].Mycol Res,2008,112:983-989.
[21] Chi Z M,He S,Yao S.Effects of Pichia pastoris INO1expression in Schizosaccharomyces pombe on phosphatidylinositol(PI)synthesis and expression of INV+encoding invertase [J].Enzyme and Microbial Technology,2005,37:395-401.
[22] Chi Z M,Chi Z,Liu G,et al.Saccharomycopsis fibuligera and its applications in biotechnology [J].Biotechnology Advances,2009,27:423-431.
[23] Cassart J P,Ostling J,Ronne H,et al.Comparative analysis in three fungi reveals structurally and functionally conserved regions in the Mig1repressor [J].Molecular and General Genetics,1997,255:9-18.
[24] Liu G L,Wang D S,Wang L F,et al.Mig1is involved in mycelial formation and expression of the genes encoding extracellular enzymes in Saccharomycopsis fibuligera A11 [J].Fungal Genetics and Biology,2011,48:904-913.
[25] Lachance M A.Kluyveromyces van der Walt em end.van der Walt[M].Netherlands:Amsterdam,Elsevier,1998:227-247.
[26] Llorente B,Malpertuy A,Blandin G,et al Genomic exploration of the hemiascomycetous yeasts,12.Kluyveromyces marxianus var.marxianus[J].FEBS Letters,2000,487:71-75.
[27] Lane M M,Morrissey J P.Kluyveromyces marxianus:A yeast emerging from its sister′s shadow [J].Fungal Biology Reviews,2010,24(2):1-10.
[28] Haas H.Molecular genetics of fungal siderophore biosynthesis and uptake:the role of siderophores in iron uptake and storage[J].Applied Microbiology and Biotechnology,2003,62:316-330.
[29] Hider R C,Kong X L.Chemistry and biology of siderophores[J].Natural Product Reports,2010,27:637-657.
[30] Scazzocchio C.The fungal GATA factors[J].Current Opinion In Microbiology,2000,3(2):126-131.
[31] Harrison K A,Marzluf G A.Characterization of DNA binding and the cysteine rich region of SRE,a GATA factor in Neurospora crassa involved in siderophore synthesis [J].Biochemistry,2002,41:15288-15295.
[32] An Z,Mei B,Yuan W M,et al.The distal GATA sequences of the sid1promoter of Ustilago maydis mediate iron repression of siderophore production and interact directly with Urbs1,a GATA family transcription factor[J].EMBO Journal,1997,16:1742-1750.
[33] Zhou L,Marzluf G A.Functional analysis of the two zinc fingers of SRE,a GATA-type factor that negatively regulates siderophore synthesis in Neurospora crassa [J].Biochemistry,1999,38:4335-4341.
[34] Renshaw J C,Robson G D,Trinci A P J,et al.Fungal siderophores:structures,functions and applications[J].Mycological Research,2002,106:1123-1142.
[35] Oberegger H,Schoeser M,Zadra I,et al.SREA is involved in regulation of siderophore biosynthesis,utilization and uptake in Aspergillus nidulans[J].Molecular Microbiology,2001,41:1077-1089.
[36] Martins L J,Jensen L T,Simon J R,et al.Metalloregulation of FRE1and FRE2homologs in Saccharomyces cerevisiae [J].Journal of Biological Chemistry,1998,273:23716-23721.
[37] Wang W,Chi Z,Liu G,et al.Chemical and biological characterization of siderophore produced by the marine-derived Aureobasidium pullulans HN6.2and its antibacterial activity [J].Biometals,2009,22:965-972.
[38] Wang W L,Chi Z M,Chi Z,et al.Siderophore production by the marine-derived Aureobasidium pullulans and its antimicrobial activity[J].Bioresource Technology,2009,100:2639-2641.
[39] Chi Z,Wang X X,Ma Z C,et al.The unique role of siderophore in marine-derived Aureobasidium pullulans HN6.2 [J].Biometals,2012,25:219-230.
[40] Johnston M,Flick J S,Pexton T.Multiple mechanisms provide rapid and stringent glucose repression of GAL gene expression in Saccharomyces cerevisiae [J].Molecular and Cellular Biology,1994,14,3834-3841.
[41] Li J,Wang S,VanDusen W J,et al.Green fluorescent protein in Saccharomyces cerevisiae:real-time studies of the GAL1promoter[J].Biotechnology and Bioengineering,2000,70:187-196.
[42] Rohde J R,Trinh Sadowski.Multiple signals regulate GAL transcription in yeast[J].Molecular and Cellular Biology,2000,20(11):3880-3886.
[43] Vodala S,Abruzzi K C,Rosbash M.The nuclear exosome and adenylation regulate posttranscriptional tethering of yeast GAL genes to the nuclear periphery [J].Molecular Cell,2008,31:104-113.
[44] Luthra R,Kerr S C,Harreman M T,et al.Actively transcribed GAL genes can be physically linked to the nuclear pore by the SAGA chromatin modifying complex [J].Journal of Biological Chemistry,2007,282:3042-3049.
[45] Kumar P R,Yu Y,Sternglanz R,et al.NADP regulates the yeast GAL induction system [J].Science,2008,319:1090-1092.

