ITS片段作为轮枝菌DNA条形码的评价研究
段维军, 张慧丽, 郭立新, 顾建锋, 陈先锋, 张祥林, 夏侯阳
(1.宁波出入境检验检疫局,宁波 315012;2.新疆出入境检验检疫局,乌鲁木齐 830063;3.宁波大学,宁波 315212)
植物病原性轮枝菌主要包括:黑白轮枝菌[Verticillium albo-atrum Reinke et Berthold(1879)]、大丽轮枝 菌 [V.dahliae Klebahn (1913)]、三 体 轮 枝 菌[V.tricorpus Isaac(1953)]、变黑轮枝菌[V.nigrescens Pethybridge(1919)]、云状轮枝菌[V.nubilum Pethybridge (1919)]、V.the obromae (Turconi)Mason et Hughes(1951)[1]以 及 长 孢 轮 枝 孢 [V.longisporum(Stark)Karapapa,Bainbr & Heale(1997)],也被称为大丽轮枝菌长孢变种[V.dahliae var.long is porum Stark(1961)][2]。它们多能引起植物维管束病害,造成植物叶片黄化、落叶等症状,严重时导致植物死亡。这些轮枝菌可产生多种休眠形态,如微菌核、厚垣孢子或休眠菌丝等,长时间在土壤中存活。由于其严重危害性,其中的一些种类,如大丽轮枝菌和黑白轮枝菌已经被列为我国重要植物检疫对象。这些轮枝菌均能够产生典型的轮枝状分生孢子梗,形态十分接近,单纯依靠形态学特征很难加以准确快速区分。
DNA条形码技术(DNA Barcoding)是利用一个或少数几个DNA片段对地球上现有物种进行识别和鉴定的一项新技术[3]。自加拿大生物学家Hebert首先倡导将条形码编码技术应用到比零售业更复杂的生物物种鉴定之后[4-5],发展十分迅猛,是近年来进展最迅速的学科前沿之一,在动物、植物等多种生物上得到了广泛重视。真菌的核糖体RNA基因簇同时存在于细胞核内与线粒体中,由高度保守区和可变区组成,以其为代表的ITS基因标记已被广泛用于构建系统发育树、预测种群结构、评估种群进化过程,生态学研究以及确定物种分类地位等[6-11],同时也被考虑作为DNA条形码。但是,由于一些真菌类群平均ITS片段小于有效进行DNA编码的最优片段临界值[12],同时对于物种相对丰富的子囊菌类群来说 ITS区段还存在变异性不足,难以有效区分不同种类的缺点。因此,在适于作真菌DNA条形码的基因区段的选择上,还需做更多研究和评估工作。
本研究选择ITS片段作为轮枝菌的候选DNA条形码,对候选ITS片段的PCR扩增和测序成功率、种内与种间差异、种内与种间距离的频率分布进行系统筛选和评价,以此来评估ITS片段作为轮枝菌条形码的可行性。
1 材料与方法
1.1 材料
1.1.1 供试菌株的分离、收集与纯化
采用常规的组织块PDA平板分离法及菌种收集工作,共得到植物病原性轮枝菌35株,分属于13个种,进行单孢分离后得到单孢分离株,保存在PDA试管斜面(4℃),备用。分离株编号、地理分布、寄主及来源见表1。
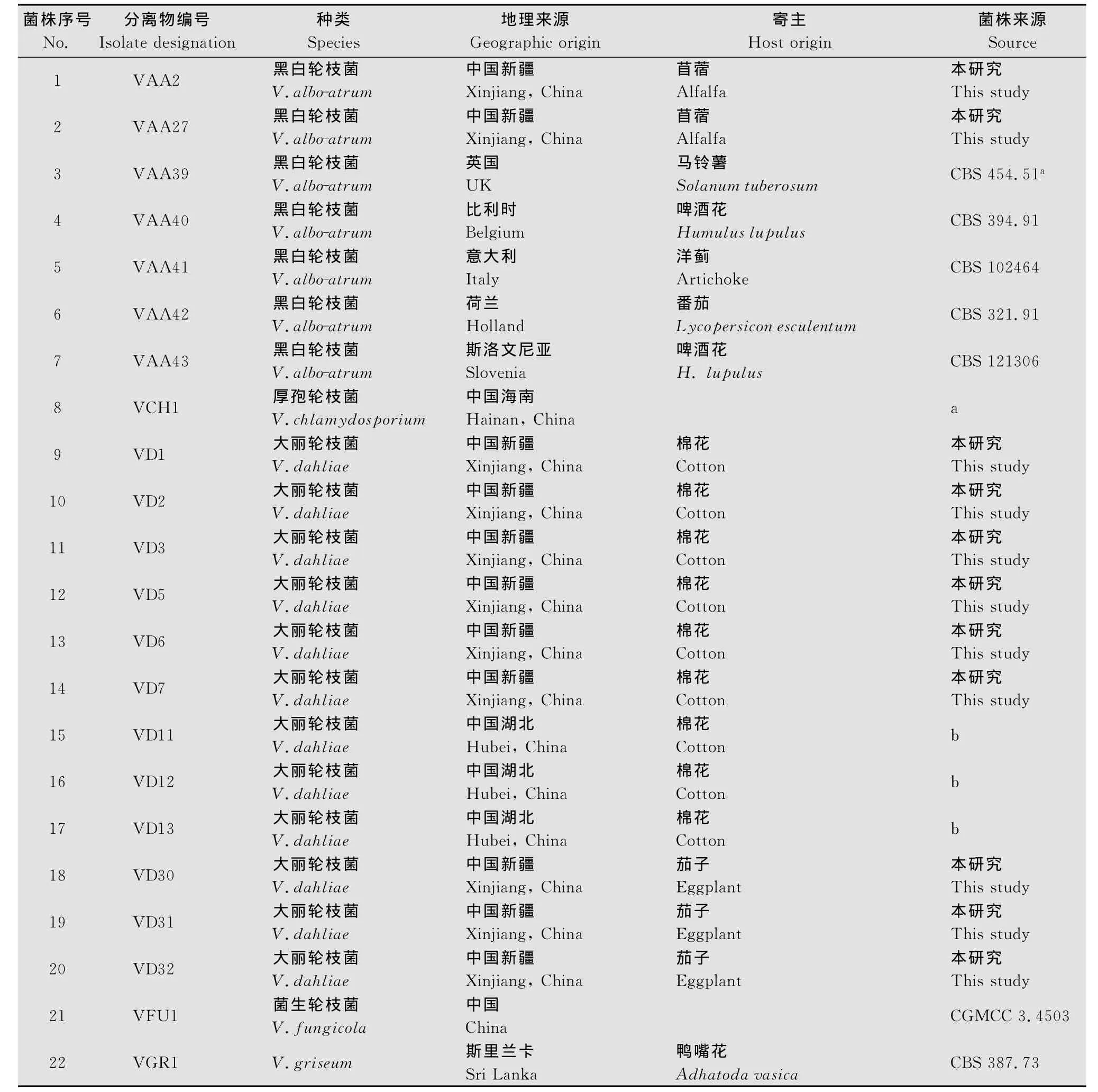
表1 供试轮枝菌属分离株Table 1 Isolates of Verticilliumspp.used in this study
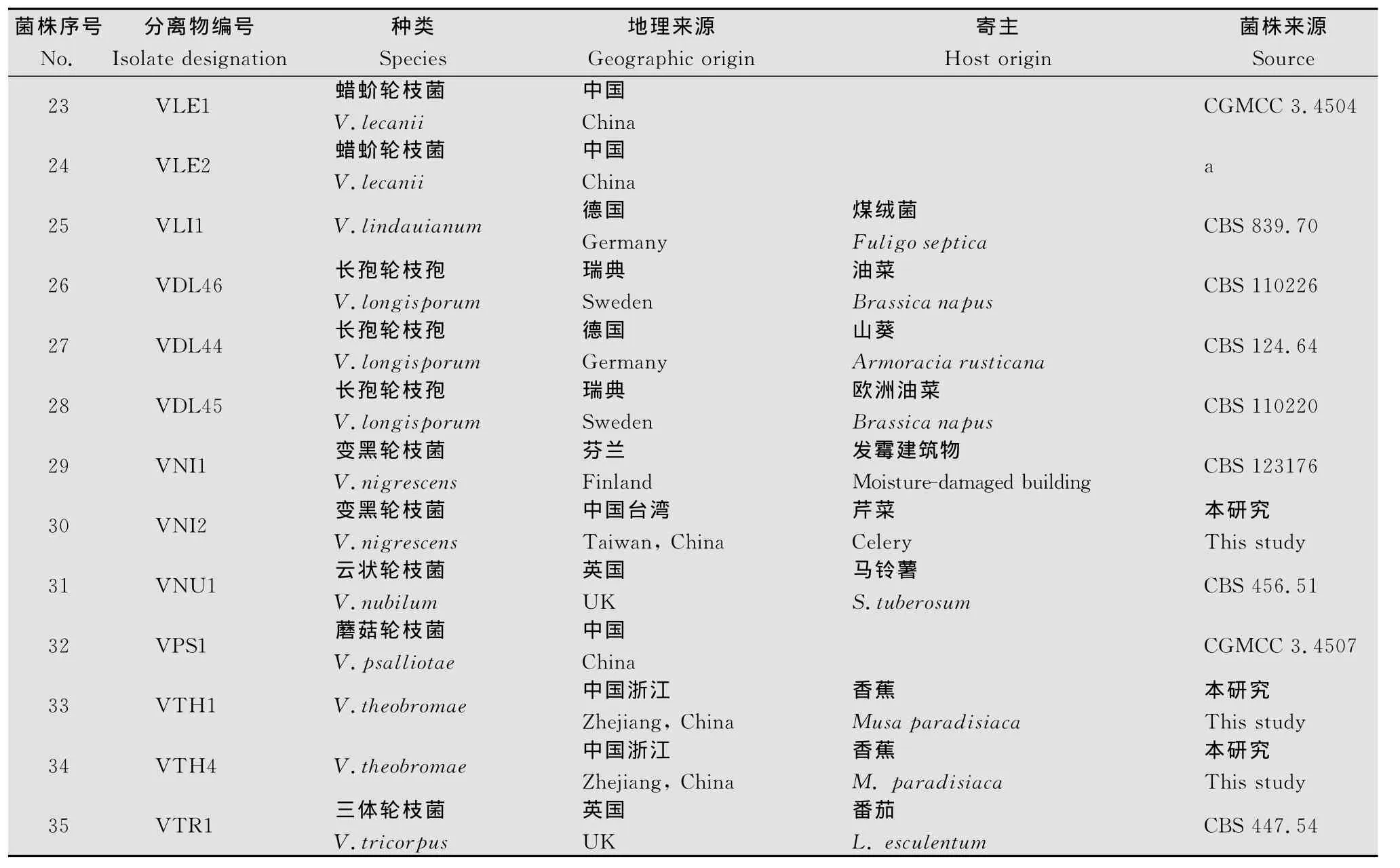
续表1 Table1(Continued)
1.1.2 培养基
采用马铃薯葡萄糖琼脂(PDA)培养基进行分离、纯化、菌丝收集和保藏。
1.1.3 仪器和试剂
培养箱为德国MMM公司生产的Friocell 222型,核酸自动化提取仪为美国ThermoFisher公司生产的Kingfisher型,PCR仪为Biometra公司生产的TProfessional Basic型,生物分光光度计为日本日立公司生产的U-0080D型,电泳设备和凝胶成像系统分别为伯乐生命医学产品有限公司生产的Power-Pac Basic型和GelDocEQ型;核酸提取试剂盒为台湾奈米技术开发有限公司生产的TANBead Plant DNA Auto Kit型、Taq DNA聚合酶、MgCl2、10×PCR buffer、DL2000Marker、DNA片段纯化试剂盒均购自宝生物(大连)工程有限公司;试验用引物由上海英俊生物技术有限公司合成;其他试剂均为分析纯。
1.2 方法
1.2.1 基因组DNA提取
用灭菌枪头刮取在PDA平板上培养20d左右菌株菌丝体,根据试剂盒及仪器使用说明,在核酸自动化提取仪上使用核酸提取试剂盒(台湾奈米技术开发有限公司)进行DNA提取。提取后DNA经分光光度计检测DNA浓度后,冻存于-20℃冰箱备用。
1.2.2 基因的扩增和测序
采用真菌通用引物ITS1和ITS4进行扩增[16]。PCR扩增在TProfessional Basic PCR仪上进行,使用50μL体系。PCR扩增产物用电泳仪在2%琼脂糖凝胶上进行检测,确认扩增成功后,使用凝胶成像系统进行拍照,并用DNA片段纯化试剂盒根据试剂盒说明书进行纯化。将纯化的PCR产物送上海英俊生物技术有限公司进行双向测序。
1.2.3 ITS片段用于轮枝菌属DNA条形码的评价
1.2.3.1 获得ITS片段的难易程度
不同基因序列获得的难易程度用PCR扩增与测序成功率表示,高质量的测序图谱,底峰较小,无杂峰、乱峰视为成功测序。PCR扩增成功率和测序成功率的乘积为PCR扩增与测序成功率。
1.2.3.2 ITS
利用NCBI网站上的BLAST程序对获得的不同序列片段进行比对分析,确认序列的可靠性。将测得的序列与GenBank中下载的其他轮枝菌相关序列用CLUSTAL X[17]程序进行分析,如果序列长度不同,则只保留共有区段。将比对好的序列输入DNAstar软件(Lasergene,WI,USA)计算相似性矩阵,然后用Taxon-Gap软件[18]分析各基因在种内与种间的变异情况。
1.2.3.3 种内与种间距离频率分布
应用Species identifier软件[19]比较种内及种间的遗传距离差异,并用EXCEL软件作图分析。使用Species identifier的Best Match功能进行条形码种类鉴定成功率的计算。
1.2.3.4 邻接树构建
使用 MEGA5.0程序[20]进行比对,以 NCBI上下载的大豆疫霉(Phytophthora sojae)的ITS序列(HQ643349)为外群,按照Saitou and Nei[21]的方法采用邻接法(neighbor-joining method,NJ)构建系统进化发育树,进行1000次重复抽样计算系统树中节点的置信度。
2 结果与分析
2.1 获得序列的难易程度
评价DNA条形码的一个重要指标是序列获得的难易程度,即PCR扩增与测序的成功率。本研究成功获得轮枝菌属13个种35个菌株的ITS片段,片段长度约为550bp左右,PCR扩增与测序成功率为97.1%。
2.2 种内与种间差异分析
本试验获得的轮枝菌属13个种35个ITS片段的序列,与GenBank下载的相关轮枝菌属ITS序列共73条序列一起进行分析,以探讨ITS片段区分不同轮枝菌种类的能力。轮枝菌属ITS片段的种内与种间序列差异分析如图1所示,图中黑色柱表示种间差异,白色柱表示种内差异。结果表明,供试轮枝菌属不同种类中V.chlamydosporium、V.fungicola、V.griseum、V.lecanii、V.lindauianum、V.nigrescens、V.nubilum、V.psalliotae、V.theobromae和V.tricorpus的种间差异均大于种内差异,占到了供 试 种 类 的 76.9% (10/13),但 V.albo-atrum、V.dahliae和V.longisporum的种内差异大于种间差异。

图1 采用TaxonGap软件分析轮枝菌属ITS片段种内与种间差异Fig.1 Comparisons of intra-and inter-specific variations of ITS fragment fromVerticilliumspp.
2.3 种内与种间距离的频率分布
应用 Taxon DNA/Species identifier软件Pairwise Summary功能对所获得的各物种ITS序列进行分析,ITS条形码片段的种内与种间距离的频率分布如图2所示。种内距离(intraspecific distance)主要集中在0~5.0%间,而种间距离(interspecific distance)则在5%~40%之间,两者存在交叉重叠,其中≤0的差异序列占42.6%。使用Species identifier的Best Match分析功能,能够进行准确鉴定的序列占到了供试序列的94.5%(69/73)。
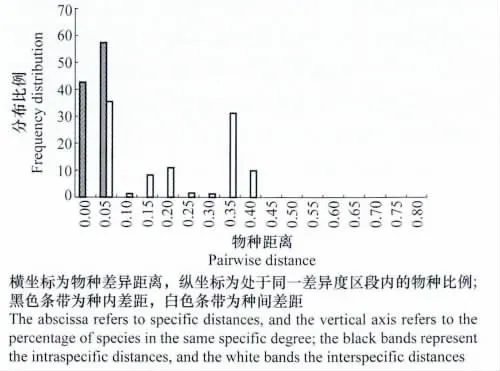
图2 轮枝菌属ITS序列种内与种间差距的频率分布Fig.2 Intraspecific and interspecific distances of ITS in Verticillium species
2.4 邻接树重建
轮枝菌属ITS基因构建的NJ树显示,同一个种的菌株聚在一起,不同的种则位于不同的末端分支,但是V.albo-atrum 和V.longisporum 不能很好分辨开(图3),这与ITS基因种内与种间差异分析结果相一致。植物病原性轮枝菌,包括黑白轮枝菌、大丽轮枝菌、三体轮枝菌、变黑轮枝菌、云状轮枝菌、V.the obromae和长孢轮枝菌聚在了一起,形成了一个大的分支(bootstrap值为99)。其中的黑白轮枝菌、大丽轮枝菌、长孢轮枝菌、三体轮枝菌和云状轮枝菌具有更加紧密的亲缘关系(bootstrap值为99)。非植物病原性轮枝菌聚为了另一个大的分支(bootstrap值为93)。
3 讨论
轮枝菌属真菌均可产生典型轮枝状分生孢子梗,且存在较大的形态变异,一些赖以区分的典型形态结构,如黑白轮枝菌的休眠菌丝,大丽轮枝菌的微菌核在培养中均有可能丧失,因此单纯依据形态特征进行轮枝菌种类区分具有较高难度。与传统分类学方法比较,DNA条形码的优势在于:(1)同一物种在其个体发育的各阶段均能作为分析对象,增加了待检样本的采集渠道;(2)待检对象不受其个体形态特征或组织特异性限制,样品形态发生改变或者残缺均不会影响识别结果;(3)识别速度快且准确性高,克服因形态相似所导致的鉴定误差,加速新物种的发现;(4)可操作性强,不需要专门的分类学知识也可以完成物种的鉴定,节省人力和物力[22]。为了实现简便、快捷而准确的物种鉴定,DNA条形码必须容易获得,这是衡量DNA条形码技术的最基本标准之一[23]。本研究表明,供试轮枝菌属ITS片段很容易直接扩增和测序,引物通用性强,扩增测序成功率高达97.1%,具有合适的片段长度(550bp左右),便于高通量测序与分析。同时,Genbank等DNA序列数据库中也已经积累了大量真菌ITS序列,便于参照和比较分析。理想条形码检测到的同属内种间遗传变异应明显大于种内遗传变异,并在两者之间存在显著差异,形成一个明显的间隔区,它是评价DNA条形码理想与否的另一个重要指标[24-25]。本研究中,供试13个种中有10个种的种间差异大于种内差异,存在交叉重叠,除无法成功区分亲缘关系很近的种外,其序列鉴定成功率达94.5%。
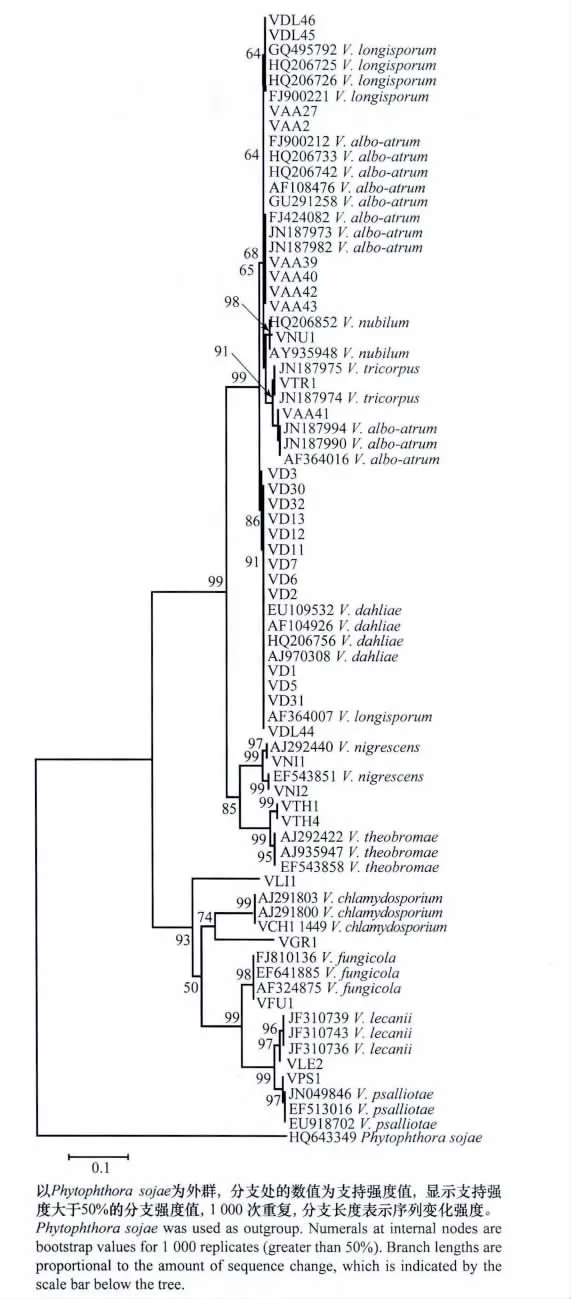
图3 根据轮枝菌ITS序列采用邻接法构建的系统进化树Fig.3 Phylogenetic tree of the neighbor-joining(NJ)analysis inferred from the ITS regions of Verticilliumspp.
近来研究表明,黑白轮枝菌和长孢轮枝菌目前在种类划分上仍然存在较大争议。在我们前期研究中发现,来自于不同寄主和地理来源的黑白轮枝菌能够被划分为3个不同分支[26]。最近,Inderbitzin等已将黑白轮枝菌重新划分为3个新种[27]。有研究表明长孢轮枝孢可能是轮枝菌种间杂交所致,可能的亲本来自于大丽轮枝菌、黑白轮枝菌或其他轮枝菌,其分生孢子中DNA含量接近大丽轮枝菌的1.75倍,由其所产生分生孢子长度可达7~9μm,微菌核较大丽轮枝菌更加细长[2,28]。Karapapa等认为应将欧洲油菜上分离的大丽轮枝菌长孢变种上升到新种高度,即V.longisporum[28]。然而,也有一些与此不一致的报道。如来自于英国球芽甘蓝(Brussels sprouts)的一株分离物具有较高的DNA含量,但是分生孢子却较短[28]。分离物V33具有较长分生孢子,但是DNA含量却比较低[29]。本研究中ITS片段对黑白轮枝菌和长孢轮枝菌划分存在一定问题可能与这些种类分类地位不稳定存在一定关系,因此在对生物物种进行DNA条形码研究时,应该选择经过可靠形态鉴定且不存在分类争议的物种为材料,然后测定其标记基因的序列,以实现对该物种的有效DNA条形编码。
[1] Barbara D J,Clewes E.Plant pathogenic Verticillium species:how many of them are there?[J].Molecular Plant Pathology,2003,4:297-305.
[2] Inderbitzin P,Davis R M,Bostock R M,et al.The ascomycete Verticillium longisporum is a hybrid and a plant pathogen with an expanded host range[J].PLoS ONE,2011,6(3):e18260.
[3] Kress W J,Wurdack K J,Zimmer E A,et al.Use of DNA barcodes to identify flowering plants[J].Proceedings of the National Academy of Sciences of the United States of America,2005,102(23):8369-8374.
[4] Hebert P D,Gregory T R.The promise of DNA barcoding for taxonomy[J].Systematic Biology,2005,54(5):852-9.
[5] Hebert P D,Penton E H,Burns J M,et al.Ten species in one:DNA barcoding reveals cryptic species in the neotropical skipper butterfly Astraptes fulgerator[J].Proceedings of the National Academy of Sciences of the United States of America,2004,101(41):14812-14817.
[6] Taberlet P,Coissac E,Pompanon F,et al.Power and limitations of the chloroplast trnL(UAA)intron for plant DNA barcoding[J].Nucleic Acids Research,2007,35(3):e14.
[7] Hajibabaei M,Singer G A,Hebert P D,et al.DNA barcoding:how it complements taxonomy,molecular phylogenetics and population genetics[J].Trends in Genetics,2007,23(4):167-172.
[8] Pfenninger M,Nowak C,Kley C,et al.Utility of DNA taxonomy and barcoding for the inference of larval community structure in morphologically cryptic Chironomus (Diptera)species[J].Molecular Ecology,2007,16(9):1957-68.
[9] Selvaraj D,Sarma R K,Sathishkumar R.Phylogenetic analysis of chloroplast matK gene from Zingiberaceae for plant DNA barcoding[J].Bioinformation,2008,3(1):24-7.
[10]Otomo P V,van Vuuren B J,Reinecke S A.Usefulness of DNA barcoding in ecotoxicological investigations:resolving taxonomic uncertainties using Eisenia malm1877as an example[J].Bulletin of Environmental Contamination and Toxicology,2009,82(3):261-264.
[11]Valentini A,Pompanon F,Taberlet P.DNA barcoding for ecologists[J].Trends in Ecology & Evolution,2009,24(2):110-117.
[12]Keith A S.Progress towards DNA barcoding of fungi[J].Molecular Ecology Resources,2009,(s1):83-89.
[13]Schubert K,Groenewald J,Braun U.Biodiversity in the Cladosporium herbarumcomplex (Davidiellaceae,Capnodiales),with standardisation of methods for Cladosporium taxonomy and diagnostics[J].Studies in Mycology,2007,58:105-156.
[14]Skouboe P,Frisvad J,Taylor J W,et al.Phylogenetic analysis of nucleotide sequences from the ITS region of terverticillate Penicilliumspecies[J].Mycological Research,1999,103:873-881.
[15]O’Donnell K,Cigelnik E.Two divergent intragenomic rDNA ITS2types within a monophyletic lineage of the fungus Fusariumare nonorthologous[J].Molecular Phylogenetics and Evolution,1997,7:103-116.
[16]White T J,Bruns T,Lee S,et al.Amplification and direct sequencing of fungal ribosomal RNA genes for phylogenetics[M]∥PCR Protocols:A Guide to Methods and Applications.Innis M A et al,Edi.1990,Academic Press:New York.
[17]Thompson J,Higgins D,Gibson T.CLUSTAL W:improving the sensitivity of progressive multiple sequence alignment through sequence weighting,position-specific gap penalties and weight matrix choice[J].Nucleic Acids Research,1994,22(22):4673-4680.
[18]Slabbinck B,Dawyndt P,Martens M,et al.TaxonGap:a visualization tool for intra-and inter-species variation among individual biomarkers[J].Bioinformatics,2008,24(6):866-867.
[19]Meier R,Kwong S,Vaidya G,et al.DNA barcoding and taxonomy in Diptera:a tale of high intraspecific variability and low identification success[J].Systematic Biology,2006,55:715-728.
[20]Tamura K,Peterson D,Peterson N,et al.MEGA5:Molecular evolutionary genetics analysis using maximum likelihood,evolutionary distance,and maximum parsimony methods[J].Molecular Biology and Evolution,2011,28:2731-2739.
[21]Saitou N,Nei M.The neighbor-joining method:a new method for reconstructing phylogenetic trees[J].Molecular Biology and Evolution 1987,4(4):406-425.
[22]张宇,郭良栋.真菌DNA条形码研究进展[J].菌物学报,2012,31(6):809-820.
[23]赵鹏,罗晶,庄文颖.COI基因作为丛赤壳科真菌DNA条形码的测试[J].菌物学报,2012,31(2):243-250.
[24]Meyer C P,Paulay G.DNA barcoding:error rates based on comprehensive sampling[J].PLoS Biology,2005,3(12):2229-2238.
[25]Lahaye R,van der Bank M,Bogarin D,et al.DNA barcoding the floras of biodiversity hotspots[J].Proceedings of the National Academy of Sciences of the United States of America,2008,105(8):2923-2928.
[26]段维军,郭立新,张祥林,等.检疫性轮枝菌及其近似种的鉴定[J].植物病理学报,2013,43(3):274-285.
[27]Inderbitzin P,Bostock R M,Davis R M,et al.Phylogenetics and taxonomy of the fungal vascular wilt pathogen verticillium,with the descriptions of five new species[J].PLoS ONE,2011,6(12):e28341.
[28]Karapapa V,Bainbridge B,Heale J.Morphological and molecular characterization of Verticillium longisporumcomb.nov.,pathogenic to oilseed rape[J].Mycological Research,1997,101(11):1281-1294.
[29]Steventon L A,Fahleson J,Hu Q,et al.Identification of the causal agent of Verticillium wilt of winter oilseed rape in Sweden,V.longisporum[J].Mycological Research,2002,106(5):570-578.

