斜纹夜蛾核型多角体病毒DNA解旋酶序列比较分析
刘艳荷, 方继朝
(1.江苏省农业科学院植物保护研究所,江苏 南京 210014;2.广东海洋大学农学院,广东 湛江 524088)
斜纹夜蛾核型多角体病毒(Spodoptera litura nucleopolyhedrovirus,SpltNPV)隶属于杆状病毒科(Baculoviridae),α 杆状病毒属 Alphabaculovirus[1]。SpltNPV在自然界存在多种分离株,具有丰富的基因型多态性。根据宿主范围、生长特性、多角体蛋白质(Polyhedrin,polh)和DNA限制性酶切图谱,Splt-NPV分为4组:AcNPV(Autographa californica NPV)型;SpliNPV(S.littoralis NPV)型:包括 SpliNPV-A和 SpliNPV-B;SpltNPV 特有基因型[2]。Kamiya 等[3]从日本采集17株病毒,空斑纯化183个克隆,根据基因组DNA限制性酶切图谱,分为A、B和C 3种基因型,A 型与 SliNPV-D[4]或 SliNPV-B[5]相似,B型与中国、日本、菲律宾的斜纹夜蛾幼虫中鉴定的SpltNPV 相似[6-8],而C 型SpltNPV 与A 型和 B 型都不同,是一种新型病毒。
DNA解旋酶(Helicase)是生物生命现象中的一种重要的酶,催化dsDNA向ssDNA的转换,并参与新生链的合成,在DNA修复和重组过程中都发挥着必不可少的作用。杆状病毒DNA解旋酶除了参与DNA复制外,在决定杆状病毒宿主域方面有着重要的作用[9]。AcNPV和家蚕核型多角体病毒(Bombyx mori NPV,BmNPV)的 DNA解旋酶基因(helicase)是决定它们寄主范围的重要基因[10-12]。克隆 AcNPV helicase,与 BmNPV 基因组DNA共转染家蚕细胞,筛选出既能感染所有Ac-NPV寄主,又可以在家蚕中表达的杂交病毒,其helicase基因首(1~780 bp)尾(2 795~3 669 bp)与BmNPV对应的DNA序列相同,但中间编码的22个氨基酸与AcNPV相同[13]。本研究克隆Splt-NPV不同基因型克隆的helicase,对其核苷酸序列和氨基酸序列进行比较分析,为探讨杆状病毒宿主域扩大机理提供理论依据。
1 材料与方法
1.1 供试材料
试验用病毒A6分离自SpltNPV埃及株,X8、X15和X17从来源于日本小笠原岛的感病斜纹夜蛾幼虫中分离获得,保存在江苏省农业科学院植物保护研究所作物虫害研究室(下文简称本实验室)。病毒SeNPV以及克隆用大肠杆菌Escherichia coli TG1为本实验室保存;Taq酶购自 TaKaRa公司,pGEM-T easy Vector、T4 DNA连接酶均购自Promega(美国)公司;其他试剂均为国产分析纯试剂。离心机型号为Eppendorf 5417R和5810R。基因核苷酸序列测定在Invitrogen(美国)公司进行。
1.2 斜纹夜蛾的饲养
斜纹夜蛾幼虫自南京郊区甘蓝地采集,带回实验室饲养繁殖。幼虫饲养温度(27±1)℃,相对湿度60%,光周期14∶10(L∶D)。幼虫以麦胚人工饲料在玻璃缸中群体饲养,4龄后以6孔板单头饲养;蛹放置于同样的玻璃缸中,即将羽化时,移入成虫笼。成虫饲喂5%蔗糖水,以普通白纸置于笼壁供其产卵。
1.3 病毒的繁殖
通过幼虫口服接种繁殖病毒。将人工饲料切成小块放入6孔板,滴加适量浓度病毒,每孔接入2~3头4龄初期斜纹夜蛾幼虫,待其吃完病毒液饲料后再补充新鲜人工饲料。接毒幼虫饲养条件为:饲养温度(27±1)℃,相对湿度60%,光周期14∶10(L∶D)。幼虫发病后收集染病虫体,4℃保存备用。
1.4 病毒基因组DNA的提取
将收集的病虫研磨,纱布过滤移除虫体碎屑。差速离心500 r/min 5 min,3 000 r/min 5 min,进行多角体纯化。将纯化的多角体(POBs)109个悬浮于300 μl ddH2O,加入 100 μl 3 × DAS 碱裂解液(0.3 mol/L Na2CO3,0.5 mol/L NaCl,0.03 mol/L EDTA,pH 10.5),37℃水浴10 min。待裂解液变清后,加入 200 μl TE(pH 8.0),8 000 r/min离心 8 min。移出上清,加入蛋白酶K至终浓度200 μg/ml,45~50℃水浴2.5 h,再加入10%SDS至终浓度1%,继续水浴30 min。苯酚抽提1次,苯酚∶氯仿∶异戊醇(体积比为25∶24∶1)抽提2次,再用无水乙醇沉淀基因组 DNA,干燥后溶解于 TE(pH 8.0)备用[14~16]。
1.5 PCR扩增及扩增产物的克隆
参照 GenBank登录的 SpltNPV(AF527603)、SpltNPV2(NC_011616)基因组全序列设计引物(表1),以方法1.4提取的病毒基因组DNA为模板,PCR扩增,将所得片段克隆入pGEM-T easy Vector,测序拼接。

表1 试验中所用的PCR引物Table 1 PCR primers used in the experiments
1.6 核苷酸及氨基酸序列的比较
将所得的病毒helicase核苷酸序列和氨基酸序列与SpltNPV不同基因型及其他核型多角体病毒的helicase序列,利用Blast、Clustal W等软件进行同源性比较和分子进化分析。
2 结果与分析
2.1 SpltNPV不同基因型helicase的克隆
SpltNPV 3种基因型,A型和B型同源性较高,而C基因型与A型和B型的同源性较低,因此需要以SpltNPV和SpltNPV2基因组全序列为模板分别设计引物。SpltNPV helicase长3 708 bp,编码1 235个氨基酸;SpltNPV2 helicase长3 654 bp,编码1 217个氨基酸,为方便PCR扩增,设计2对引物。利用这些引物,分别以A6、X8、X15和X17病毒基因组DNA为模板,PCR扩增出8条1 800 bp左右的片段,克隆到pGEM-T easy Vector中测序(图1)。进行序列拼接后,得到 A6、X8、X15和 X174个病毒克隆 helicase的完整编码区。

图1 斜纹夜蛾核型多角体病毒A6、X8、X15和 X17株helicase PCR扩增产物Fig.1 Electrophoresis of helicase PCR products from SpltNPV A6,X8,X15,and X17isolates
2.2 SpltNPV不同基因型helicase的序列比较分析
SpltNPV 病毒克隆 A6、X8、X15的 helicase编码区全长相同,为3 756 bp,比SpltNPV helicase长48 bp,分别在302~316 nt、333~360 nt和2 599~2 604 nt插入15 bp、27 bp 和 6 bp。SpltNPV helicase与 A6、X8、X15的helicase核苷酸序列相似性分别为89%、89%和91%,而A6和X8与X15的相似性都是97%,A6和 X8的相似性为99%。A6、X8和 X15的 helicase氨基酸序列全长1 251个氨基酸,比SpltNPV helicase长16个氨基酸,分别在95~104个氨基酸、117~121个氨基酸和837~838个氨基酸处插入9个、5个和2个氨基酸。A6、X8和 X15的 helicase与SpltNPV helicase氨基酸序列相似性均为93%,A6和X8与X15氨基酸序列相似性均为99%。
SpltNPV病毒克隆X17的helicase编码区全长3 699 bp,比 SpltNPV2 helicase 长 45 bp,分别在2 167~2 178 nt、2 345~2 374 nt和2 483~2 485 nt处插入12 bp、30 bp和3 bp,核苷酸序列的相似性为90%。X17helicase比SeNPV helicase长30 bp,在26~28 nt和2 491~2 493 nt处各缺失 3 bp,而在2 157 ~2 159 nt、2 172 ~ 2 186 nt、2 353 ~2 364 nt、2 389 ~2 391 nt、2 440 ~2 442 nt处分别插入 3 bp、15 bp、12 bp、3 bp 和 3 bp,核苷酸序列的相似性为82%。X17helicase全长1 232个氨基酸,比SpltNPV2-helicase长15个氨基酸,在720~723个氨基酸、784~793个氨基酸和813个氨基酸处分别插入4个氨基酸、10个氨基酸和1个氨基酸,氨基酸序列相似性为92%。X17-helicase比 SeNPV-helicase多编码10个氨基酸,在第8个氨基酸、812~813个氨基酸和827~830个氨基酸处分别缺失1个氨基酸、2个氨基酸和4个氨基酸,而在725~730个氨基酸、837~847个氨基酸处分别插入6个氨基酸、11个氨基酸,氨基酸序列相似性为83%。
A6、X8、X15的 helicase 和 X17的 helicase 同源性低,核苷酸序列相似性均为52%,氨基酸序列的相似性均为42%。
2.3 SpltNPV-helicase的进化分析
从GenBank下载33种NPV的helicase氨基酸序列(表2),利用 Clustal W 和 Treeview(V.1.6.6)软件,绘制SpltNPV不同基因型及克隆和下载的33种NPV的系统进化树(图2)。在进化树上SpltNPV不同基因型及克隆分别处于两大分枝上:X17、Splt-NPV2、SeNPV和SfMNP处在一个分支;因SpliNPV的helicase序列在GenBank上没有登录,SpltMNP与分离自日本小笠原株的X8和X15、分离自埃及株的A6及LsNPV处在一个分支上,这与多角体蛋白质基因核苷酸序列的进化分析结果基本一致[17]。本试验克隆的A6和X8同源性高,在一个小的分支上,然后和X15汇合一个较大的分支,再与SpltNPV汇合在一起,最后与LsNPV构成一个较大的分支。
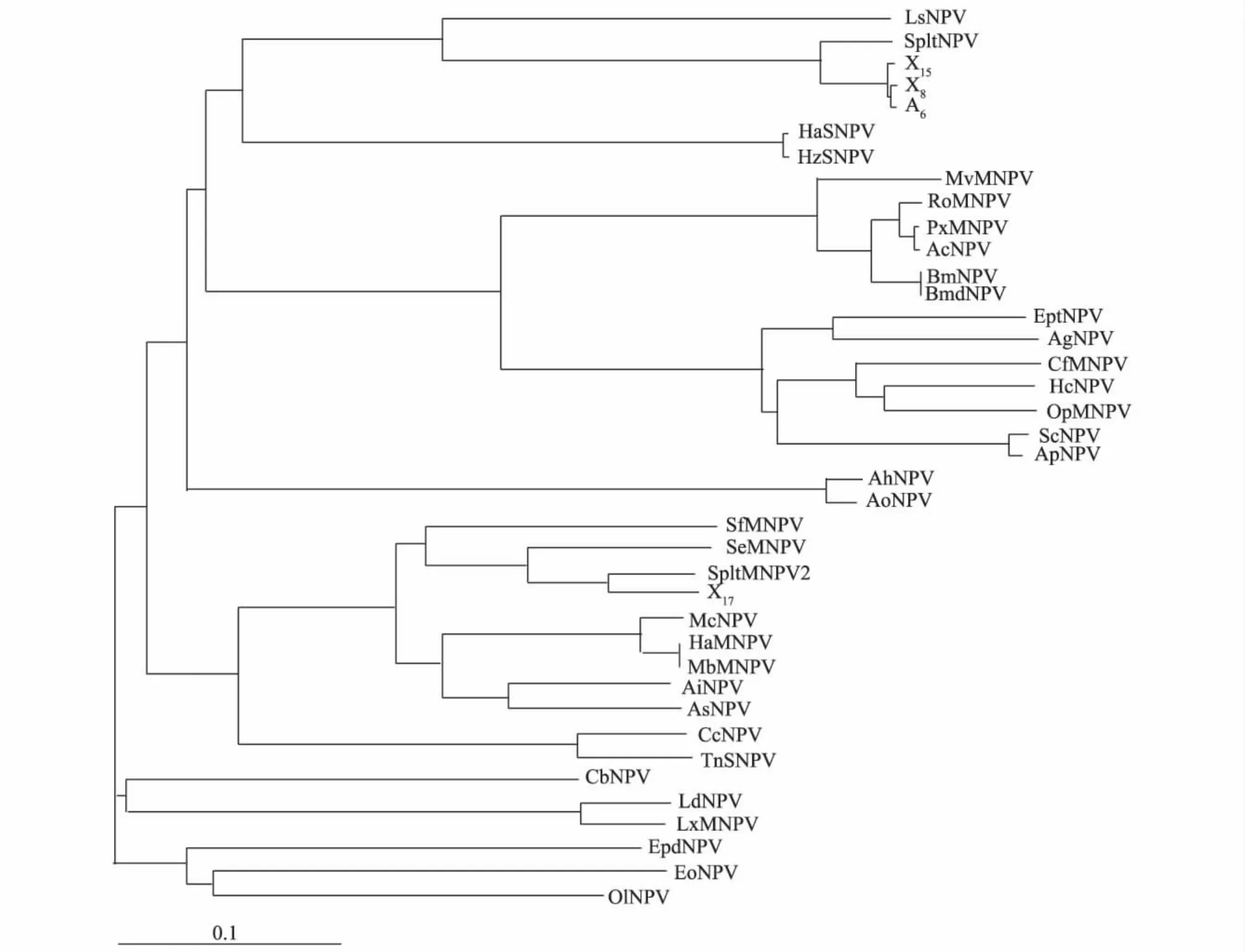
图2 SpltNPV 6种基因型及克隆和33种杆状病毒helicase氨基酸序列的NJ进化树Fig.2 The NJ phylogenetic tree of 6 SpltMNPV genotypes and 33 nucleopolyhedrovirus based on helicase amino acid sequences

表2 进化分析中所用SpltNPV不同基因型和其他33种NPV的helicase基因Table 2 Helicase genes of SpltMNPV genotypes and other 33 NPVs used in the phylogenetic analysis
3 讨论
本研究克隆了 4株(A6、X8、X15和 X17)病毒的helicase基因,从同源性分析结果看,A6、X8、X15与SpltNPV同源性较高,核苷酸序列的相似性分别为89%、91%和89%,氨基酸序列的相似性都是99%。因为SpliNPV的helicase基因在GenBank上没有登录,无法进行同源性比较,因此单就helicase序列,无法判断A6、X8、X15属于 SpliNPV型(A基因型)或SpltNPV型(B基因型)。X17与SpltNPV2、SeNPV同源性较高,核苷酸序列的相似性分别为90%和82%,氨基酸序列的相似性分别为92%和83%,而与SpltNPV同源性较低,核苷酸序列的相似性为52%,氨基酸序列的相似性为42%,因此X17属于SeNPV型(C基因型)。从进化分析结果看,A6、X8和X15在同一大的分支,而X17与SpltNPV2和SeNPV在同一大的分支,这与以多角体蛋白质基因核苷酸序列的进化分析结果一致。在克隆的4株病毒helicase基因中,A6、X8、X15之间序列差异比较小,可能由于克隆的病毒株数较少,没有包括与SpltNPV亲缘关系最近的病毒株,也可能在自然界中,SpltNPV和SpliNPV发生同源重组,使得该基因逐渐趋于相似,如这两种基因型的几丁质酶基因、核苷酸序列和氨基酸序列的相似性均为99%[18]。
SpltNPV2基因组全序列已被测定(NC_011616),全长148 634 bp,150 bp以上的 ORFs 149个,基因组比SpltNPV长9 kb,多9个ORFs,两者之间96个ORFs有同源性,氨基酸序列相似性平均为44.6%,基因顺序存在很大差异。SpltNPV2比SeNPV基因组长13 kb,但两个病毒间133个ORFs具有同源性,氨基酸序列相似性平均76.5%,而且基因顺序存在明显一致性。虽然SpltNPV2与SpltNPV来源于相同的宿主。但是它们之间的同源性远不如SpltNPV2和 SeNPV 高[19]。X17是从日本小笠原采集的病毒株分离获得,已对其部分基因进行了克隆,发现其基因与SpltNPV2和SeNPV同源性高[17]。本试验以X17和SeNPV饲喂甜菜夜蛾幼虫,发现该病毒对甜菜夜蛾的感染率为44%,但甜菜夜蛾幼虫致死时间与感染斜纹夜蛾致死时间相当,远远长于SeNPV感染甜菜夜蛾的致死时间。另外也有报道SpltNPV能够侵染甜菜夜蛾的细胞核幼虫[20]。目前已证实DNA helicase是决定AcNPV和BmNPV宿主范围的重要基因,但不同杆状病毒之间,其宿主域决定机制不同。至于在SpltNPV中,helicase基因是否与病毒宿主域或侵染时间有关,还需要进行基因删除等试验进一步研究确定。
[1]KING A M,LEFKOWITZ E,ADAMS M J,et al.Virus taxonomy:ninth report of the international committee on taxonomy of viruses[M].Elsevier:Academic Press,2011.
[2]MAEDA S,MUKOHARA Y,KONDO A.Characteristically distinct isolates of the nuclear polyhedrosis virus from Spodoptera litura[J].J Gen Virol,1990,71:2631-2639.
[3]KAMIYA K,ZHU J,MURATA M,et al.Cloning and compara-tive characterization of three distinct nucleopolyhedroviruses isolated from the cotton cutworm,Spodoptera litura(Lepidoptera:Noctuidae)in Japan[J].Biological Control,2004,31(1):38-48.
[4]KISLEV N,EDELMAN M.DNA restriction pattern differences from geographic isolates of Spodoptera littoralis nuclear polyhedrosis virus[J].Virology,1982,119:219-222.
[5]CHERRY C L,SUMMERS M D.Genotypic variation among wild isolates of two nuclear polyhedrosis viruses isolated from Spodoptera littoralis[J].J Invertebr Pathol,1985,46:289-295.
[6]LAVIÑA-CAOILI B A,KAMIYA K,KAWAMURA S,et al.Comparative in vitro analysis of geographic variants of nucleopolyhedrovirus of Spodoptera litura isolated from China and the Philippines[J].J Insect Biotechnol Sericol,2001,70:199-209.
[7]PANG Y,YU J,WANG L,et al.Sequence analysis of the Spodoptera litura multicapsid nucleopolyhedrovirus genome [J].Virology,2001,287(2):391-404.
[3]TAKATSUKA J,OKUNO S,NAKAI M,et al.Genetic and biological comparisons of ten geographic isolates of a nucleopolyhedrovirus that infects Spodoptera litura(Lepidoptera:Noctuidae)[J].Biological Contro1,2003,26:32-39.
[9]肖庆利,张志芳,何家禄.杆状病毒DNA解旋酶[J].生命的化学,2001,21(6):456-458.
[10]MAEDA S,KAMITA S G,KONDO A.Host range expansion of Autographa californica nuclear polyhedrosis virus(NPV)following recombination of a 0.6-kilobase-pair DNA fragment originating from Bombyx mori NPV[J].J Virol,1993,67(10):6234-6238.
[11]JIN B R,YOON H J,CHOUDARY P V,et al.Characterization of recombinant Autographa californica nuclear polyhedrosis virus(NPV)expressing the beta-galactosidase gene in both Sf21 and Bm5 cells by Bombyx mori NPV p143 helicase gene[J].Mol Cells,1997,7(6):762-768.
[12]KAMITA S G,MAEDA S.Sequencing of the putative DNA helicase-encoding of the Bombyx mori nuclear polyhedrosis virus and fine-mapping of a region involved in host range expansion[J].Gene,1997,190(1):173-179.
[13]吴小锋,曹翠萍,许雅香,等.BmNPV和AcNPV扩大寄主域杂交重组病毒表达载体的构建和改进[J].中国科学(C辑,生命科学),2004,34(2):156-164.
[14]郭慧芳.昆虫核型多角体病毒的增效途径和机理[D].南京:南京农业大学,2005.
[15]GUO H F,FANG J C,WANG J P,et al.Interaction of Xestia cnigrum granulovirus with peritrophic matrix and Spodoptera litura nucleopolyhedrovirus in Spodoptera litura[J].Journal of Economic Entomology,2007,100(1):20-25.
[16]GUO H F,FANG J C,LIU B S,et al.Enhancement of the biological activity of nucleopoly-hedrovirus through disruption of the peritrophic matrix of insect larvae by chlorfluazuron [J].Pest Management Science,2007,63(1):68-74.
[17]刘艳荷,郭慧芳,方继朝.C型斜纹夜蛾核型多角体病毒X17病毒株部分基因的序列分析[J].昆虫学报,2011,54(9):1010-1017.
[18]刘艳荷,方继朝.斜纹夜蛾核型多角体病毒不同分离株基因序列的同源性分析[J]. 昆虫学报,2008,51(8):817-823.
[19]李轶女.东方粘虫颗粒体病毒和斜纹夜蛾核型多角体病毒Ⅱ基因组全序列分析[D].北京:中国农业科学院,2008.
[20]TAKATSUKA J,OKUNO S,ISHII T,et al.Host range of two multiple nucleopolyhedroviruses isolated from Spodoptera litura[J].Biological Control,2007,41:264-271.

