酰胺酶高通量筛选方法研究进展
田慧,郑仁朝,郑裕国
(浙江工业大学生物与环境工程学院生物工程研究所,浙江杭州310014)
酰胺酶(Amidase,EC 3.5.1.4)是一类催化酰胺C-N键水解生成相应羧酸和氨的重要生物催化剂。该酶促反应的本质是催化酰基从其供体(底物酰胺)向受体(如共底物水)的转移[1]。因此,当体系中存在比水更强的亲核试剂如羟胺或肼为受体时,可生成氧肟酸或酰基肼[2-3](图1),酰胺酶底物谱极广,能高效催化各种非天然脂肪族、杂环族及芳香族酰胺的水解[4-5],并具有高度化学选择性、区域选择性、立体选择性和不需辅酶等优点。大量研究表明,在其与腈水合酶(Nitrile hydratase,EC 4.2.1.84)耦联的腈立体选择性生物转化中,手性识别行为决定于酰胺水解步骤[6]。酰胺酶对映选择性生物催化在复杂结构手性羧酸、氧肟酸及酰胺衍生物的合成中显示出巨大潜力,成为手性生物催化的重要工具酶。对于特定的生物催化过程,合适、高效的生物催化剂,尤其是立体选择性生物催化剂的发现是最为关键的一步。这是因为一方面用传统的基于手性GC或HPLC的筛选方法,去逐一测定“海量”样品的活性及立体选择性十分费时费力。另一方面,随着蛋白分子改造技术的快速发展,突变文库包含的突变体数量巨大(通常含104~106个突变子),对快速、准确的高通量(High-throughput screening,HTS)酰胺酶筛选模型的建立提出了更为迫切的要求。一个成功的高通量筛选方法需满足以下条件:准确、快速和廉价[7]。本文综述了近年来发展起来的酰胺酶高通量筛选技术,为酰胺酶这一重要生物催化剂的发现提供借鉴。
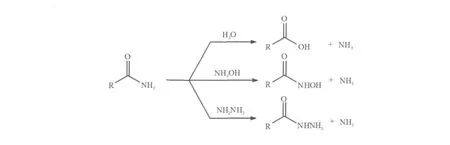
图1 酰胺酶催化的酰基转移反应(分别以水、羟胺和肼为酰基受体)Fig.1 Acyl transfer reactions catalyzed by amidase(water,hydroxylamine and hydrazine as acyl acceptor)
1 酰胺酶高通量筛选模型建立的基本思路
高通量筛选模型一般都是根据酶活测定方法建立起来的,而酶活测定方法又建立在酶反应产物的相关特性基础上。对于酰胺酶,可以根据其催化酰胺水解生成羧酸和氨[8]或催化酰基转移反应生成氧肟酸和氨[9]建立相应的酶活检测方法(图2)。如可用HPLC、GC等方法直接测定酰胺的消耗或羧酸的生成来测定酶活及光学选择性,或根据反应前后电导率的不同检测酶活。由于反应产物中含NH3,可用比色法和荧光法检测酶活。此外,还可根据氧肟酸在酸性条件下特异性地与Fe3+形成棕褐色复合物的特性进行定量检测。

图2 检测产物的生成或底物的减少来测定酰胺酶的活性Fig.2 Determination of products formed and substrates reduced in a amidase-catalyzed reaction
2 高通量酰胺酶筛选模型
2.1 基于NH3的显色、荧光反应
Pan等[10]建立了一种简单、快速、微量的奈斯勒试剂显色法,可直接从平板上筛选出产L-门冬酰胺酶(L-asparaginase,L-ASP)的菌株。其原理是利用该酶催化产生的NH3与奈斯勒试剂作用后生成棕红色碘化双汞铵络合物,可借助比色法进行定量测定。彭中建等[11]利用此方法成功选育高产L-门冬酰胺酶和性状稳定的大肠埃希菌突变株。氨与邻苯二醛及亚硫酸盐可生成荧光复合物(图3)。虽然目前还不清楚该化合物的精确结构,但该方法已经成功地用于酰胺酶活性的检测[12]。Genfa等[13]借助微量滴定板荧光读数器筛选能水解α-H-α-氨基酰胺的酰胺酶,从2 880个样品中快速筛选到8个活性较好的酰胺酶。

图3 氨的邻苯二醛/亚硫酸盐衍生反应Fig.3 Derivatization reaction of ammonia with o-phthalaldehyde and sulfite
谷氨酸脱氢酶(glutamate dehydrogenase,EC 1.4.1.3,GDH)可催化NH3与α-酮戊二酸生成L-谷氨酸,同时NAD(P)H氧化为NAD(P)+(图4)。NAD(P)H的氧化可在340 nm下定量检测。Sonke等[14]利用此方法筛选氨基酸酰胺消旋酶,通过待检测样品与光学选择性酰胺酶联合使用,从11 272个大肠埃希菌克隆中筛选到32个阳性克隆。

图4 谷氨酸脱氢酶GDH氨检测法原理Fig.4 The principle of Glutamate dehydrogenase(GDH)to detect ammonia
由于微生物培养基中通常含有酵母提取物和蛋白胨,微生物生长时会利用肽和氨基酸生成氨,导致使用上述高通量筛选方法时产生严重的背景干扰[12]。因此,实际使用时要通过离心收获细胞,缓冲液洗涤等步骤尽量减少干扰。
2.2 荧光法
Ramarao等[15]建立了荧光法脂肪酸酰胺酶(Fatty acid amide hydrolase,FAAH)高通量筛选技术。其原理是将荧光素连接在底物上,此时荧光被淬灭。在脂肪酸酰胺酶作用下,生成花生四烯酸和荧光素,荧光素重新释放产生荧光(图5)。

图5 荧光快速检测脂肪酸酰胺酶FAAH活性的原理Fig.5 The principle of fluorescence method for rapid detection the FAAH activity
Henke等[16]利用酰胺酶催化水解产生的胺类物质经4-氯-7-硝基苯并-2-氧氯-1,3-二唑(NBD-Cl)衍生化生成荧光染料容易检出的原理,建立了高通量筛选模型(图6)。该方法的不足是不能用于筛选氨基酰胺酶,因为底物氨基酰胺本身也是游离胺,也会被衍生化。
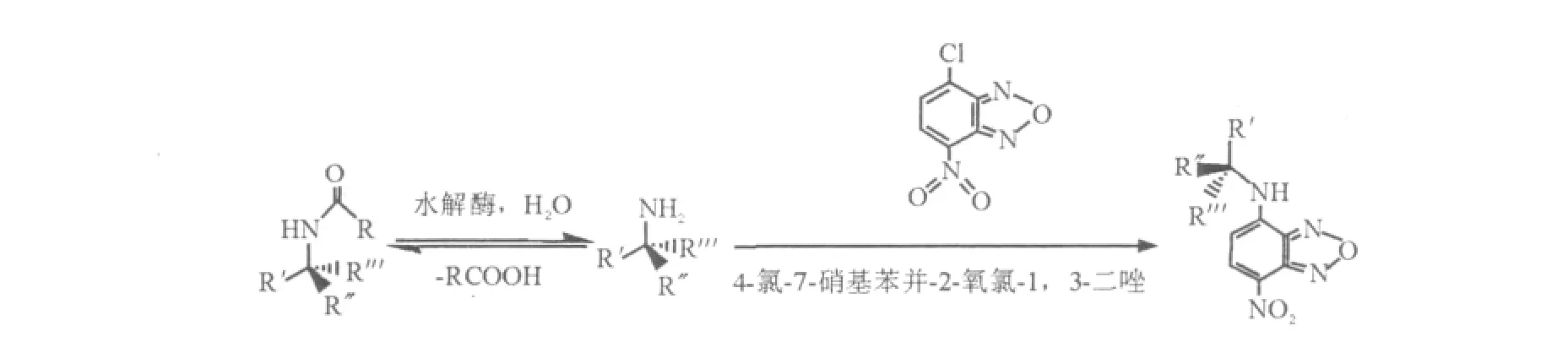
图6 胺类物质衍生化生成荧光染料反应式Fig.6 Reaction of derivatization amine to fluorescent dyes
酰胺酶可以催化氨基酰胺生成相应的氨基酸,氨基酸是很强的金属离子(特别是Cu2+)螯合剂,而氨基酰胺则无络合能力。伯尔尼大学的Dean等[17]利用铜的大环螯合物作为荧光指示剂,建立了氨基酸酰胺酶筛选模型(图7)。该方法的原理是Cu2+易与商品化的荧光素形成复合物,此时荧光素的荧光基团被Cu2+淬灭。而酰胺酶催化产物氨基酸能够更强烈地螯合Cu2+,重新释放荧光素,使其重获荧光。
2.3 基于氧肟酸与Fe(Ⅲ)的螯合特性
Zheng等[9]基于酰胺酶的酰基转移反应产物氧肟酸与铁离子在酸性条件下螯合显色特性,建立了高通量立体选择性酰胺酶筛选模型(图8)。从523株菌种中筛选得到2株能够R-选择性水解2,2-二甲基环丙甲酰胺的酰胺酶产生菌。该方法与现有的基于底物官能团特性的酰胺酶筛选模型相比,其更显著的优势在于对底物的普适性。
2.4 基于产物氨基酸与铜离子的螯合作用
Duchateau等[18]用Cu2+作为金属指示剂进行氨基酸酰胺酶活性的比色法检测,成功地用于检测转化α-甲基缬氨酸酰胺为α-甲基缬氨酸的酰胺酶活性(图9)。此方法的依据是碱性环境下α-氨基酸及其相应的氨基酰胺与Cu(Ⅱ)形成的复合物在光谱特性上的差异,当pH>8时,底物和产物即与Cu2+形成了不同颜色的络合物,Cu2+只作为金属指示剂。这有别于早期使用Cu2+或Ni2+作为金属指示剂并与大环的荧光金属螯合剂联用的方法。此方法的缺点是只适用于氨基酸酰胺酶的筛选,同时在碱性条件下影响酶活检测的准确性。

图7 铜配合物检测氨基酸的荧光法原理Fig.7 Principle of fluorescence assay for amino acid using copper-ligand complexes
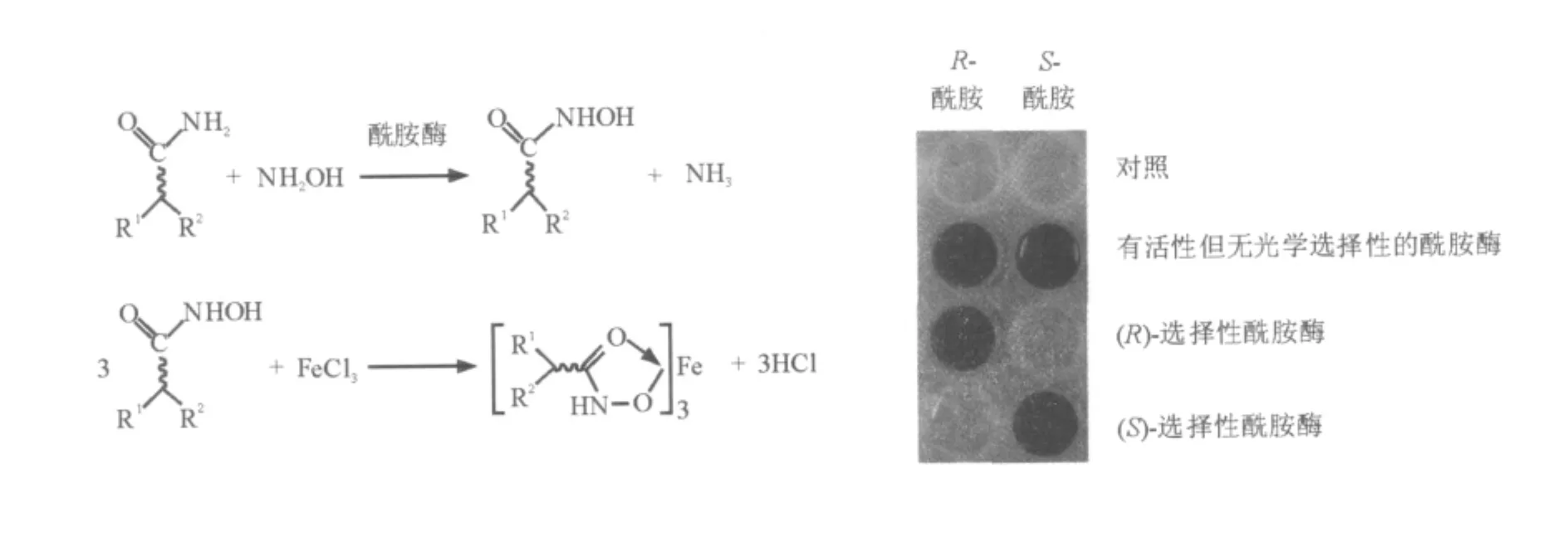
图8 立体选择性酰胺酶筛选模型构建机理与筛选实例Fig.8 The mechanism and examples of the colorimetric screening method for enantioselective amidase

图9 基于Cu2+与氨基酸络合反应的氨基酸酰胺酶筛选模型Fig.9 The model for screening amino acid amidase based on the Cu2+-complexed amino acid
2.5 基于琼脂平板显色法
利用过氧化物酶能够与D-氨基酸氧化酶作用产物过氧化氢发生反应并将其转化为有色产物的原理,建立了一种基于琼脂平板显色法的酰胺酶筛选模型。Komeda等[19]定向进化Ochrobactrum anthropi SV3 D-氨基酸酰胺酶,通过该法对突变文库进行筛选。将硝酸纤维膜上的菌落浸泡在含有底物D-苯丙氨酸酰胺、苯酚、D-氨基酸氧化酶、过氧化物酶和4-氨基安替比林的溶液中,挑出产生粉红/红色的菌落,最终筛到2个阳性突变株,其相对野生型酰胺酶的Vmax约提高了3倍,而表观Km值几乎不变。
2.6 核磁共振(Nuclear Magnetic Resonance,NMR)法筛选酰胺酶
NMR波谱是一种完善的分析技术,能够直接检测分子结构并进行定量。由于酶促反应中的底物和产物通常是具有不同磁性的化学个体,产生不同的NMR波谱。同时由于NMR波谱的定量性质,可通过测定底物和产物的量来监测反应进程。随着技术不断进步,测量速度大大加快,如今H-NMR已经能够适用于各种酶的中/高通量筛选。
Sonke等[20]用该方法筛选能水解D,L-α-甲基亮氨酸酰胺的酰胺酶,共筛选了120个微量滴定板(共11 520个样品),最终获得2个阳性克隆。另外,通过易错PCR、DNA shuffling等定向进化技术,Bovenberg等[21]使O.anthropi NCIMB40321 L-酰胺酶对DL-α-甲基苯基甘氨酰胺的比活提高了约5倍。
3 讨论
本文较为详细地介绍了近年来发展起来的高通量酰胺酶筛选方法及应用。这些方法的建立大大加快了酰胺酶的筛选速度,为从自然环境和突变文库中获得符合要求的酰胺酶提供了有效手段。总的来说,基于显色或荧光的高通量体系应用最为广泛。但是,筛选过程的重要原则之一是“获得目的菌株”,这些方法又通常经过发色团等基团的衍生化,筛选结果并不能完全代表酶对真实底物的活性和立体选择性。因此,在高通量筛选方法的构建过程中,保证底物的真实性是必须注意的问题。
[1] Maestracci M,Thiery A,Arnaud A,et al.A study of the mechanism of the reactions catalyzed by the amidase Brevibacterium sp.R312[J].Agric.Biol.Chem.,1986,50(9):2237-2241.
[2] Fournand D,Bigey F,Ratomahenina R,et al.Biocatalyst improvent for the production of short-chain hydroxamic acid[J].Enzyme Microbiol Technol,1997,20(6):424-431.
[3] Hirrlinger B,Stolz A.Formation of a chiral hyroxamic acid with an amidase from Rhodococcus erythropolis MP50 and subsequent chemical lossen rearrangement to a chiral amine[J].Appl.Environ.Microbiol.,1997,63(9):3390-3393.
[4] Sharma M,Sharma NN,Bhalla TC.Amidases:versatile enzymes in nature[J].Rev Environ Sci Biotechno,2009,8:343-366.
[5] Martínková L,Kïen V.Nitrile-and amide-converting microbial enzymes:stereo-,regio-and chemoselectivity[J].Biocatal Biotransform,2002,20(2):73-93.
[6] Mayaux J,Cerbelaud E,Soubrier F,et al.Purification,cloning,and primary structure of an enantiomer selective amidase from Brevibacterium sp.R312:structural evidence for genetic coupling with nitrile hydrates[J].J.Bacteriol,1990,172(12):6764-6773.
[7] 刘艳莉,杨广宇,王秋岩,等.脂肪酶和酯酶的定向进化及其应用[J].生物加工过程,2006,4(1):16-25.
[8] Martínková L,Vejvoda V,Kïen V.Selection and screening for enzymes of nitrile metabolism[J].Journal of Biotechnology,2008,133(3):318-326.
[9] Zheng RC,Zheng YG,Shen YC,A screening system for active and enantioselective amidase based on its acyl transfer activity[J].Applied Microbiology and Biotechnology,2007,74(11):256-262.
[10] Pan Pei-gen,Xu Jin,Liu Jin-Lin,et al.Fast screening of bacteria producing L-asparaginase[J].West China Journal of Pharmaceutical Sciences,1998,13(2):108-109.
[11] 彭中健,梁淑娃,谭颖嫦,等.L门冬酰胺酶高产菌株的选育[C].2007年全国生化与生物技术药物学术年会论文:344-347.
[12] Reymond J L(著),林章凛,蔡真(译).酶活检测—高通量筛选,遗传选择以及指纹分析(第1版)[M].北京:化学工业出版社,2008:94-94.
[13] Genfa Z,Dasgupta P K.Fluorometric measurement of aqueous ammonium ion in a flow injection system[J].Anal.Chem,1989,61(5):408-412.
[14] Sonke T,Boesten WH,Euverink GJ,et al.Polypeptides having a-H-a-amino acid amide racemase activity and nucleic acids encoding the same.WO 03/106691 to DSM IP Assets B.V.,2003.
[15] Ramarao MK,Murphy EA,Shen MW,et al.A fluorescencebased assay for fatty acid amide hydrolase compatible with highthroughput screening[J].Analytical Biochemistry,2005,343(1):143-151.
[16] Henke E,Uwe,Bornscheuer U.Fluorophoric Assay for the High-Throughput Determination of Amidase Activity[J].Anal.Chem,2003,75(2):255-260.
[17] Dean KE,Klein G,Renaudet O,et al.A green fluorescent chemosensor for amino acids provides a versatile high-throughput screening(HTS)assay for proteases[J].Bioorganic&Medicinal Chemistry Letters,2003,13(10):1653-1656.
[18] Duchateau AL,Hillemans-Crombach MG,Duijnhoven AV,et al.A colorimetric method for determination of amino amidase activity[J].Analytical Biochemistry,2004,330(2):362-364.
[19] Komeda H,Ishikawa N,Asano Y.Enhancement of the thermostability and catalytic activity of D-stereospecific amino-acid amidase from Ochrobactrum anthropi SV3 by directed evolution[J].Journal of Molecular Catalysis B:Enzymatic,2003,21(4-6):283-290.
[20] Sonke T,Kaptein B,Boesten WH,et al.Stereoselective Biocatalysis[M].New York,2000,55:23-58.
[21] Bovenberg RA,Kerkman R.Process for preparing variant polynucleotides.WO 03/010183 to DSM N.V.2003.

