基于g23基因的武汉东湖T4类浮游病毒遗传多样性
黄慧珍,程 凯,许 敏,赵以军(华中师范大学城市水环境生态学湖北省重点实验室,湖北 武汉 430079)
基于g23基因的武汉东湖T4类浮游病毒遗传多样性
黄慧珍,程 凯,许 敏,赵以军*(华中师范大学城市水环境生态学湖北省重点实验室,湖北 武汉 430079)
选取武汉东湖的2个子湖——郭郑湖和庙湖,分别在冬季和夏季采样,对T4类浮游病毒的g23基因进行PCR扩增,经连接转化后,随机挑选克隆子进行测序.结果表明,共得到46条有效序列,根据系统发育分析发现其明显分为6个组,显示出较高的多样性且具有明显的时空差异,说明富营养化水平的差异和季节变化将对浮游病毒的种群结构产生影响.仅部分g23序列与海洋T4类浮游病毒同源性较高,另一部分序列则可能代表了淡水富营养化水体中特有的浮游病毒类群.人为干扰会明显影响某些淡水水体中T4类浮游病毒的遗传多样性.
g23基因;东湖;T4;浮游病毒;遗传多样性
浮游病毒泛指悬浮于水体中的各种病毒,包括噬菌体、噬藻体和真核藻类病毒等[1].浮游病毒个体虽然微小,却是水体微生物群落中丰度最高的有机成分,其在水体生态系统中占据重要地位[2].国际上对浮游病毒多样性的研究是在20世纪80年代以后兴起,并且大部分研究都是针对海洋水体[3-4],以及稻田土壤和灌溉水[5].我国相关的研究刚刚起步,对淡水湖泊中的浮游病毒的多样性研究还集中在电镜观察和脉冲场电泳等方法
[6-8].刘艳鸣等[8]对东湖浮游病毒研究发现,其病毒基因组大小以 20~60kb的核酸带为强,这符合噬菌体和噬藻体的基因组主要范围.证实了东湖浮游病毒的优势类群为噬菌体和噬藻体.
g23基因编码 T4类噬菌体的主要衣壳蛋白(MCP)gp23(该蛋白由 521个氨基酸构成),在应用全球海洋采样(GOS)研究海洋浮游病毒宏基因组学的过程中,被发现是最具有代表性的蛋白质家族[3].Jia等[9]也建立了灌溉期间日本稻田表层土样的g23克隆子文库,由于土壤与海洋环境条件的明显不同,其T4类浮游病毒的种类也大不相同.
鉴于目前国内外对淡水湖泊浮游病毒研究尚少,国内对浮游病毒的遗传多样性研究仍为空白,本实验以T4类浮游病毒的g23基因为对象来研究淡水富营养化水体中T4类浮游病毒的遗传多样性,不但有助于填补这方面的研究空白,而且将有助于推测淡水浮游病毒的生态功能及其对淡水水体富营养化的响应机制.
1 材料与方法
1.1 水样采集与处理
选取武汉东湖的 2个子湖——郭郑湖和庙湖,分别代表轻度富营养化和重度富营养化湖泊.在2009年3月8日和8月5日上午,用有机玻璃采水器采取郭郑湖和庙湖表层水样,装入棕色玻璃瓶,用 1%氯仿或戊二醛固定,4℃保存,一周内进行浓缩处理.
冬季采集的水样须先滤过孔径为0.22μm的混合纤维膜,再用 TFF膜包(PALL公司,100kD),进行反冲超滤浓缩[17],1L水样最终浓缩为 20~30mL,分装入1.5mL塑料离心管,-20℃保存待用.夏季采集的水样在滤过0.22μm的混合纤维膜前,先用普通定性滤纸过滤,其余步骤同冬季水样.
1.2 方法
1.2.1 浮游病毒总 DNA提取 取 600μL病毒浓缩液,加入粗制的DNase和RNase,使终浓度均为 1μg/mL,室温放置 30~60min,8000r/min 离心5min,上清转入新的塑料离心管,加入蛋白酶k和SDS,使终浓度分别为50μg/mL和0.5%,56℃裂解1~3h,8000r/min离心 5min,上清转入新的塑料离心管,加入等体积饱和平衡酚,颠倒混匀,8000r/min离心 5min,上清转入新的塑料离心管;加入等体积酚:氯仿:异戊醇(25:24:1),颠倒混匀,8000r/min离心 5min,上清转入新的塑料离心管;加入等体积氯仿:异戊醇(24:1),颠倒混匀,8000r/min离心 5min,上清转入新的塑料离心管;加入 50μL3M 醋酸钠和 1000μL 冰无水乙醇,-20℃沉淀过夜;13200r/min离心 15min,用预冷的 70%乙醇洗涤两次,室温干燥.用 20~30μL TE溶解30min,1%琼脂糖电泳检查.提取的DNA放于-20℃保存待用.
1.2.2 PCR扩增 50μL扩增体系中加入 10×Buffer 5μL,15mmol Mg2+3μL,10mmol dNTP 1μL,5U Taq 酶 0.25μL,上下游引物 10mmol各1μL,模板 DNA6μL.使用的程序是 94℃预变性5min,30个循环的 94℃变性 45s,50℃退火 60s,72℃延伸45s,72℃终延伸10min.2%琼脂糖凝胶电泳检测产物.所用的引物为 MZIA1bis:5′-G ATATTTGIGGIGTTCAGCCIATGA-3′,MZIA6:5′-CGCGGTTGATTTCCAGCATGATTTC-3′[4](inv itrogen公司合成),能扩增出T4类浮游病毒的g23基因中450bp的片段.
1.2.3 克隆测序 使用 PMD18-T载体试剂盒(takara公司),随机挑选10~20个阳性克隆,交由金斯瑞科技南京有限公司测序.
1.2.4 分析 使用 DNAstar5.0软件包进行序列分析,用 mega4.0进行组内和组间发散度分析、CLUSTALW排序以及进化树的建立(NJ法).
2 结果与分析
郭郑湖和庙湖冬夏两个季节共 4个水样,经随机克隆共得到 46条序列,具体如下(Genebank序列号为HM005246-HM005291):
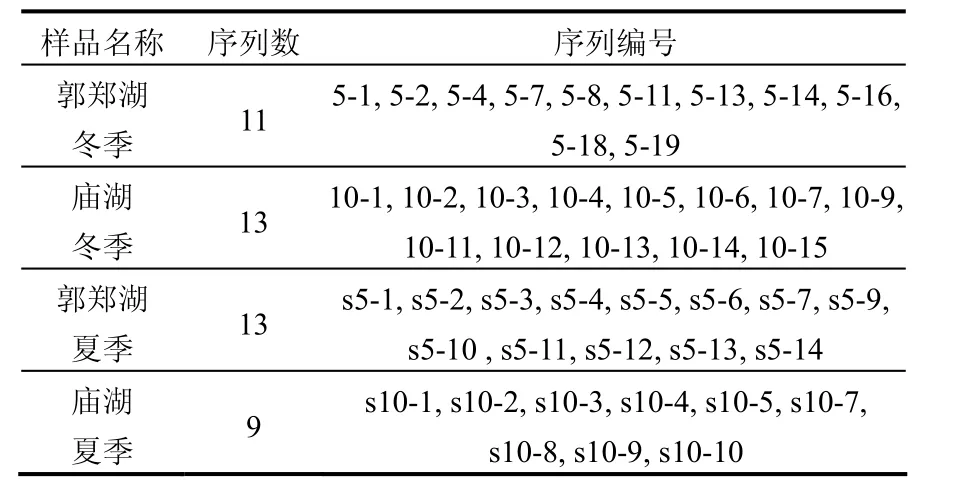
表1 序列来源Table 1 Sources of sequences
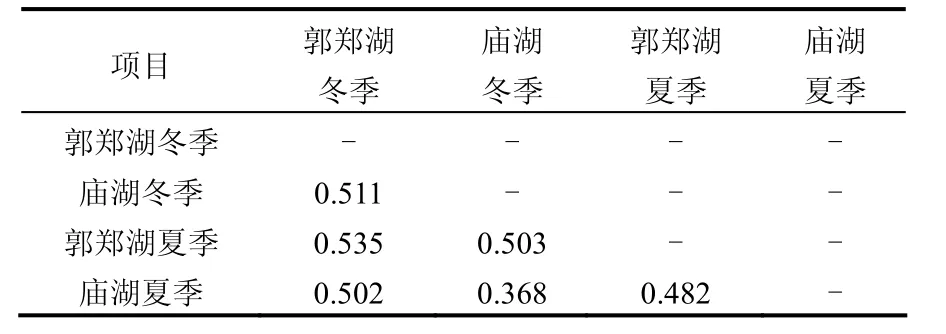
表2 组间发散度Table 2 Estimates of evolutionary divergence over sequence pairs between groups
根据组内平均发散度计算结果得到:冬季郭郑湖为0.365,庙湖为 0.411;夏季郭郑湖为 0.517,庙湖为 0.342.郭郑湖夏季水体的病毒之间的距离最大,即多样性最大,庙湖夏季水体的病毒之间距离最小,多样性最小.
每个水样所得病毒序列作为一个整体,两两相比较发散度的计算见表 2,其结果显示庙湖冬季与夏季病毒之间的差异最小,而郭郑湖两季之间的差异最大.
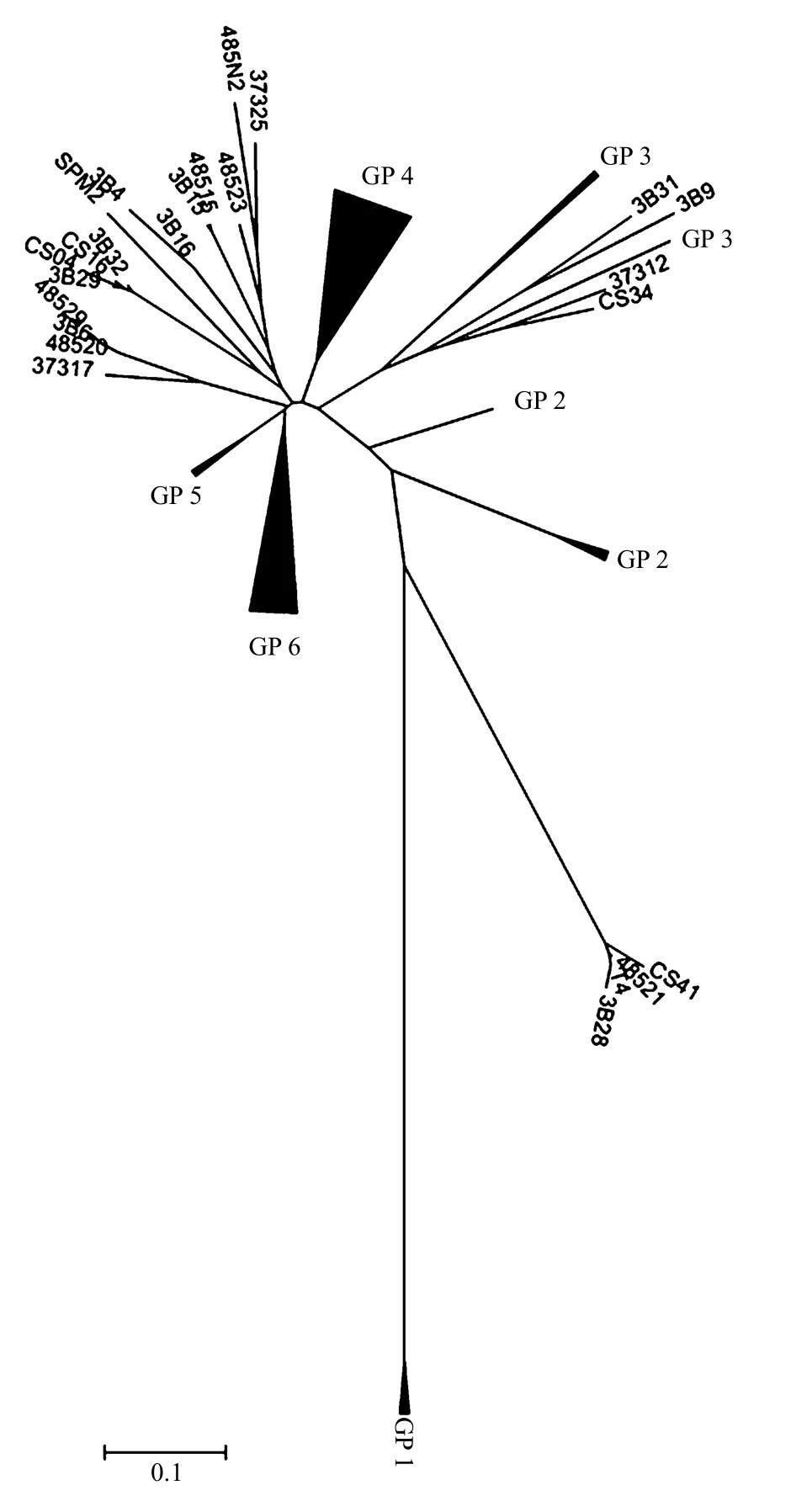
图1 本研究所得序列与T4和典型海洋噬菌体[4]的系统发育树Fig.1 Phylogenetic tree of the sequences we obtained, T4 and the typical marine bacteriophages[4]
3 讨论
3.1 基于g23基因序列的东湖T4类浮游病毒进化关系分析
根据本研究所得的 g23序列与部分已报道的海洋噬菌体T4类浮游病毒的g23序列所构建系统发育树(图1),可将本研究所得序列分为6组(GP1~6)(具体分组情况如图 2所示).由图 2可见:GP1、GP2、GP5和 GP6的进化方向明显不同于海洋T4类浮游病毒,而GP 3(包括3条序列)和GP 4(包括24条序列)则明显与海洋起源的噬菌体的亲缘关系较近,很可能与海洋噬菌体有共同的起源,说明淡水噬菌体与海洋噬菌体可能来源于共同的祖先[10].GP 4和GP 6分别包括有24和10条序列,共占所有序列的73.9%,均涵盖本次研究中全部4个来源的样品,是东湖中T4类浮游病毒的主要类群,其中,由于GP6与海洋噬菌体的亲缘关系更远,具有更加独立的进化途径,很可能代表淡水富营养化水体中T4类浮游病毒的典型类群;GP 1包括 3条序列(5-8,10-2,10-14),均来源于两个湖泊的冬季水样,很可能是冬季典型序列,并且该枝与其他所有序列的亲缘关系都非常远;GP 2分为2个进化方向,包括4条序列(s5-9,s5-11,s10-9,s10-10),均来源于两个湖泊的夏季水样,很可能都是夏季典型序列;GP 5仅包括两个序列(s10-7,s10-8),均来自庙湖夏季样品.
有报道表明海洋噬菌体群落的多样性非常高[11-12],Ursula Dorigo等[10]对淡水噬菌体g20基因的研究结果也显示出淡水噬菌体的多样性十分丰富,本实验从来自2个湖区的46条序列中即得到了6个明显的类群,显示东湖中T4类噬菌体也具有很高的遗传多样性.这可能是由于病毒和宿主间或感染相同宿主的病毒间的基因交换所导致[12-13].另一个假说认为,噬藻体群落的多样性可能与宿主群落多样性有关[14-16].
3.2 T4类浮游病毒遗传多样性的时间变化
3.2.1 郭郑湖 T4类浮游病毒遗传多样性的时间变化 郭郑湖为轻度富营养化,且受人为干扰较小(相对于庙湖而言).在系统发育树上(图1),该湖冬季序列与夏季界限明显,几乎完全位于不同的分支上,并且冬季序列的分布更加集中,多样性较小,而夏季序列则占据较多分支,多样性更丰富,这与组内发散度计算结果也一致.可能由于夏季浮游病毒数量远高于冬季,且冬夏两季宿主类型的不同,导致浮游病毒的种类区别较大[18-19].
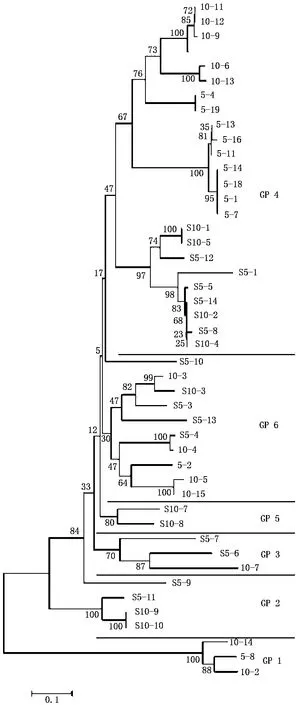
图2 本研究所得序列系统发育树Fig.2 Phylogenetic tree of the sequences we obtained
3.2.2 庙湖 T4类浮游病毒遗传多样性的时间变化 庙湖为重度富营养化,2009年 6~10月期间,该湖形成了严重的铜绿微囊藻水华.与郭郑湖不同的是,庙湖冬夏两季的 g23序列之间并没有明显的界限,它们在进化树上分布相对均匀(图1),冬夏两季的病毒种类之间并无明显差异.由于庙湖毗邻居民区和餐饮点,常年受到严重的人为干扰,人为因素将对浮游病毒的多样性产生明显影响,并在有可能一定程度上掩盖自然(季节)变化对浮游病毒的影响[20],因此该湖两个季节浮游病毒优势种之间的亲缘关系很近.另外,根据组内发散度结果,庙湖夏季病毒多样性比冬季小,很可能是因为该水体夏季发生了严重的铜绿微囊藻水华,导致宿主生物的多样性下降,从而使浮游病毒的多样性随之下降.
4 结论
4.1 随机克隆的方法用来检验淡水水体浮游病毒多样性是可行的.
4.2 淡水富营养化水体中的 T4类浮游病毒的遗传多样性较高且具有明显的时空差异,说明富营养化水平的差异和季节变化将影响浮游病毒的种群结构.
4.3 淡水富营养化水体中,仅部分g23序列与海洋 T4类浮游病毒同源性较高,另一部分序列则可能代表了淡水富营养化水体中特有的T4类浮游病毒.
4.4 人为干扰会明显影响 T4类浮游病毒的遗传多样性.
[1] Wommack K E, Ravel J, Hill R T, et al. Population dynamics of Chesapeake Bay virioplankton: total-community analysis by pulse-field gel electrophoresis [J]. Appl. Environ. Microbiol.,1999,65(1):231-240.
[2] Bergh O, orsheim K Y, Bratbak B G, et al. High abundance of viruses found in aquatic environments [J]. Nature, 1989,340:467-468.
[3] Andre´ M Comeau, Henry M Krisch. The capsid of the T4 phage superfamily: the evolution, diversity, and structure of some of the most prevalent proteins in the biosphere [J]. Mol. Biol. Evol.,2008,25(7):1321-1332.
[4] Filee J, Tetart F, Suttle C A, et al. Marine T4-type bacteriophages,a ubiquitous component of the dark matter of the biosphere [J].PNAS, 2005,102(35):12471-12476.
[5] Fujii T, Nakayama N, Nishida M, et al. Novel capsid genes (g23)of T4-type bacteriophages in a Japanese paddy field [J]. Soil Biol.Biochem., 2008,40(5):1049-1058.
[6] Liu Yan-Ming, Zhang Qi-Ya, Yuan Xiu-Ping. Studies on abundance and morphological diversity of virioplankton in the Donghu Lake, Wuhan [J]. Acta Hydrobiol. Sinica, 2005,29(1):1-6.
[7] Liu Y M, Yuan X P, Zhang Q Y. Spatial distribution and morphologic diversity of virioplankton in Lake Donghu, China [J].Acta OECOL., 2006,29(3):328-334.
[8] 刘艳鸣,张奇亚.利用脉冲场凝胶电泳测定东湖浮游病毒基因组的大小 [J]. 武汉大学学报(理学版), 2005,51(s2):238-240.
[9] Jia Z, Ishihara R, Nakajima Y, et al. Molecular characterization of T4-type bacteriophages in a rice field [J]. Environ. Microbiol.,2007,9:1091-1096.
[10] Ursula Dorigo, Stéphan Jacquet. Cyanophage diversity, inferred from g20 gene analyses, in the largest natural lake in France,Lake Bourget [J]. Appl. Environ. Microbiol., 2004,70(2):1017-1022.
[11] Marston M F, Sallee J L. Genetic diversity and temporal variation in the cyanophage community infesting marine Synechococcus species in Rhode Island’s coastal water [J]. Appl. Environ.Microbiol., 2003,69:4639-4647.
[12] Zhong Y, Chen F, Wilhelm S W, et al. Phylogenetic diversity of marine cyanophage isolates and natural virus communities as revealed by sequences of viral capsid assembly protein gene g20[J]. Appl. Environ. Microbiol., 2002,68:1576-1584.
[13] Sullivan M B, Waterbury J B, Chisholm S W. Cyanophages infecting the oceanic cyanobacterium Prochlorococcus [J]. Nature,2003,424:1047-1051.
[14] Becker S, Fahrbachl M. Quantitative tracing, by Taq nuclease assays, of a Synechococcus ecotype in a highly diversified natural population [J]. Appl. Environ. Microbiol., 2002,68:4486-4494.
[15] Urbach E, Scanlan D J, Distel D L, et al. Rapid diversification of marine picoplankton with dissimilar light harvesting structures inferred from sequences of Prochlorococcus and Synechococcus(cyanobacteria) [J]. J. Mol. Evol., 1998,46:188-201.
[16] Crosbie N D, Pőckl M, Weisse T. Dispersal and phylogenetic diversity of nonmarine picocyanobacteria, inferred from 16S rRNA gene and cpcBA-intergenic spacer sequences analyses [J].Appl. Environ. Microbiol., 2003,69:5716-5721.
[17] 罗文清,鞠 川,程 凯,等.一种浓缩噬藻体的反冲超滤技术[J]. 中国病毒学, 2003,18(4):397-400.
[18] Wommack K E, Hill R T, Kessel M, et al. Distribution of viruses in the ChesapeakeBay [J]. Appl. Environ. Microbiol., 1992,58:2965-2970.
[19] Suttle C A, Cyanophages and their role in the ecology of cyanobacteria. In: Brian A, Malcolm P, eds. The ecology of cyanobacteria. Netherlands Dordrecht: Kluwer academic publishers, 2000.563-589.
[20] Jiao N Z, Zhao Y L, Luo T W, et al. Natural and anthropogenic forcing on the dynamics of virloplankton in the yangtze river estuary [J]. J Mar. Biol. Assoc. UK., 2006,86(3):543-550.
Genetic diversity of T4 virioplankton, inferred from g23 gene, in Wuhan Donghu Lake.
HUANG Hui-zhen, CHENG Kai, XU Min, ZHAO Yi-jun*(Hubei Key Laboratory of Urban Water Environmental Ecology, Central China Normal University, Wuhan 430079, China). China Environmental Science, 2011,31(3):443~447
2 sub-lakes in Donghu Lake: Guozheng Lake and Miaohu Lake were chosen, and sampled seperately in winter and summer. g23 gene of T4 virioplankton was amplified, randomly sequenced certain clones. 46 sequences were got.Phylogenetic analysis showed that these sequences fell into 6 independent groups and exihibited high diversity. The phylogenetic analysis also suggests that: 1) the eutrophication level and season change have something to do with the structure of virioplankton community; 2) only part of the sequences are closely related to marine T4 virioplankton, so the others may represent eutrophicated freshwater virioplankton; 3) anthropogenic forcing can obviously influence the genetic diversity of T4 virioplankton in certain freshwaters.
g23 gene;Donghu Lake;T4;virioplankton;genetic diversity
X172
A
1000-6923(2011)03-0443-05
2010-08-08
国家自然科学基金资助项目(30670088);国家科技重大专项资助项目(2009ZX07105-001);华中师范大学基本科研业务费资助项目
* 责任作者, 教授, zhaoyj@mail.ccnu.edu.cn
黄慧珍(1984-),女,湖北武汉人,硕士研究生,研究方向为水体浮游病毒遗传多样性鉴定.发表论文1篇.

