金黄色葡萄球菌特异性PCR检测靶点的自动化筛选
范一灵,朱东升,胡瑜,史贤明
1 上海交通大学农业与生物学院陆伯勋食品安全研究中心 中美食品安全联合研究中心,上海 200240
2 上海市食品药品检验所,上海 201203
3 联合利华 (上海) 研发中心 联合利华中国研究所,上海 200335
金黄色葡萄球菌特异性PCR检测靶点的自动化筛选
范一灵1,2,朱东升1,3,胡瑜1,史贤明1
1 上海交通大学农业与生物学院陆伯勋食品安全研究中心 中美食品安全联合研究中心,上海 200240
2 上海市食品药品检验所,上海 201203
3 联合利华 (上海) 研发中心 联合利华中国研究所,上海 200335
旨在挖掘用于鉴定金黄色葡萄球菌的高特异性靶点及其PCR检测引物。采用C++语言编程,以金黄色葡萄球菌Staphylococcus aureus MRSA 252基因组编码序列为对象,对2 656个可编码区进行分析,获得特异性靶点序列,并设计PCR扩增引物。对包括葡萄球菌属11个种及其他细菌属在内的共计137株细菌验证引物特异性,筛选获得9个DNA序列,并设计了4对引物。经验证2对引物的特异性较好,其中引物SA3的基因组DNA检测限为13.7 fg/µL,菌体检测限为9.25×102CFU/mL。结果验证了特异性DNA靶点筛选平台的实用性,该方法突破了传统特异性靶点挖掘方法对检测对象的限制,适用性广,可移植性强。
金黄色葡萄球菌,基因组,生物信息比对分析,编码序列,C++编程
Abstract:The aim of this study was to establish a fast and accurate method for developing specific DNA sequences and PCR primers for the detection of Staphylococcus aureus. An automatic C++ program for genomic comparison was used to identify specific DNA sequences from the genome of S. aureus MRSA 252. Four primer pairs were obtained from 9 specific target sequences by comparison of 2656 coding sequences with our local genome database, and 2 pairs of primers were confirmed to be specific to S. aureus by PCR evaluation against 137 bacterial strains, including 11 species of Staphylococcus. Furthermore, the DNA detection sensitivity of primer SA3 was 13.7 fg/µL and the cell sensitivity for this primer was 9.25×102CFU/mL. This method has overcome the limitations of specific target mining in conventional assays, and it could be easily and widely used forother foodborne pathogens.
Keywords:Staphylococcus aureus, genome, bioinformatic comparison analysis, coding sequence, C++ programming
金黄色葡萄球菌 Staphylococcus aureus是一种重要的食源性和医源性致病菌[1],它可以分泌多种毒力因子,如肠毒素 (Staphylococcal enterotoxins,SEs)、毒素休克综合征毒素-1 (Staphylococcal toxic shock syndrome-1,TSST-1)、表皮剥脱毒素 (Exfoliative toxins,ETs)、葡萄球菌溶素 (Staphylolysin) 和凝固酶 (Coagulase) 等,可以引起食物中毒、毒素休克综合征、骨髓炎、坏死性肺炎和心内膜炎等严重疾病[2-3]。在国家标准检验方法中金黄色葡萄球菌检测方法仍然采用传统培养分离法,需要 5~6 d才能得到检验结果[4]。快速、有效地检测和鉴别金黄色葡萄球菌是急需解决的问题之一。近些年,以特异性DNA片段为靶点的分子生物学检测手段作为一种快速检测方法已被广泛采用[3,5-10]。
细菌特异性 DNA是指仅存在于某一目标细菌基因组内、而在其他非目标细菌基因组内缺失或者变异、可用于特异性检测和鉴定该目标细菌的DNA片段[3]。在细菌的分子生物学检测中,常用的特异性保守区域DNA片段多位于16S rRNA和23S rRNA区。另外,nuc1基因是检测金黄色葡萄球菌的重要靶点,它可以编码产生一种金黄色葡萄球菌特有的与凝固酶相关的耐热核酸酶 (TNase)[6-8,11]。除了上述检测靶点外,其他特异基因片段还有coa基因[12]和tRNA间隔序列等[3,5]。然而,上述目标DNA的检测位点的发现经历了十分漫长的研究过程。
随着致病微生物基因组数据的增加,生物信息学、基因组学和蛋白质组学的不断发展,大规模的基因组比对分析和蛋白质功能预测为快速、高效地发掘特异性DNA靶点提供了可能[13-15]。通过自动化程序对基因组数据库编码序列 (Coding sequences,CDS) 的比对和筛选,可以大幅度提高特异性检测位点的发掘效率。
本研究在组建本地细菌基因组数据库的基础上,建立了基于 C++编程的金黄色葡萄球菌特异性DNA序列自动化筛选方法。利用该方法对金黄色葡萄球菌MRSA 252的全基因组编码序列进行分析,以获得特异性DNA片段。
1 材料与方法
1.1 细菌培养及基因组DNA提取
本实验所用菌株共 137株,其中金黄色葡萄球菌分离株来自临床样本 (表 1)。实验所用菌株均接种于Luria-Bertani (LB) 培养基中,160 r/min、37 ℃振荡培养18 h备用。
细菌基因组DNA提取和纯化采用QIAGEN公司DNeasy Blood and Tissue Kit试剂盒,按试剂盒说明书操作,用 100 µL TE缓冲液洗脱细菌基因组DNA,存放于−20 ℃备用。
1.2 基因组CDS自动化比对
在GenBank细菌全基因组数据库 (ftp://ftp.ncbi.nlm.nih.gov) 中,选取具有代表性的139个细菌的基因组数据,利用Blast软件 (版本号Blast-2.2.9-ia32)建立本地基因组数据库 (不包含 14个金黄色葡萄球菌基因组信息)。采用基于C++平台的基因组CDS比对程序[16],分析金黄色葡萄球菌MRSA252的全基因组CDS信息 (文件来源NCBI数据NC_002952.ffn)。筛选参数为E>4.0,L>400 bp,由程序自动比对每个Blast生成文件中的E值。对程序筛选出的CDS序列通过网上 Blast比对 (http://blast.ncbi.nlm.nih.gov),验证自动筛选的CDS特异性。
1.3 引物的设计及PCR检测
采用PRIMER PREMIER 5.0软件,对筛选出的CDS区 DNA序列设计引物 (表 1)。引物由上海生工生物工程技术服务有限公司合成。
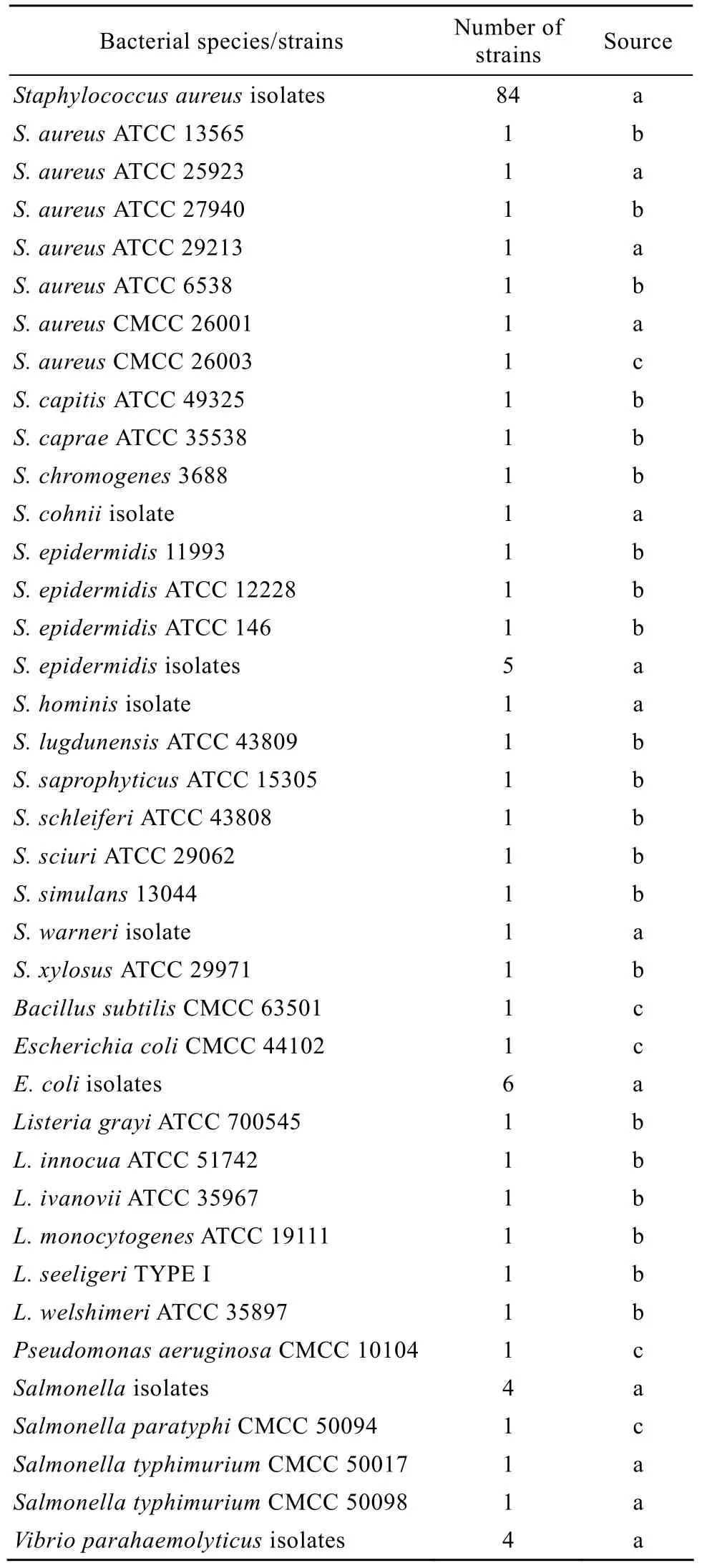
表1 实验菌株Table 1 Bacteria strains tested in this assay
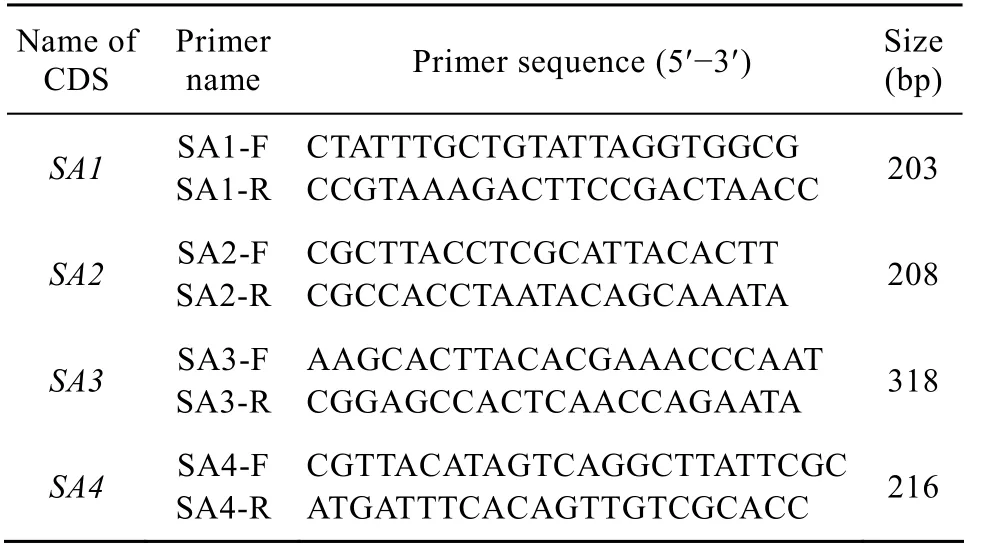
表2 特异性CDS区段的PCR扩增引物Table 2 PCR primers against specific CDS
PCR扩增体系为:1.0 µL细菌基因组DNA;1 U rTaq DNA聚合酶 (天根生化科技有限公司,北京);1×PCR 缓冲液 [200 mmol/L Tris-HCl(pH 8.4);200 mmol/L KCl;100 mmol/L (NH4)2SO4];2.0 mmol/L MgCl2;4种 dNTPs各 0.25 mmol/L;上、下游引物各10 nmol/L;加蒸馏水至25 µL。
采用 Peltier Thermal Cycler PTC-200 PCR仪(Bio-Rad Laboratories,USA) 进行PCR扩增。反应条件为:105 ℃热盖;95 ℃变性5 min;95 ℃变性30 s,56 ℃退火30 s,72 ℃延伸 30 s,30个循环;72 ℃延伸 5 min。
将6 µL PCR产物经1.0%琼脂糖凝胶电泳,用10 µg/mL的EB溶液染色15 min,置于紫外成像仪(MultiImage Light Cabinet, Alphalmager) 内观察。DNA分子量标准选用100 bp DNA ladder (天根生化科技有限公司,北京)。
1.4 引物特异性评价和检测限评价
初步评价实验选择15株细菌,包括11株金黄色葡萄球菌和4株非金黄色葡萄球菌。在上述PCR检测体系中,利用设计的 4对引物对细菌基因组DNA进行扩增,评价引物的特异性。将初步评价结果较好的特异性引物,应用于全部其他细菌的检测验证中。
选取特异性较高的引物进行检测限的评价,分别采用金黄色葡萄球菌标准菌株 CMCC 26003的基因组 DNA浓度 10倍稀释组和细菌菌体浓度 10倍稀释组,对引物的DNA检测限和菌体检测限两项指标进行评价。基因组 DNA的浓度梯度为:1.37×107、1.37×106、1.37×105、1.37×104、1.37×103、1.37×102、13.7、1.37 fg/µL。细菌菌体浓度梯度为:9.25×106、9.25×105、9.25×104、9.25×103、9.25×102、9.25、0.925、0 CFU/mL (空白对照)。
1.5 人工污染样品的制备
取新鲜鸡蛋蛋液200 g,混匀后分成蛋液含量为25.0 g的样品8份。其中随机抽取2份蛋液,按照国标 GB/T 4789.10-2008金黄色葡萄球菌检验中规定的方法进行检验。其余6份蛋液分为3组,向3组蛋液中加入金黄色葡萄球菌标准菌株 CMCC 26003菌悬液,接种量分别为9.25×102CFU/25 g、9.25 CFU/25 g和0.925 CFU/25 g。将人工污染的蛋液样品加入到225 mL生理盐水中混匀,吸取5 mL上述液体加入50 mL 7.5% NaCl肉汤中培养8 h。取1 mL肉汤培养物,按1.3中所述步骤进行PCR检测。每组做 2个平行试验。人工污染的样品的后续步骤仍按国标方法进行检测,比较两种方法的检测结果。
1.6 食品污染调查应用
调查市售加工点心类产品,包括汉堡、寿司、三明治、中西式糕点、包子、月饼、批萨和肉卷等共计185件,按国标规定的前处理方法进行增菌培养,取增菌肉汤1 mL采用本研究所用引物和PCR扩增条件进行检查;并同时取增菌肉汤分别采用DuPont BAX Q7荧光定量PCR方法和国标方法进行检查,比较三者对金黄色葡萄球菌的检出情况。
2 结果
2.1 特异性CDS的自动化筛选
金黄色葡萄球菌MRSA 252全基因组包含已注释的编码序列共有2 656个,通过C++筛选程序,获得9个E>4.0且L>400 bp的CDS,分别命名为编码区 SA1至 SA9 (表 3)。通过比对验证,选出 4个特异性较好的CDS区设计检测引物。
2.2 引物的特异性评价
将SA1-SA4这4对引物分别对11株金黄色葡萄球菌和4株非金黄色葡萄球菌的基因组DNA进行PCR扩增实验。引物SA2和SA3对所用金黄色葡萄球菌 DNA 具有特异性 (图 1)。137株细菌基因组DNA的PCR试验结果表明:引物SA2和SA3可以特异性鉴别金黄色葡萄球菌基因组DNA,试验所用非金黄色葡萄球菌基因组DNA未见PCR扩增条带(包括葡萄球菌属内的其他13个种)。
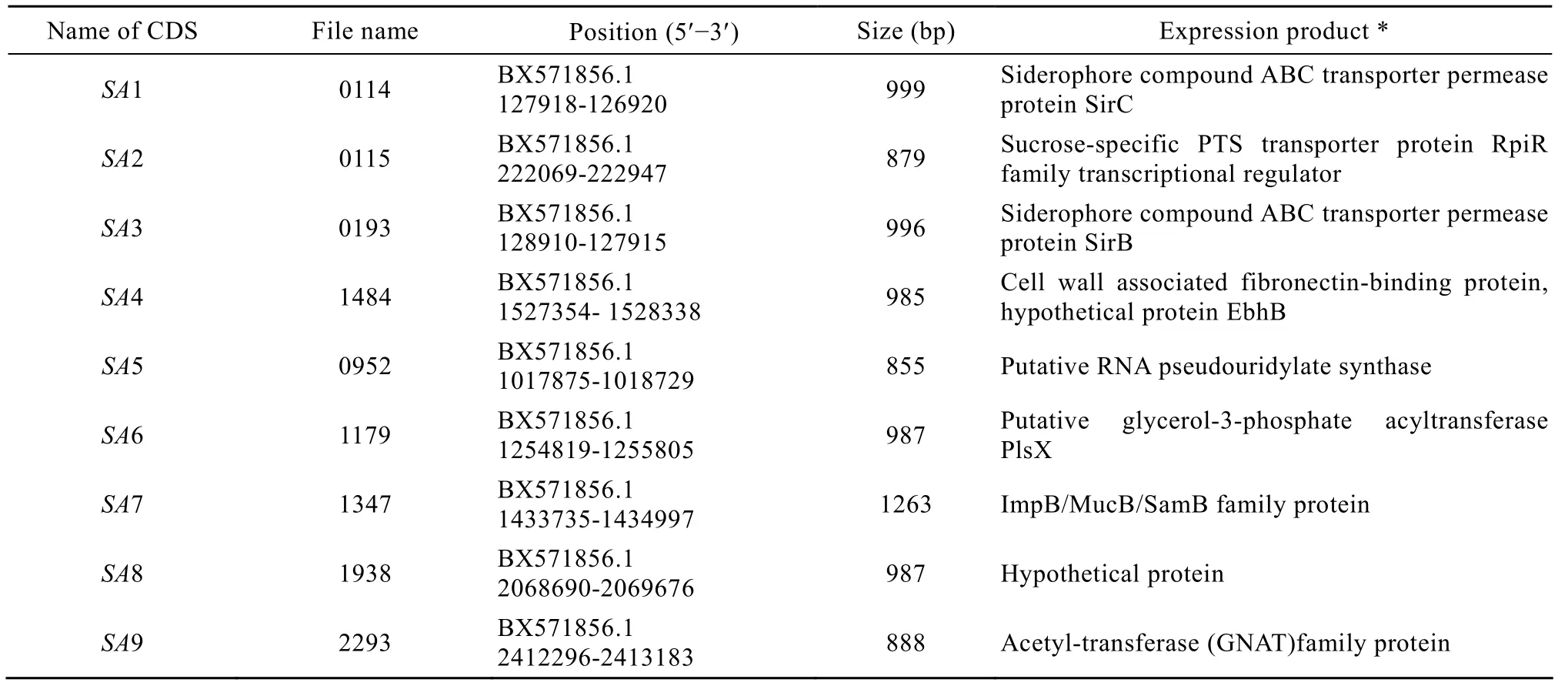
表3 金黄色葡萄球菌特异性CDS的自动化筛选Table 3 Screening of specific CDS from S. aureus
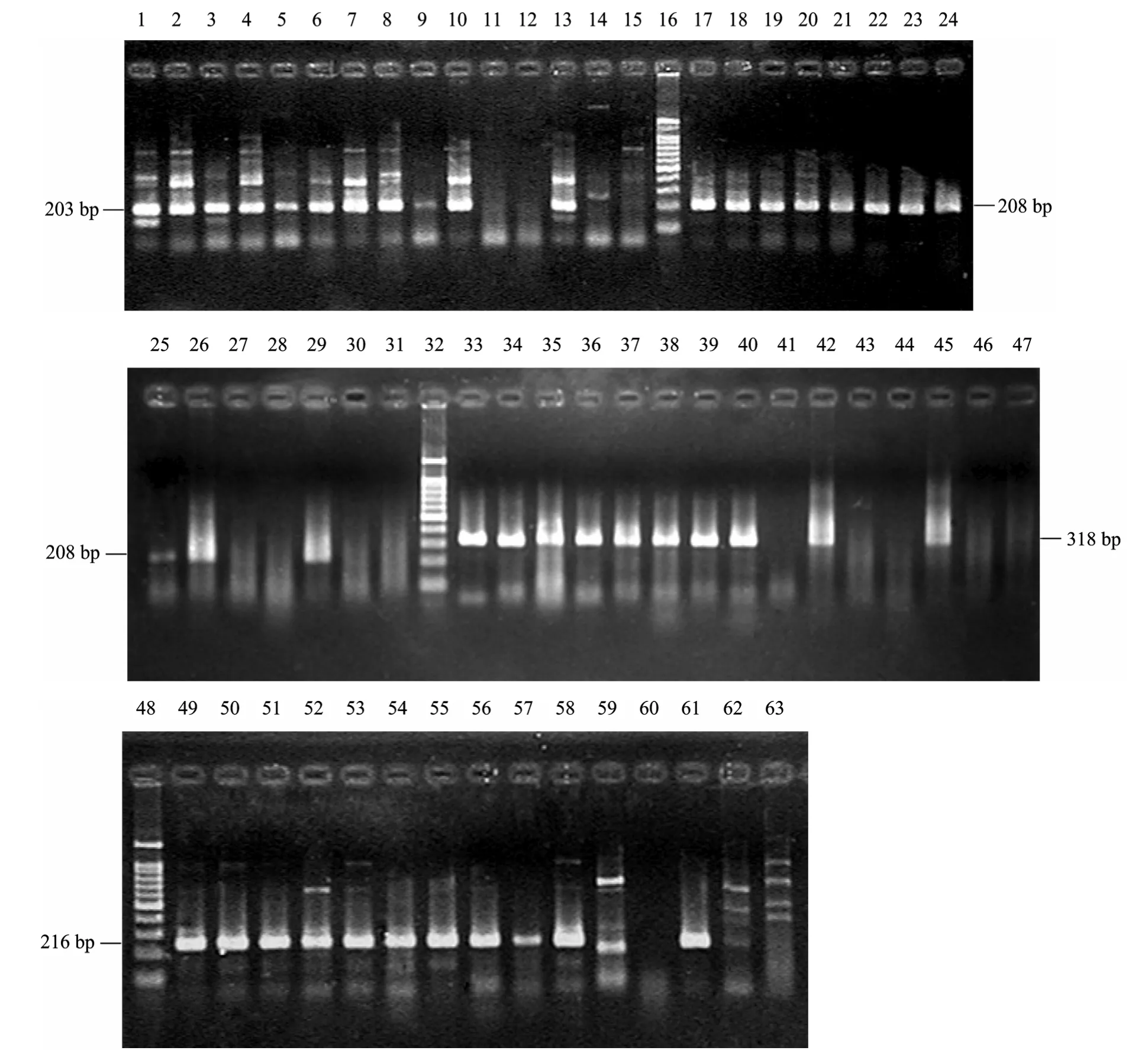
图1 金黄色葡萄球菌4对PCR引物的特异性验证图Fig. 1 PCR specificity evaluations on four primers for S. aureus. 1−15: results for primer SA1; 17−31: results for primer SA2;33−47: results for primer SA3; 49−63: results for primer SA4. The bacterial strains (From left to right) tested for each pair of primers were S. aureus ATCC 27940, S. aureus ATCC 13565, S. aureus ATCC 25923, S. aureus ATCC 6538, S. aureus CMCC 26001, S. aureus ATCC6538, S. aureus ATCC25923, S. aureus ATCC12228, S. aureus ATCC29213, S. aureus strain B255, V.Parahaemolyticus strian 39001, Salmonella strain T16, S. aureus strain G64, S. capitis ATCC 49325 and E. coli strain 133; 16, 32,48: standard DNA marker.
2.3 引物的检测限评价
通过PCR退火温度梯度试验 (48 ℃~62 ℃) 和镁离子浓度梯度试验 (0.5~3.0 mmol/L),对引物进行反应条件优化。在56 ℃温度下退火,镁离子浓度为2.0 mmol/L时,引物SA2和SA3的PCR产物具有明亮的单一扩增条带,PCR扩增效率较高。
根据特异性评价结果,采用优化的 PCR反应条件评价引物的检测限。引物SA2的基因组DNA检测限为 137 fg/µL (图 2,Lane 6);引物 SA3的基因组 DNA检测限为 13.7 fg/µL (图 2,Lane 15)。另外,引物SA2和引物SA3的菌体检测限都能达到9.25×102CFU/mL (图 3,Lane 5 和 Lane 15)。
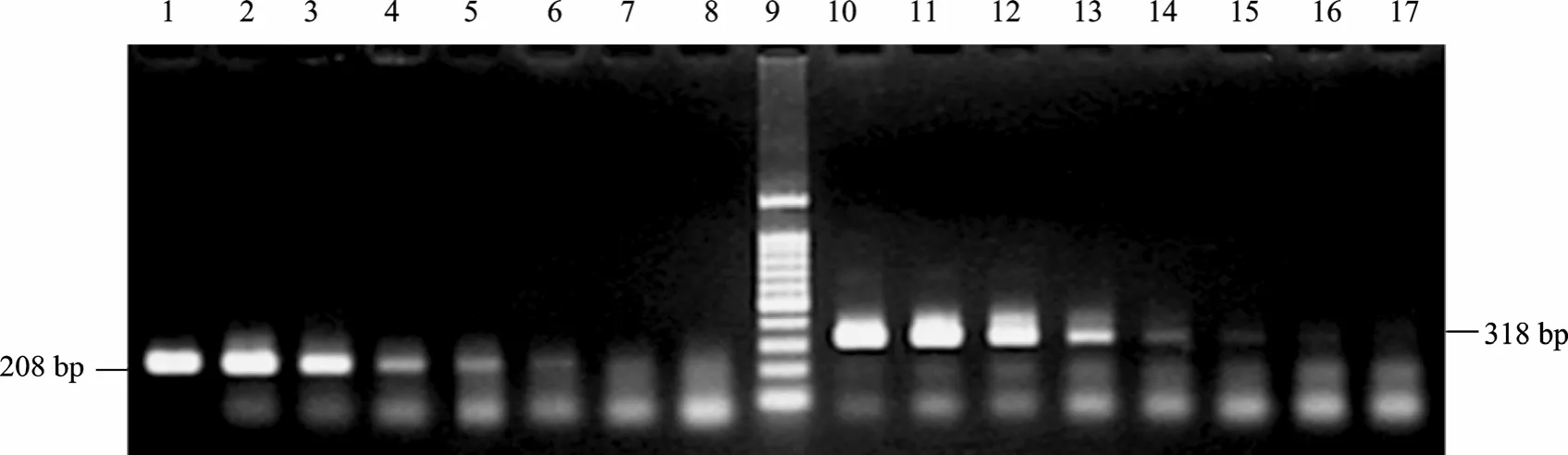
图2 引物SA2和SA3的基因组DNA检测限试验PCR扩增结果Fig. 2 PCR amplification of sensitivity tests genomic DNA of primer SA2 and SA3. 1−8: primer SA2 were applied; 10−17: primer SA3 were applied; 9: standard DNA marker. The genomic DNA of S. aureus ATCC 6538 were 1.37×107fg/µL, 1.37×106fg/µL,1.37×105fg/µL, 1.37×104fg/µL, 1.37×103fg/µL, 1.37×102fg/µL, 13.7 fg/µL and 1.37 fg/µL, respectively.

图3 引物SA2和SA3细菌菌体检测限试验PCR扩增结果Fig. 3 PCR amplification of cell sensitivity tests for primer SA2 and SA3. 1−9: primer SA2 were applied; 11−19: primer SA3 were applied. 1−9 and 11−19: the cell concentrations of S. aureus CMCC 26003 were 9.25×106CFU/mL, 9.25×105CFU/mL,9.25×104CFU/mL, 9.25×103CFU/mL, 9.25×102CFU/mL, 9.25 CFU/mL, 0.925 CFU/mL and negative control, respectively; 10:standard DNA marker (From bottom to top: 100 bp, 250 bp, 500 bp, 750 bp, 1 000 bp and 2 000 bp).
2.4 人工污染鸡蛋样品和食品污染调查应用
采用PCR方法 (含有引物SA2或SA3) 和国标方法均能检出样品中含接种量为9.25×102CFU/25 g和 9.25 CFU/25 g的金黄色葡萄球菌;然而,在含0.925 CFU/25 g金黄色葡萄球菌样品检测中,由于
接种量太低,国标方法和PCR方法均得到阴性结果。在调查检验市售 185件样品中,通过本研究的检测方法共检出金黄色葡萄球菌阳性3株,占全部样品的1.62%,与BAX Q7检测系统和国标检测方法得到的结果相同。
3 讨论
传统微生物特异性 DNA序列的发掘是基于对已知细菌特异性蛋白质的分析,然后对调控或编码该特异蛋白质的基因或DNA片段进行研究[3]。其中,不少序列既有种内特异性又存在多态性,如 coa基因和 16S rRNA序列在金黄色葡萄球菌种的最低相似度仅为79%,只有寻找这些区段内相对保守的部分进行引物设计来提高特异性。本实验通过生物信息学方法筛选出的4个金黄色葡萄球菌CDS区不存在种内多态性,保守性好。SA2基因在金黄色葡萄球菌种内相似性在 99%以上,SA1、SA3和 SA4基因在金黄色葡萄球菌种内相似性不低于93%。
分析金黄色葡萄球菌CDS序列对应的蛋白质产物信息可知,这些编码序列均与细胞膜和细胞壁物质转运和调控的基因有关 (表3)。其中,SA1和SA3的表达产物分别为透性酶SirC和SirB (Siderophore compound ABC transporter permease protein SirC and SirB)。通过对Sir系列蛋白质序列的分析研究显示,许多细菌都含有类似 ABC转运子功能的离子转运蛋白[18-20],如表面葡萄球菌和沙门氏菌等[20-22]。通过DNA序列分析,金黄色葡萄球菌的sirB和sirC基因都具有很好的保守性[23],和本研究的结论相符。由于sirB和sirC基因可能是多拷贝基因[24],更适合用于对灵敏度要求较高的致病菌检测方法。SA2的表达产物为蔗糖专属 PTS转运蛋白转录调控基因(Sucrose-specific PTS transporter protein RpiR family transcriptional regulator)。RpiR蛋白可能与编码磷酸核糖异构酶的rpiB基因调控有关[25]。SA4则被推定为细胞壁纤维连接蛋白基因,其表达产物是一个推定蛋白EbhB (Cell wall associated fibronectin-binding protein,hypothetical protein EbhB)。通过基因组比对发现 ebh基因广泛存在于金黄色葡萄球菌基因组中[26],与本实验结果相吻合。
大量可供分析的微生物基因组序列信息已成为生物信息学研究的重要内容[13-15]。本研究结合 C++程序调用Blast软件,对金黄色葡萄球菌基因组进行自动比对,筛选编码区特异性DNA序列。该方法简单实用,可以快速锁定微生物特异性检测位点,提高比对效率,为建立基于微生物特异性DNA序列的快速检测方法提供了大量关键的备选目标区间。
致谢:真诚感谢美国农业部东部研究中心George C.Paoli博士为本研究提供了大量标准菌株。
REFERENCES
[1] Crossley KB, Archer GL. The Staphylococci in Human Disease. 2nd ed. New York: Churchill Livingstone, 1998:253−255.
[2] Cowie SE, Ma I, Lee SK, et al. Nosocomial MRSA infection in vascular surgery patients: impact on patient outcome. Vasc Endovas Surg, 2005, 39(4): 327−334.
[3] Fan YL, Pan F, Shi XM. Common target genes of Staphylococcus aureus in molecular detection technique. J Microbiol, 2008, 28(3): 72−76.
范一灵, 潘峰, 史贤明. 金黄色葡萄球菌分子检测技术的常用靶基因. 微生物学杂志, 2008, 28(3): 72−76.
[4] GB/T 4789.10-2008, Detection of Staphylococcus aureus.Beijing: Standards Press of China, 2008.GB/T 4789.10-2008, 金黄色葡萄球菌检验. 北京: 中国标准出版社, 2008.
[5] Drancourt M, Raoult D. rpoB gene sequence-based identification of Staphylococcus species. J Clin Microbiol,2002, 40(4): 1333−1338.
[6] Brakstad OG, Aasbakk K, Maeland JA. Detection of Staphylococcus aureus by polymerase chain reaction amplification of the nuc gene. J Clin Microbiol, 1992,30(7): 1654−1660.
[7] Wilson IG, Cooper JE. Gilmour A. Detection of enterotoxigenic Staphylococcus aureus in dried skimmed milk: use of the polymerase chain reaction for amplification and detection of staphylococcal enterotoxin genes entB and entC1 and the thermonuclease gene nuc.Appl Environ Microbiol, 1991, 57(6): 1793−1798.
[8] Thomas LC, Gidding HF, Ginn AN, et al. Development of a real-time Staphylococcus aureus and MRSA (SAM-)PCR for routine blood culture. J Microbiol Methods, 2007,68(2): 296−302.
[9] Fan YL, Pan F, Paoli GC, et al. Development of a multiplex PCR method for detection of the genes encoding 16S rRNA, coagulase, methicillin resistance and enterotoxins in Staphylococcus aureus. J Rapid Methods Autom Microbiol, 2008, 16(4): 394−411.
[10] Wang XH, Xie BJ, Shi XM. Detection of Staphylococcus aureus pathogenetic factor by PCR. Food Machin, 2004,20(3): 48−50.
王小红, 谢笔钧, 史贤明. 金黄色葡萄球菌致病因子检测的PCR方法. 食品与机械, 2004年, 20(3): 48−50.
[11] Tang JN, Zhou R, Shi XM, et al. Two thermostable nucleases coexisted in Staphylococcus aureus: evidence from mutagenesis and in vitro expression. FEMS Microbiol Lett, 2008, 284(2): 176−183.
[12] Maes N, De Gheldre Y, De Ryck R, et al. Rapid and accurate identification of Staphylococcus species by tRNA intergenic spacer length polymorphism analysis. J Clin Microbiol, 1997, 35(10): 2477−2481.
[13] Altschul SF, Gish W, Miller W, et al. Basic local alignment search tool. J Mol Biol, 1990,215(3): 403−410.
[14] Johnson M, Zaretskaya I, Raytselis Y, et al. NCBI BLAST:a better web interface. Nucleic Acids Res, 2008(36): 5−9.
[15] Karlin S, Altschul SF. Methods for assessing the statistical significance of molecular sequence features by using general scoring schemes. Proc Natl Acad Sci USA, 1990,87(6): 2264−2268.
[16] Zhu DS, Zhou M, Fan YL, et al. Identification of new target sequences for PCR detection of Vibrio parahaemolyticus by genome comparison. J Rapid Methods Autom Microbiol, 2009, 17(1): 67−79.
[17] Tian J, Ji R, Yang J, et al. Staphylococcus aureus in food determined by polymerase chain reaction. J Hygiene Res,2007, 36(2): 183−186.
田静, 计融, 杨军, 等. PCR方法快速检测食品中的金黄色葡萄球菌. 卫生研究, 2007, 36(2): 183−186.
[18] Linton KJ, Higgins CF. The Escherichia coli ATP-binding cassette (ABC) proteins. Mol Microbiol, 1998, 28(1):5−13.
[19] Nikaido H, Hall JA. Overview of bacterial ABC transporters. Methods Enzymol, 1998(292): 3−20.
[20] Boos W, Eppler T. Prokaryotic Binding Protein-Dependent ABC Transporters. Microbial Transport Systems,Germany: Wiley-VCH, 2003: 77−114.
[21] Heinrichs JH, Gatlin LE, Kunsch C, et al. Identification and characterization of SirA, an iron-regulated protein from Staphylococcus aureus. J Bacteriol, 1999, 181(5):1436−1443.
[22] Hill PJ, Cockayne A, Landers P, et al. SirR, a novel iron-dependent repressor in Staphylococcus epidermidis.Infect Immun, 1998, 66(9): 4123−4129.
[23] Dale SE, Sebulsky MT, Heinrichs DE. Involvement of SirABC in iron-siderophore import in Staphylococcus aureus. J Bacteriol, 2004, 186(24): 8356−8362.
[24] Rakeman JL, Bonifield HR, Miller SI. A HilA-independent pathway to Salmonella typhimurium invasion gene transcription. J Bacteriol, 1999, 181(10):3096−3104.
[25] Sorensen KI, Hove-Jensen B. Ribose catabolism of Escherichia coli: characterization of the rpiB gene encoding ribose phosphate isomerase B and of the rpiR gene, which is involved in regulation of rpiB expression. J Bacteriol, 1996, 178(4): 1003−1011.
[26] Tanaka Y, Sakamoto S, Kuroda M, et al. A helical string of alternately connected three-helix bundles for the cell wall-associated adhesion protein Ebh from Staphylococcus aureus. Structure, 2008, 16(3): 488−496.
Screening of specific target sequences for the PCR detection of Staphylococcus aureus by automatic genomic comparison
Yiling Fan1,2, Dongsheng Zhu1,3, Yu Hu1, and Xianming Shi1
1 Joint Sino-US Food Safety Research Center & Bor Luh Food Safety Center, School of Agriculture and Biology, Shanghai Jiao Tong University,Shanghai 200240, China
2 Shanghai Institute for Food and Drug Control, Shanghai 201203, China
3 Unilever Discover Shanghai, Unilever Research and Development Shanghai, Shanghai 200335, China
Received: July 8, 2010; Accepted: January 20, 2011
Supported by: National Key Technology Research and Development Program of China (No. 2009BAK43B31), Science and Technology Commission of Shanghai Municipality (Nos. 10142201300, 08142200700, 08391911000).
Corresponding author: Xianming Shi. Tel/Fax: +86-21-34206616; E-mail: xmshi@sjtu.edu.cn
国家科技支撑计划 (No. 2009BAK43B31),上海市科委项目 (Nos. 10142201300, 08142200700, 08391911000) 资助。

