混配氧钒配合物[VO(Naph-Phe)(Phen)]的合成、晶体结构及与DNA作用研究
边 琳 李连之 王 霞 黄 蕾 蒲雪炜 董建方
(聊城大学化学化工学院,聊城 252059)
混配氧钒配合物[VO(Naph-Phe)(Phen)]的合成、晶体结构及与DNA作用研究
边 琳 李连之*王 霞 黄 蕾 蒲雪炜 董建方
(聊城大学化学化工学院,聊城 252059)
合成了一个新的L-苯丙氨酸萘酚醛希夫碱(Naph-Phe)和邻菲咯啉(Phen)混配氧钒配合物[VO(Naph-Phe)(Phen)],通过元素分析和红外光谱进行了表征。X-射线单晶衍射分析表明,该晶体属于单斜晶系,P21/c空间群,晶胞参数为:a=1.02785(11)nm,b=2.1865(3)nm,c=1.171150(13)nm,β=94.7470(10)°,V=2.6229(4)nm3,Z=4,F(000)=1164,R1=0.0767,wR2=0.1655,S=1.092。利用紫外吸收光谱、荧光光谱、圆二色光谱和粘度测量等研究了配合物与小牛胸腺DNA(CT-DNA)的作用,结果表明配合物以插入方式与CT-DNA作用。琼脂糖凝胶电泳法研究了配合物与pBR322 DNA的作用,表明它可切割超螺旋型DNA为缺刻型DNA。
钒配合物;希夫碱;晶体结构;DNA
金属配合物与DNA的相互作用一直是化学生物学的研究热点[1-2]。许多金属配合物能与DNA结合并切割DNA,例如抗癌药物博来霉素的DNA切割活性就依赖于与金属离子的配合。因此,近年来人们致力于合成以DNA为靶点的金属配合物药物,并研究它们与DNA的相互作用及机理。钒是人体必需的微量元素,其配合物具有抗肿瘤、抗癌、类胰岛素样等生物活性[3-6]。氨基酸希夫碱与第一过渡金属元素形成的配合物表现了良好的抗菌、抗结核等生物活性[7]。1,10-邻菲咯啉是一种重要的杂环配体,其配合物具有DNA结合性和DNA切割活性[8-9],是潜在的抗肿瘤药物。我们实验室已合成出一系列以希夫碱与1,10-邻菲咯啉为配体的氧钒配合物[10-13]。本文报道了L-苯丙氨酸萘酚醛希夫碱(Naph-Phe)及1,10-邻菲咯啉(Phen)混配体氧钒配合物的合成表征及晶体结构,并用紫外吸收光谱、荧光光谱、圆二色光谱和粘度测量等研究了它与小牛胸腺DNA(CT-DNA)相互作用,还利用琼脂糖凝胶电泳法研究了配合物对pBR322 DNA的切割活性。
1 实验部分
1.1 试剂与仪器
溴化乙锭 (EB)、琼脂糖和小牛胸腺DNA(CTDNA)均购自华美生物工程公司,CT-DNA用10 mmol·L-1Tris-HCl/10 mmol·L-1NaCl,pH 7.1 缓冲溶液配制,经UV谱测定A260/A280>1.8,浓度以ε260=6600 L·mol-1·cm-1确定[14]。 三羟甲基氨基甲烷(Tris)购自济南爱博经贸有限公司,L-苯丙氨酸 (生化试剂)购自北京经科宏达生物技术有限公司,硫酸氧钒、1,10-邻菲咯啉和 2-羟基-1-萘甲醛(萘酚醛)等均为分析纯试剂。
Bruker Smart-1000 CCD型衍射仪,Nicolet 460型红外光谱仪,Perkin-Elmer 2400Ⅱ型元素分析仪,HP 8453A型紫外-可见分阵列二极管分光光度计 (USA),LS55 型荧光光谱仪 (Perkin Elmer,USA),JASCO J-810型圆二色光谱仪(Japan),乌氏粘度计,Powerpac 300 电泳仪(Bio-Rad),DYY-Ⅲ电泳槽(北京六一仪器厂)。
1.2 配合物的合成
将 1.0 mmol L-苯丙氨酸和 1.0 mmol氢氧化钾溶于5 mL无水甲醇中,加热搅拌直至完全溶解后,向溶液中逐滴加入1 mmol萘酚醛的无水甲醇溶液(5 mL),于60℃下搅拌1 h。然后加入1 mmol硫酸氧钒的水溶液(2 mL),加热回流反应2 h后,继续加入1 mmol 1,10-邻菲咯啉的无水甲醇溶液 (5 mL)。加热搅拌3 h后,冷却过滤,沉淀用二氯甲烷与无水甲醇的混合溶液溶解,过滤,滤液室温下静置数天,得到适合单晶衍射的红褐色晶体。产率约为60%。元素分析按 C32H23N3O4V 计算值(%):C 68.03,H 4.08,N 7.44;实测值(%):C 67.97,H 4.00,N 7.32。 配合物的 IR(cm-1):3 435.8(s,νO-H);1 647.3(vs,νC=N);1 620.9(vs,νCOO);935.7(s,νV=O);447.5(w,νV-N)。
1.3 晶体结构测定及解析
选取尺寸为 0.40 mm×0.36 mm×0.30 mm 的单晶置于Bruker Smart-1000 CCD型衍射仪上,以石墨单色化的 Mo Kα(λ=0.071073 nm)辐射为光源,在1.86°≤θ≤25.02°范围内以 φ-ω 扫描方式于 298(2)K下共收集衍射点12 694个,其中独立衍射点4 503 个(Rint=0.094 6),选取 I>2σ(I)的 2 623 个可观察点用于结构解析和修正,全部数据均经经验吸收校正。晶体结构解析和结构精修均采用SHELXTL软件完成[15]。结构由直接法解出,其余的非氢原子的坐标在以后的数轮差值Fourier合成中陆续确定。对全部非氢原子的坐标及各向异性参数进行了全矩阵最小二乘法修正。最终的偏差因子为R1=0.0767,wR2=0.1655,S=1.092。差值电子密度最高和最低峰分别为 768 e·nm-3和-300 e·nm-3,主要晶体学数据列于表1。
CCDC:770157。
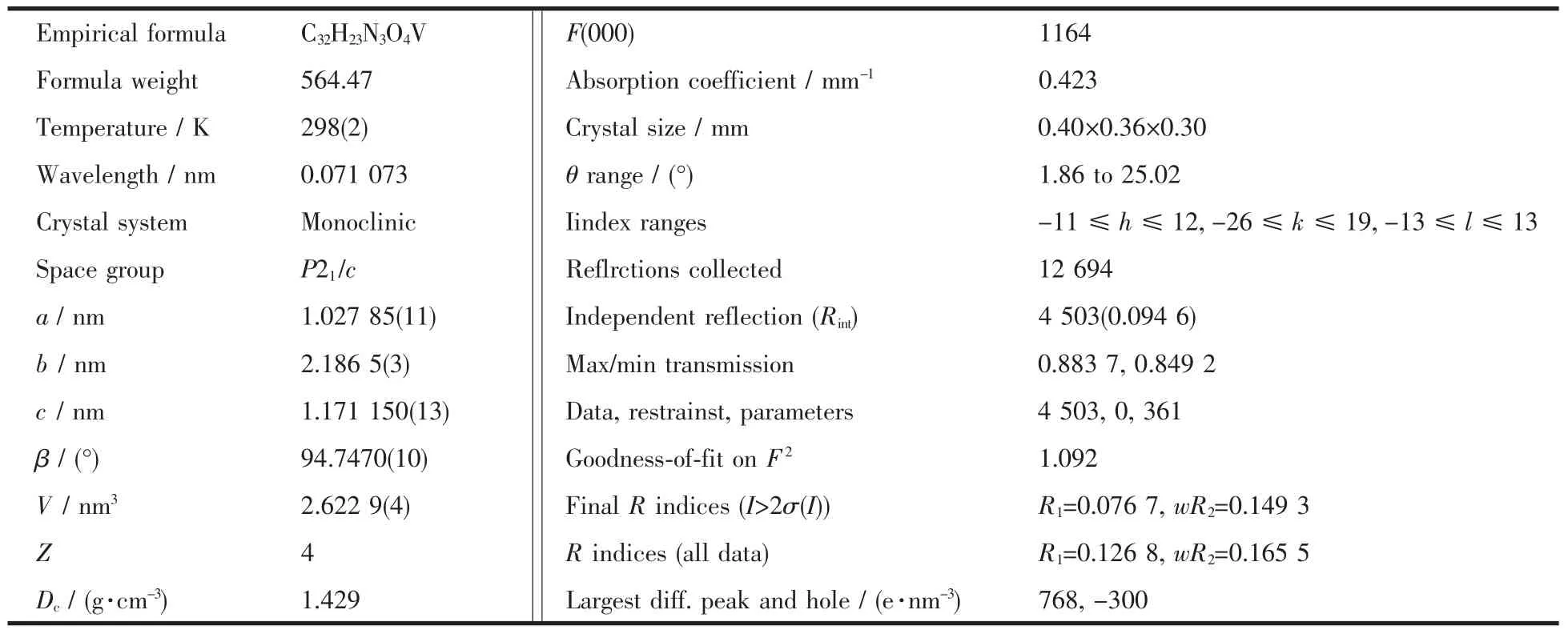
表1 配合物的晶体学数据Table 1 Crystallographic data for the title complex
1.4 配合物与DNA作用的实验方法
1.4.1 紫外吸收光谱测定
反应体系为 10 mmol·L-1Tris-HCl/10 mmol·L-1NaCl,pH 7.1缓冲溶液。固定配合物的浓度为 20 μmol·L-1,逐渐增大 CT-DNA 的浓度,反应溶液混合均匀后静止1 h,以缓冲溶液或含相同浓度DNA缓冲溶液为参比,在200~400 nm波长范围内扫描,分别测定不同DNA浓度下配合物与DNA混合溶液的紫外吸收光谱。
1.4.2 配合物对EB-DNA荧光光谱影响的测定
反应体系为 10 mmol·L-1Tris-HCl/10 mmol·L-1NaCl,pH 7.1缓冲溶液。固定 CT-DNA浓度为 25 μmol·L-1,EB 浓度为 3 μmol·L-1, 逐渐增大配合物的浓度,将反应溶液混合均匀静止2 h进行测量。仪器测量参数:激发波长为258 nm;狭缝宽度为5 nm;扫描波长范围为540~700 nm。
1.4.3 圆二色光谱测定
反应体系为 10 mmol·L-1Tris-HCl/10 mmol·L-1NaCl,pH 7.1缓冲溶液。固定CT-DNA的浓度为10 mmol·L-1,逐渐增加配合物的浓度,混合均匀后室温静止2 h后,分别测定DNA溶液和配合物与DNA混合液的圆二色光谱。设置仪器参数:扫描速度为100 nm·min-1;分辨率为 0.2 nm;累计次数 3 次;路径长度为1 cm;波长范围为220~320 nm。
1.4.4 粘度测量
反应体系为 10 mmol·L-1Tris-HCl/10 mmol·L-1NaCl,pH 7.1缓冲溶液。 固定CT-DNA的浓度100 μmol·L-1,依次增加配合物的浓度,温度恒定在(30±0.1)℃,分别测定不同 r值(r=cVOL/cDNA,cVOL为配合物的浓度)下配合物(或EB)与DNA混合液流经毛细管的时间t,每个样品溶液重复测定时间3次,取平均值。 后根据相对粘度公式 η=(t-t0)/t0,以(η/η0)1/3对 r作图,其中t0为缓冲溶液流经毛细管所需的时间,η0为r=0时DNA溶液的相对粘度。
1.4.5 琼脂糖凝胶电泳实验
固定质粒pBR322 DNA的浓度,逐渐增加配合物的浓度,混合均匀,37℃水浴温热3 h后进行电泳分析。在0.8%的琼脂糖凝胶上,60 V的电压下电泳约2 h,电泳溶液为TBE缓冲溶液。取出凝胶,在凝胶成像分析系统上照相。
2 结果与讨论
2.1 晶体结构解析
配合物的主要键长和键角列于表2,配合物的晶体结构见图1。分子结构表明,钒(Ⅳ)为六配位,V(1)-O(4)的键长为 0.160 0(3)nm,为典型的钒氧双键 (V=O)[16]。由表2中配合物的相关键长和键角可知,钒原子处于变形的八面体配位环境中,三齿的L-苯丙氨酸萘酚醛Schiff碱配体上的酚羟基O(3)原子、亚氨基 N(1)原子、羧基 O(1)原子和 1,10-邻菲咯啉配体上的N(3)原子处于变形八面体的赤道平面,而钒氧O(4)原子和1,10-邻菲咯啉上的N(2)原子则占据八面体的轴向位置。O(4)原子和N(2)原子距离中心钒原子的距离分别为 0.1600(3)和0.2399(4)nm,中心钒原子偏向O(4)原子一方,偏离赤道平面的距离为0.03792(19)nm。O(4)和N(2)距离赤道平面的距离则分别为0.19747(36)和0.20052(41)nm。

图1 配合物的分子结构Fig.1 Molecular structure of the complex

表2 配合物的主要键长和键角Table 2 Selected bond lengths(nm)and angles(°)for the complex
另外,以V(1)原子为中心形成了一个五元环V(1)-O(1)-C(1)-C(2)-N(1)和一个六元环V(1)-O(3)-C(12)-C(11)-C(10)-N(1),六元环与五元环所处的平面形成的二面角为17.88(18)°。说明这两个环的共面性不是很好,但是它们的形成却可以增加配合物的稳定性。在配合物的晶体结构中,分子间通过弱的分子间氢键(表3)等相互作用彼此连接,形成了一维链状结构(图 2)。

表3 配合物的氢键键长和键角Table 3 Hydrogen bond lengths and angles for the complex
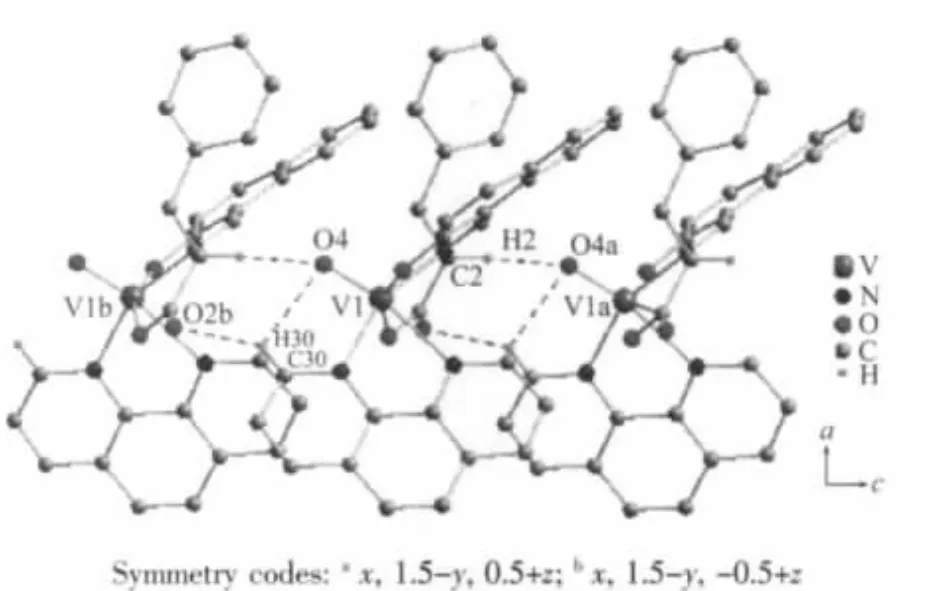
图2 配合物的一维链状图Fig.2 One-dimensional chain structure of the complex
2.2 配合物与DNA的作用
2.2.1 紫外吸收光谱
紫外吸收光谱是研究配合物与DNA作用的常用方法。含杂环配体的配合物在紫外区有一定的电子吸收,当保持配合物的浓度不变,而逐渐增大DNA的浓度后,若配合物的吸光度减少,波长红移,即减色红移效应可认为配合物与DNA发生了插入作用[17-18]。由于插入配体与DNA碱基对发生了π电子堆积,配体的π*空轨道与碱基的π电子轨道发生偶合,使能级下降,导致π-π*跃迁的能量减少,从而发生红移现象;同时,偶合后的π*轨道因部分填充电子,使π-π*跃迁几率减少,产生减色效应[18-19]。减色红移效应与配合物的插入程度也有关系,减色效应越明显,吸收峰红移越大,说明配合物插入程度越强。
在不同浓度的CT-DNA存在时,配合物的紫外吸收光谱如图3。由图3可知,配合物与DNA作用后其吸收光谱随DNA浓度的逐渐增大而发生明显的减色效应,说明配合物与DNA可能发生了插入作用。为定量研究配合物与DNA作用程度的强弱,由方程 cDNA/(εa-εf)=cDNA/(εb-εf)+1/[Kb(εb-εf)]可求得结合常数 Kb[20]。εa为配合物的表观摩尔吸光系数,εb、εf分别为键合和自由配合物的摩尔吸光系数,Kb为配合物与 DNA 的结合常数。将-cDNA/(εa-εf)对 cDNA作图(图3内嵌图),通过斜率与截距之比即可得到结合常数Kb=2.97×104L·mol-1。远小于比以经典插入方式与 DNA 结合的 EB(Kb=3.3×105L·mol-1[21])。 这说明,配合物与DNA以插入方式结合,但插入能力小于EB。

图3 不同浓度CT-DNA存在下配合物的紫外吸收光谱Fig.3 Absorption spectra of the complex in the absence and presence of CT-DNA
2.2.2 荧光光谱
溴化乙锭为具有共轭芳香环的扁平分子,本身荧光极弱,当它专一插入DNA分子的碱基对时,其体系的荧光大为加强[22]。而当其它不产生荧光的分子也可以与DNA发生插入作用时,会与EB竞争DNA碱基对的作用位点,使EB-DNA的荧光强度大为减弱即发生了荧光猝灭,因而EB可作为配合物与DNA作用的结构探针。
由配合物与EB竞争实验的荧光光谱图 (图4)可见,在EB-DNA体系中逐渐增加配合物的浓度,体系的荧光强度明显减弱,可以推断配合物分子可能与CT-DNA发生了插入作用,将EB分子从EBDNA体系中挤出。荧光猝灭行为遵循Stern-Volmer方程[18]。

I0为不加配合物时EB-DNA体系的荧光强度,I为添加不同浓度配合物时配合物与EB-DNA体系的荧光强度,Ksq为猝灭常数,r=cVOL/cDNA。经I0/I vs r作图(内嵌图)可求得Ksq为1.60。此值与以前报道的配合物[VO(Naph-Glu)(Phen)]的Ksq(0.33)[10]相比要大,但比配合物[Cu2(Dmbiim)4(H2O)2]4+的Ksq(2.39)[23]却小。这与这些配合物的结构有关。

图4 配合物对EB-DNA体系荧光光谱的影响Fig.4 Effects of the complex on the fluorescence spectra of EB-DNA system
2.2.3 圆二色(CD)光谱
CT-DNA是手性生物大分子,其旋光性是由于组成核苷酸的戊糖具有不对称碳原子,因此在吸收区域具有圆二色性。CT-DNA的CD光谱中有两个明显的特征峰,273 nm左右的正峰是由CT-DNA碱基对的π-π堆积作用引起的,244 nm左右的负峰则主要是由于DNA的右手螺旋性[24]。当DNA溶液中加入其它小分子后,由于小分子与DNA作用,特别是插入到DNA碱基对时,会影响碱基对之间的作用,使DNA的构象发生变化,从而引起DNA的CD谱发生变化[25]。
配合物与CT-DNA作用的圆二色光谱如图5所示,当相同DNA浓度的溶液中逐渐增加配合物的浓度后,CD光谱正峰的椭圆率逐渐增大,负峰基本不变,说明配合物与DNA发生了插入作用,且主要是影响了DNA碱基对之间的π-π堆积作用。这与紫外吸收光谱和荧光光谱的结论是一致的。
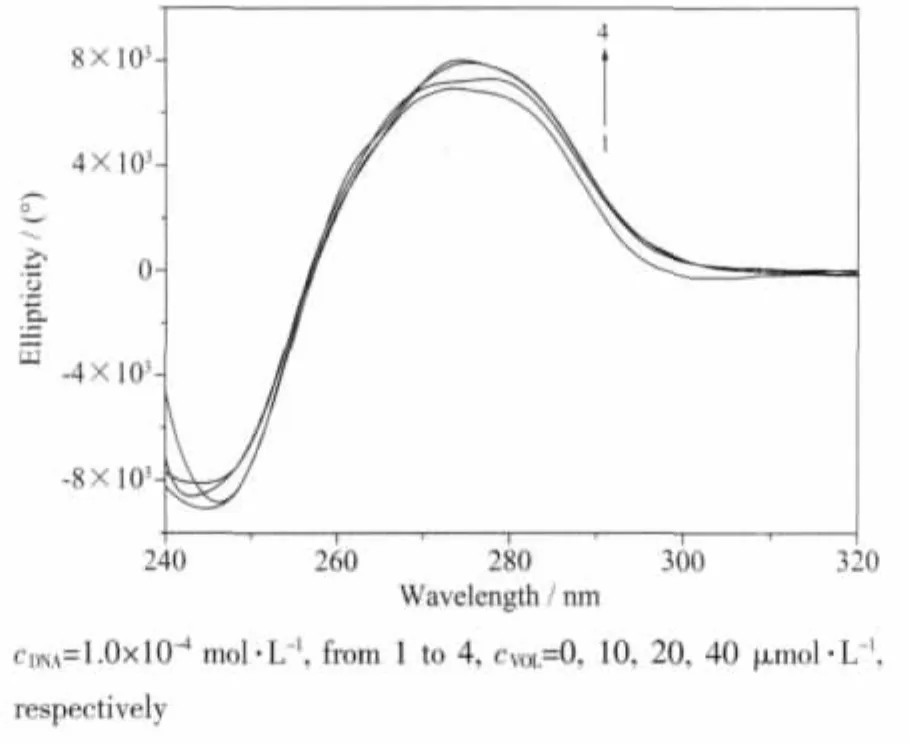
图5 配合物对CT-DNA圆二色光谱的影响Fig.5 Effects of the complex on CD spectra of CT-DNA
2.2.4 粘度测量
在缺少晶体结构证据的情况下,粘度测量是检测溶液状态下配合物与DNA作用模式的重要手段[26]。当配合物以插入模式与DNA作用时,碱基对之间的距离将变大以容纳进入的配体,导致DNA双螺旋伸长,则溶液粘度增加;当配合物以静电或沟面结合等非插入模式与DNA作用时,粘度没有明显的变化;当配合物以部分插入模式与DNA作用时,会造成DNA双螺旋扭结,使DNA粘度减小。图6为配合物和EB对CT-DNA相对粘度的影响曲线。从图6中可以看出,随着配合物浓度的增大,CT-DNA粘度略有增加,说明配合物以插入方式与CT-DNA作用,但比典型的插入剂EB要弱。在配合物中的邻菲咯啉等部分配体的共面性较大,会以插入方式进入到CT-DNA的碱基对中,造成碱基对之间的距离增大使DNA的双螺旋伸长,导致溶液粘度增加。这与光谱法得出的结论一致。

图6 配合物对CT-DNA相对粘度的影响Fig.6 Effects of the complex and EB on the relative viscosity of CT-DNA
2.2.5 琼脂糖凝胶电泳法
以琼脂糖为凝胶的电泳技术已被用于帮助了解小分子金属配合物对DNA的切割作用机理[27]。质粒DNA存在3种构型:共价闭环的超螺旋(FormⅠ)的结构最为紧密,迁移率最大;线型(FormⅢ)其次;而开环缺刻型(FormⅡ)由于结构比较松散,迁移率最小。图7为配合物切割pBR322 DNA的电泳图,由图 7 可见,当配合物浓度为 60 μmol·L-1,质粒DNA中出现了开环缺刻FormⅡ型。并且随着配合物浓度的增大,超螺旋FormⅠ型越来越少,开环缺刻 FormⅡ型逐渐增多。当浓度达到150 μmol·L-1时,超螺旋FormⅠ型完全消失。说明配合物对质粒DNA具有一定的切割活性。

图7 配合物切割pBR322 DNA的电泳图Fig.7 Submarine gel electrophoresis of pBR322 DNA in the presence of the complex
3 结 论
合成了L-苯丙氨酸萘酚醛希夫碱邻菲咯啉三元氧钒配合物,利用元素分析和IR进行了表征并测定了其晶体结构。通过紫外吸收光谱、荧光光谱、圆二色光谱和粘度测量等研究了配合物与CT-DNA的相互作用,结果表明配合物以插入方式与CTDNA作用。利用琼脂糖凝胶电泳初步研究了配合物对pBR322 DNA的切割作用,结果表明配合物可切割超螺旋型DNA为缺刻型DNA。
[1]Friedman A E,Kumar C V,Turro N J,et al.Nucleic Acids Res.,1991,19(10):2595-2602
[2]Chaviara A T,Cox P J,Repana K H,et al.J.Inorg.Biochem.,2005,99(2):467-476
[3]Thompson K H,Orvig C J.Inorg.Biochem.,2006,100:1925-1935
[4]Heyliger C E,Tahiliani A G,McNeil J H.Science,1985,227:1474-1477
[5]ShechterY,Karlish S J.Nature,1980,284:556-558
[6]Thompson K H,McNeill J H,Orvig C.Chem Rev.,1999,99:2561-2572
[7]Nath M,Pokharia S,Yadav R.Coord.Chem.Rev.,2001,215:99-149
[8]Sasmal P K,Patra A K,Chakravarty A R.J.Inorg.Biochem.,2008,102:1463-1472
[9]Kwong D W J,Chan O Y,Shek L K,et al.J.Inorg.Biochem.,2005,99:2062-2073
[10]Li L Z,Guo Z H,Zhang Q F,et al.Inorg.Chem.Commun.,2010,13(10):1166-1169
[11]Xu T,Li L Z,Zhou S F,et al.J.Chem.Crystallogr.,2005,35:263-267
[12]LI Lian-Zhi(李连之),XU Tao(许涛),WANG Da-Qi(王大奇),et al.Chinese J.Inorg.Chem.(Wuji Huaxue Xuebao),2004,20:236-240
[13]LI Lian-Zhi(李连之),XU Tao(许涛),WANG Da-Qi(王大奇),et al.Chinese J.Struc.Chem.(Jiegou Huaxue),2004,23:865-869
[14]Reichmann M E,Rice S A,Thomas C A,et al.J.Am.Chem.Soc.,1954,76:3047-3053
[15]Sheldrick G M.SHELXTL6.10,Bruker Analytical Instrumentation,Madison,Wisconsin,USA,2000.
[16]Kenji K,Makoto T,Ken H,et al.Inorg.Chem.Acta,2000,305:172-183
[17]Tysoe S A,Morgan R J,Baker A D,et al.J.Phys.Chem.,1993,97:1707-1711
[18]Lakowicz J R,Weber G.Biochemistry,1973,12:4161-4170
[19]ZHONG Wen-Tian(钟 文 添 ),WU Jian-Zhong(吴 建 中 ).Chinese J.Inorg.Chem.(Wuji Huaxue Xuebao),2003,19(2):196-200
[20]Wolfe A,Shimer G H Jr,Meehan T.Biochemistry,1987,26:6392-6396
[21]Strothkamp K G,Strothkamp R E.J.Chem.Edu.,1994,71:77-79
[22]WU Jian-Zhong(吴建中),WANG Lei(王雷),YANG Guang(杨光),et al.Chem.J.Chinese University(Gaodeng Xuexiao Huaxue Xuebao),1996,17(7):1010-1015
[23]Li Y P,Wu Y B,Zhao J,et al.J.Inorg.Biochem.,2007,101:283-290
[24]Uma V,Kanthimathi M,Weyhermuller T,et al.J.Inorg.Biochem.,2005,99:2299-2307
[25]Lincoln P,Tuite E,Norden B.J.Am.Chem.Soc.,1997,119:1454-1455
[26]Satyanarayana S,Dabrowiak J C,Chaires J B.Biochemistry,1993,32:2573-2584
[27]Barton J K,Raphael A L.J.Am.Chem.Soc.,1984,106:2466-2468
Synthesis,Crystal Structure and DNA-Binding Studies of an Oxovanadium髧Complex[VO(Naph-Phe)(Phen)]with Mixed-Ligands
BIAN Lin LI Lian-Zhi*WANG Xia HUANG LeiPU Xue-WeiDONG Jian-Fang
(School of Chemistry and Chemical Engineering,Liaocheng University,Liaocheng,Shandong 252059,China)
A new oxovanadium complex with mixed-ligands of a Schiff base(Naph-Phe)derived from L-phenylalanine and 2-hydroxy-1-naphthaldehyde and 1,10-phenanthroline(Phen),[VO(Naph-Phe)(Phen)],has been synthesized and characterized by elementary analysis,IR and single crystal X-ray diffraction.It was shown that the complex crystallized in monoclinic crystal system,P21/c space group with the cell parameters:a=1.02785(11)nm,b=2.186 5(3)nm,c=1.171 150(13)nm,β=94.747 0(10)°,V=2.622 9(4)nm3,Z=4,F(000)=1 164,R1=0.076 7,wR2=0.165 5,S=1.092.The DNA-binding properties have been investigated by UV absorption,fluorescence,CD spectra and viscosity measurement.The results indicate that the complex binds to CT-DNA in an intercalative mode.Meanwhile,the cleavage reaction on plasmid DNA has been monitored by submarine gel electrophoresis.The complex could cleave circular plasmid pBR322 DNA to nicked form.CCDC:770157.
vanadium complex;Schiff base;crystal structure;DNA
O614.51+1
:A
:1001-4861(2011)04-0649-06
2010-09-21。收修改稿日期:2010-12-03。
山东省自然科学基金(No.Y2004B02)资助项目。
*通讯联系人。 E-mail:lilianzhi1963@163.com

