杨树基因组SSR标记在康定柳中的通用性分析
王红莉,蒙嘉文,罗建勋* ,王朱涛,蔡应君,任树平
(1.四川省林科院,四川 成都 610081;2.四川农业大学林学院,四川 雅安 625014;3.若尔盖县林业局,四川 若尔盖 624500)
康定柳(Salix ParaplesiaSchneid)是杨柳科(Salicaceae)柳属(Salix)落叶小乔木或灌木,为川西北高原特有乡土树种。近年来广泛用于该地区防沙治沙,该树种为雌雄异株,天然更新容易,群体内雌雄个体数量相当。为选出优良防沙治沙种植材料,冯毅等[1]分别了研究5个群体遗传多样性。
简单重复序列(SSR,Simple Sequence Repeat)也称微卫星DNA(Microsatellite DNA)或短串联重复(Short Tandem Repeat,STR),是近年来发展起来的基于PCR技术的一种新的分子标记技术,同时,作为一种基于DNA长度多态性的分子标记技术。SSR由2~4个多次串联重复的核苷酸组成,广泛分布于真核生物的基因组中[2]。SSR分子标记因具有共显性遗传、高度多态性、重复性和稳定性好、易于实现自动化等优点[3]。在遗传和物理图谱构建、基因定位、遗传多样性和物种进化分析、亲缘关系鉴定、DNA指纹图谱构建、比较基因组学、分子标记辅助选择育种等方面有着广泛的应用前景[4~6]。
SSR引物开发工作量大,操作复杂,并且需要了解一定的基因组序列信息,有研究表明:从一个基因组中分离出来的SSRs可以用于同科、同属中其它个体[7,8]。这样一来,不仅大大降低了开发成本,并且也使得SSR技术在种质资源的研究方面得到更广泛的应用。本实验以川西高原地区特有的乡土树种——康定柳为材料,在杨树基因组SSR标记中筛选引物,分析其引物通用性,为下一步在DNA分子水平上通过SSR分子标记探求康定柳雌雄群体间/内遗传多样性、遗传结构和遗传分化,为生产上使用雌雄群体或混合群体防沙治沙遗传稳定性和成效提供理论依据。
1 材料与方法
1.1 实验材料
本研究所采用的康定柳的5个群体分别来自四川省的红原、若尔盖红星、阿坝、巴西和壤塘。每个群体采集雌雄单株各20株,共计200个个体。
本实验采用的622对SSR引物来源于杨树SSR数据库Populusmolecular genetics cooperative(http://www.ornl.gov/sci/ipgc/ssr_resource.htm)随机抽取合成。
1.2 实验方法
1.2.1 康定柳基因组DNA的提取
采取CTAB法提取康定柳新鲜叶片的基因组DNA[9]。将其浓度最终定为 50 ng·μl-1~ 100 ng·μl-1。
1.2.2 PCR 反应及产物检测
从5个群体中随机选取两个个体,用622对引物分别进行PCR扩增。
在以上能扩增出条带的引物的基础上,分别从5个群体的雌雄群体中各随机抽取一个样,共计10个个体,进一步进行PCR扩增。
PCR反应体系为:20 μl体系,DNA模板 0.5 μl,rTaqDNA 聚合酶 0.2 μl,10 × PCR buffer 2.0 μl,dNTPs 0.8 μl,上下游引物各 1.0 ul,灭菌蒸馏水14.5 μl。
PCR扩增程序:95℃预变性3 min;30个循环(95℃ 变性 30 s,57℃ 退火 30 s,72℃ 延伸 30 s);72℃延伸10 min;4℃保存。
PCR扩增产物用1.5%的琼脂糖凝胶电泳检测,溴化乙锭(EB)染色后对扩增结果进行记录。
2 结果与分析
2.1 初筛
从5个群体中随机选取两个个体(来自不同群体的雌雄株),用622对引物分别进行PCR扩增。
622对引物对随机选取的引物扩增情况见表1。

表1 初筛引物统计表
其中两对样品都能扩增出来的有105对,占实验总数的16.88%。
初步筛选中,引物可利用率达到16.88%。部分引物扩增结果见图1。

图1 两个样品部分引物扩增结果
2.2 复筛
在以上能扩增出条带的引物的基础上,分别从5个群体的雌雄群体中各随机抽取一个样,减去初筛时取过的2个群体的个体,共计8个个体,进一步进行PCR扩增。
上一步中筛选出的105对引物对随机选取的8个个体扩增结果见表2。
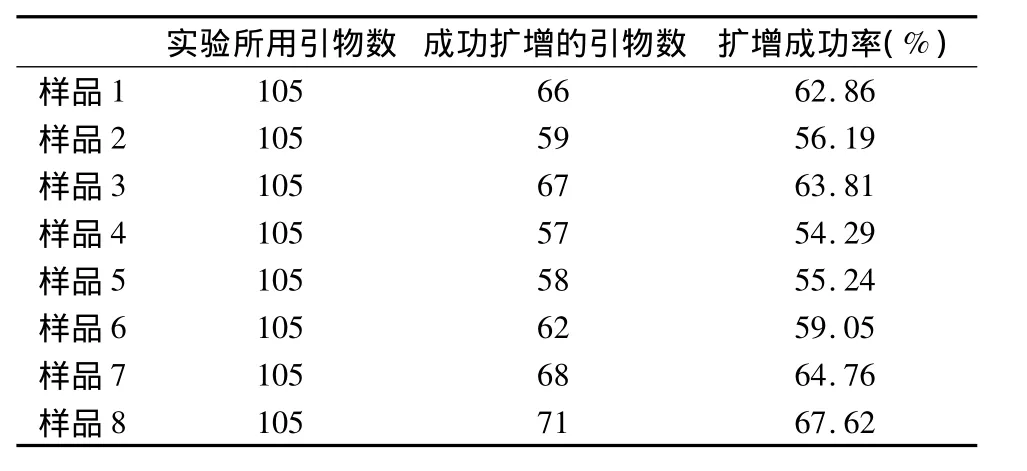
表2 复筛引物统计表
其中8个个体都能扩增出清晰条带的有49对引物,占复筛引物总数的46.67%。
复筛选中,引物可利用率达到7.88%。部分引物扩增结果见图2。
3 结论

图2 8个样品部分引物扩增结果
从来自杨树基因组的622对引物对与其同科不同属的康定柳的扩增结果来看,引物的扩增成功率为7.88%。虽然概率比较低,但是最终得到的49对引物对所有代表性的样品都可以稳定地扩增出清晰的条带,因而其引物可以在该物种的SSR分析中通用。
通用性引物的存在能够有效地弥补物种分子标记的不足,丰富标记数量[10]。本研究在杨柳科中的康定柳这一川西高原特有的柳树品种中证实了杨树基因组SSR标记在该树种中的通用性。这对进一步从分子水平上研究康定柳的遗传多样性、遗传结构、遗传分化和构建遗传图谱等具有重要的学术和应用价值。
[1] 冯毅,罗建勋,等.川西北地区康定柳天然群体表型多样性研究[J]。西南林学院学报,2010,30(4):11 ~15.
[2] Litt M,and Luty J A.A hypervariable microsatellite revealed byin vitroamplification of a dinucleotide repeat within the cardiac muscle actin gene,Am J Hum Genet,1989,44:397 ~401.
[3] Powell W,Machray G C,and Provan J.Polymorphism revealed by simple sequence repeats.Trends in Plant Science,1996,1(7):215~222.
[4] Tuskan G A,Gunter L E,Yang Z K,et al.Characterization of microsatellites revealed by genomic squencin ofPopulus trichocarpa,Can.J.For.Res.,2004,34:85 ~93.
[5] Varshney R K,Graner A,and Sorrells M E.Genic microsatellite markers in plants:features and applications.T rends in Biotechnology,2005,23(1):48 ~55.
[6] Yin T M,DiFazio S P,Gunter L E,et al.Genome structure and emerging evidence of an incipient sex chromosome inPopulus.Genome Res,2008,18:422 ~430.
[7] Kijas J M H,Fowler J C S,Thomas M R.An evaluation of sequence tagged microsatellite site marker for genetic analysis withinCitrusand related species.Genome,1995,38:349 ~355.
[8] Peakall R,Gilmore S,Keys W,Morgante M,Rafalski A.Cross-species amplification of soybean(Glycine max)simple sequence repeats(SSRs)within the genus and other legume genera.Mol.Biol.Evol,1998,15:1275 ~1287.
[9] Doyle J J,and Doyle J L.A rapid DNA isolation procedure for small quantities of fresh leaf tissue,Phytochemistry Bulletin,1987,19:11~15.
[10] 李宏伟,刘曙东,高丽锋,等.小麦EST-SSRs的通用性研究[J].植物遗传资源学报,2003,4(3):252~255.

