花色苷生物代谢途径中相关酶的研究进展
刘 飞,郭美丽(第二军医大学药学院生药学教研室,上海 200433)
花色苷是植物体内一类重要的次生代谢产物,主要是花青素的糖苷类化合物。植物之所以呈现出各种颜色,与花色苷有着重要的关系[1]。在植物次生代谢途径中,花色苷代谢途径可以算是研究较深入的一个。再加上近几十年来,突变体、重组DNA、差示扫描量热法、遗传位点标记、蛋白质纯化、PCR扩增等新技术和新方法的应用,大大推动了花色苷代谢途径中相关酶及其编码基因的研究。
1 花色苷相关药学研究
花色苷类化合物易溶于极性溶剂,因此常用含有少量盐酸或甲酸的甲醇溶液提取植物体内的花色苷。高等植物体内主要存在以下6种常见的花色苷类化合物:天竺葵色素(pelargonidin)、芍药色素(peonidin)、矢车菊色素(cyanidin)、锦葵色素(malvidin)、矮牵牛花色素(petunidin)和飞燕草色素(delphinidin)。花色苷的化学结构见表1。毛细管区带电泳、核磁共振、基质辅助激光解吸质谱(MALDI-MS)等新的药物分析方法,使得花色苷的分离和纯化效果得到了很大提高。关于花色苷的药理作用研究的报道很多,但主要集中在抗肿瘤、抗心血管病变和抗诱变等方面。Sarma等研究发现,小
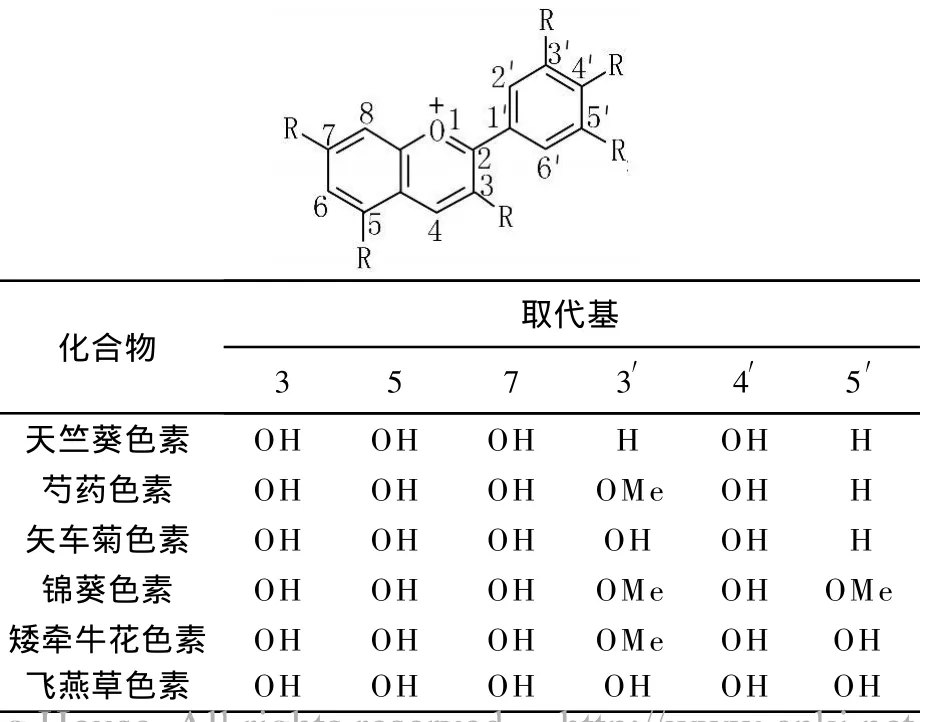
表1 高等植物中常见的6种花色素Table 1 Six common anthocyanidins in high plants
牛胸腺DNA与矢车菊色素形成的复合体能有效地防止羟基自由基(OH◦)对矢车菊色素和DNA的损伤。该研究对花色苷在机体内具有抗氧化活性提供了一种解释。Dai等[2]对含有花色苷的黑莓提取物在提取方法、稳定性、抗氧化活性、抗肿瘤活性及其机制等进行了系统的研究,发现黑莓提取物是很具有潜力的植物药。
2 花色苷生物代谢途径
2.1 代谢途径中的主要作用酶 花色苷生物代谢途径见图1,该途径涉及以下各种酶。
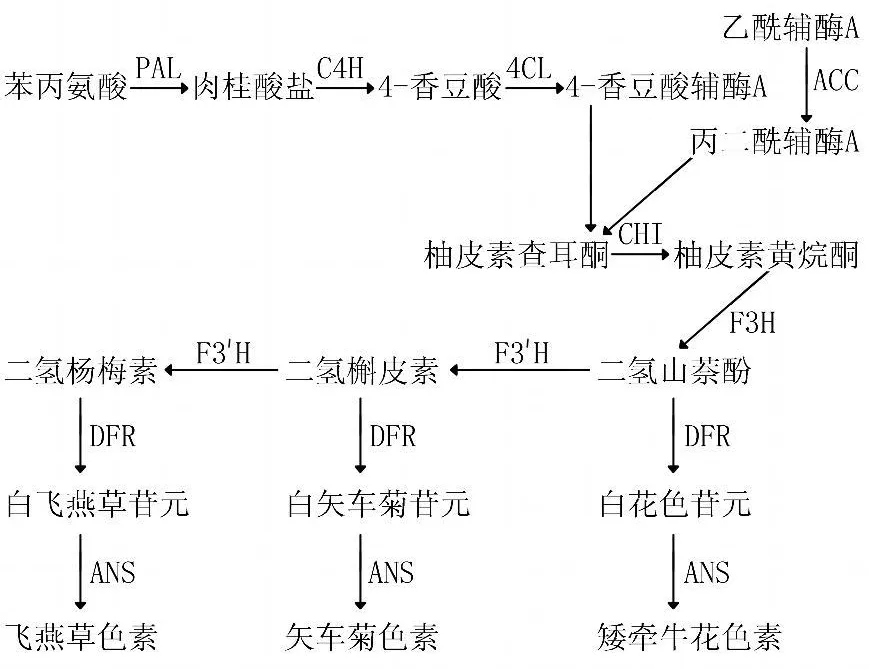
图1 花色苷生物代谢途径Figure 1 The pathway of anthocyanin biometabolism
2.1.1 乙酰辅酶A羧化酶(acetyl-CoA carboxylase,ACC) ACC催化乙酰辅酶A羧化成丙二酸单酰辅酶A。在植物体内,ACC除了在脂肪酸代谢中有显著作用外,还参与了包括黄酮类化合物合成在内的各种代谢途径。Egli等[3]在玉米中分离得到了两种ACC,分别为227 U的ACCaseⅠ和219 U的ACCaseⅡ。Shorrosh等[4]通过合成引物对cDNA的延伸和扩增,获得了紫花苜蓿中ACC的核酸全长序列。
2.1.2 苯丙氨酸氨裂解酶(phenylalanine ammonia-lyase,PAL) PAL催化L-苯丙氨酸生成肉桂酸。Olsen等[5]分析了处于莲座期的拟南芥叶子在不同环境下4种PAL基因的表达,发现在环境因素触发下黄酮类生物合成途径中PAL1和PAL2有功能特异性。
2.1.3 肉桂酸-4-羟化酶(cinnamate-4-hydroxylase,C4H) C4H 催化反式-肉桂酸生成反式-4-香豆素。Ro等[6]在一种杂交白杨内分离出了编码C4H的cDNA,并将其导入到拟南芥中,通过分析找到了C4H的亚细胞定位。Achnine等[7]通过对C4H和PAL两个在代谢途径相承的酶的定位研究发现,两者有共区域化特点。Chen等[8]报道了对C4H的催化底物种类,并对相关的动力学进行了研究。
2.1.44 -香豆素辅酶A连接酶(4-coumarate CoA ligase,4CL)4CL催化4-香豆素生成4-香豆素辅酶A。Schneider等[9]找出了拟南芥的4CL的2号异构体(At4CL2)的底物结合域(SBP),并对其进行了功能分析。Hamberger等[10]对At4CL基因中的四个基因进行分析,并与其他种类植物的4CL基因进行了比较,把At4CL基因家庭从相似基因中界定出来。
2.1.5 查尔酮合酶(chalconesynthase,CHS) Kreuzaler等通过UV诱导荷兰芹悬浮培养细胞高表达出了CHS基因。Reimold等[11]克隆出了CHS的全长基因,并分析了其表达的氨基酸序列。Ferrer等[12]通过对查尔酮以及和底物结合时的晶体结构进行分析,发现了CHS的活性中心。
2.1.6 查尔酮异构酶(chalcone isomerase,CHI) Boland等[13]从大豆种子中提纯了CHI并对其理化性质做了研究。Mehdy等[14]从大豆(Phaseolus vulgaris L.)中找到了CHI的mRNA,并比较了在不同环境因素的刺激下其表达量,得出了CHI由单基因控制并受多种环境因素影响的结论。Druka等[15]利用突变技术比较了大米(Oryza sativa)和大麦(Hordeum vulgare)的CHI的物理图谱、遗传图谱和突变图谱。
2.1.7 黄烷酮-3β-羟化酶(flavanone-3β-hydroxylase,F3H) F3H是一种非血红素铁酶,依赖于Fe2+、分子氧、2-酮戊二酸和抗坏血酸而起作用。Lukacin 等[16]研究发现 ,His220、His278 和 Asp222位于F3H的离子结合区域,Arg288位于2-酮戊二酸区域。之后,他们又从矮牵牛(Petunia hybrida)中分离纯化重组的F3H,并找出了Glu337-Leu338部分为该酶水解作用部位[17]。Owens等[18]完整地描述了拟南芥F3H(AtF3H)的生化特性,并通过等位基因的研究发现,在植物体内,黄酮醇合酶(flavonol synthase,FLS)和花青素合酶(anthocyanidin synthase,ANS)能在一定程度上补偿F3H的功能。
2.1.8 二氢黄酮醇-4-还原酶(dihydroflavonol-4-reductase,DFR) Lo Piero等[19]利用突变体对比研究发现,编码DFR的基因d f r是单克隆基因,在不同突变体中都存在,而不同的突变体有不同的颜色与d f r表达量有关。Petit等[20]通过大肠杆菌表达了葡萄(Vitis vinif era)的DFR并获得了晶体,随后对其结构和理化性质进行了研究。
2.1.9 花青素合酶 ANS又名百花色素双加氧酶(leucoanthocyanidin dioxygenase,LDOX),是一种2-酮戊二酸依赖性酶。Pelletier等[21]在拟南芥中得到一种表达序列标签(EST YAY780)并分析了其序列,发现这个EST编码的蛋白质序列与ANS序列有同源性。Shimada等[22]在石竹目的菠菜(Spinacia oleracea)中发现了与ANS功能很相似的酶并通过基因表达谱指出:只有在菠菜种子中,ANS和DFR对原花色素的合成有作用。因此他们提出了在石竹目中花青素含量较少的原因是编码ANS和DFR的基因表达受到抑制的假说。
2.1.10 类黄酮-3′-羟化酶(flavonoid-3′-hydroxylase,F3′H) F3′H 和 类 黄 酮-3′,5′-羟 化 酶(flavonoid-3′,5′-hydroxylase,F3′5′H)都属于细胞色素P450酶,分别在黄酮类化合物B环的3′或者3′和5′位置引入羟基。这两个酶在花色苷代谢途径中能催化二氢黄酮醇(DHK、DHQ和DHM)的相互转化。Seitz等[23]将紫菀科内的7个编码 F3′5′H的cDNA导入到酵母中,得到重组的蛋白质。通过对这些蛋白质的活性分析发现,紫菀科的F3′5′H是单独进化的。Seitz等[24]利用重组基因发现,F3′H和F3′5′H 的底物特异性主要由近 N 端区域决定,而两者在功能上的不同主要是因为近C端的不同。他们进一步研究发现,在487位上由Thr到Ser的保守转变,导致了重组的非洲菊(Gerbera hybrida)F3′H 有了5′-羟基化活性。相反地,当由Ser转变成 Thr时,重组的F3′5′H转变成了具有少量活性的F3′H 。
2.1.11 花色苷酰基转移酶(anthocyanin acyltransferases,AATs) 根据乙酰基的供体不同,AAT s可分为两大类:脂肪族类和芳香族类。Nakayama等[25]通 过 对 Ss5MaT1、Ss5MaT2、Dv3MaT、Sc3MaT、Pf5MaT、Gt5AT 和 Pf3AT 的氨基酸对比分析发现,AATs的序列中含有基序1(-His-Xaa3-Asp-)和基序 3(-Asp-Phe-Gly-Trp-Gly-)。通过将Dv3MaT的cDNA引入到矮牵牛中,Suzuki 等[26]发现 ,6′-O-丙二酰化的 cyanidin 3-O-glucoside能有效地防止β-糖基化,这说明丙二酰化是花色素保持稳定的一种方式。
2.1.12 花色苷甲基转移酶(anthocyanin methyltransferases) 甲基化在花色苷代谢途径中研究得不多。花色苷中,只有矢车菊色素和飞燕草色素存在甲基化反应。甲基化主要能让花色苷更加稳定地存在。Jonsson等[27]在矮牵牛中发现基因Mr1、Mt2、Mf 1和M f2分别控制4种甲基转移酶的合成。
2.1.13 花色苷糖基转移酶(anthocyanin glycosyltransferases) 在花色苷糖基化修饰中,主要有F3GT(UDP-Glc:anthocyanidin 3-O-glucosyltransferase orUDP-Glc:flavonoid 3-O-glucosyltransferase),A5GT(UDP-Glc:anthocyanin 5-O-glucosyltransferased),A3′GT(UDP-glucose:anthocyanin 3′-O-glucosyltransferase),A3RT(UDP-rhamnose:anthocyanidin 3-O-glucoside-O-rhamnosyltransferase)和 A3′5′GT(UDP-glucose:anthocyanin 3′,5′-O-glucosyltransferase)等糖基转移酶起作用。Gerats等[28]通过杂交的方法,研究了矮牵牛中基因An1、An2、和 An4对F3GT酶活性的影响。Ford等[29]分离出了一种葡萄(Vitis vini f era)的F3GT。酶代谢动力分析的结果显示,该酶催化矢车菊色素的kcat是其催化黄酮醇槲皮素的48倍。Yamazaki等[30]利用差显技术克隆出了紫苏(Perilla f rutescens)的A5GT的cDNA,并验证了其功能。Fukuchi-Mizutani[31]从三花龙胆(Gentiana tri f lora)中提纯了A3′GT,并根据其氨基酸残基分离出了cDNA。通过对A3′GT的性质研究发现,该酶有严格的底物特异性,只能对与飞燕草色素有相似结构、在3位和5位上有Glc基团的花色苷的3′位的羟基进行糖基化。A3RT将3-花色素糖苷转化成3-花色素芦丁苷。Kogawa等[32]在蝶豆(Clitoria ternatea)中分离提纯了A3′5′GT,该酶主要在飞燕草苷元-3-O-(6″-O-丙二酰)-β-葡萄糖苷 3′位和 5′位葡萄糖苷化的过程中起作用。
2.2 酶的亚细胞定位和酶复合物的研究 早在1978年就有关于花色苷代谢途径中相关酶的亚细胞定位的研究。Hrazdina等[33]对朱顶红属(Hippeastrum)和郁金香属(Tulipa)植物原生质体的各亚细胞组分中二氢黄酮合酶、CHI和A3GT三种酶的活性进行了研究。结果显示,在细胞质中这三种酶的活性最高。Wurtele等[34]发现葡萄糖基转移酶在高粱(Sorghum bicolor)种子的表皮细胞中的活性与质体有关。关于花色苷途径的上游阶段起作用的酶的亚细胞定位也有不少研究。PAL主要存在于细胞壁内,C4H则能在叶绿体和细胞核内找到,4CL主要存在于二次增厚的细胞壁和中果皮维管束组织的薄壁细胞中[35]。在研究代谢酶的亚细胞定位过程中发现,这些酶往往是以酶复合物的形式起催化作用 在1985,关 丙醇和黄酮类化合物代谢途径中的酶是以酶复合物形式起作用的观点就被提出来了。Burbulis等[36]发现CHS、CHI和DFR相互作用有方向依赖性,并通过亲和色谱和免疫共沉淀法在拟南芥种子中进一步确证了它们之间的相互作用。
3 总 结
作为一类很有潜力的药用天然化合物,花色苷以其分布广、含量高、活性强、作用多样性的特点而受到越来越多的关注,对花色苷的研究也不断得到可喜的进展,特别是在其生物代谢过程的研究中不断有新发现。对代谢途径中主要代谢酶的研究,已获得大量的文献资料。而对花色苷代谢下游途径的研究,特别是一些修饰酶的研究则相对较少。目前,关于花色苷的临床作用的研究对象往往是一些富含花色苷的植物提取物,并非单体化合物。而通过花色苷代谢途径的研究就有可能有目的地改变某一单体化合物的含量,从而为单体化合物的研究打下基础。随着研究方法的改进,一些新的代谢途径可能会被发现,一些新的、高药效并且能稳定存在于人体内的花色苷类化合物可能会被发掘出来。
[1] Winkel-Shirley B.Flavonoid biometabolism.A colorful model for genetics,biochemistry,cell biology,and biotechnology[J].Plant Phy siol,2001,126(2):485-493.
[2] Dai J,Gupte A,Gates L,et al.A comprehensive study of anthocyanin-containing ex tracts from selected blackberry cultivars:ex traction methods,stability,anticancer properties and mechanisms[J].Food Chem Toxicol,2009,47(4):837-847.
[3] Egli M A,Gengenbach B G,G ronwald J W,et al.Characterization of maize acetyl-coenzyme A carboxylase[J].Plant Phy siol,1993,101(2):499-506.
[4] Shorrosh B S,Dix on R A,Ohlrogge J B.Molecular cloning,characterization,and elicitation of acetyl-CoA carboxy lase from alfalfa[J].Proc Natl Acad Sci USA,1994,91(10):4323-4327.
[5] Olsen K M,Lea U S,Slimestad R,et al.Differential expression of four Arabidopsis PAL genes;PAL1 and PAL2 have functional specialization in abiotic environmental-triggered flavonoid sy nthesis[J].J Plant Physiol,2008,165(14):1491-1499.
[6] Ro D K,Mah N,Ellis B E,et al.Functional characterization and subcellular localization of poplar(Populus trichocarpa×Populus deltoides)cinnamate4-hy droxylase[J].Plant Physiol,2001,126(1):317-329.
[7] Achnine L,Blancaflor E B,Rasmussen S,et al.Colocalization of L-phenylalanine ammonia-lyase and cinnamate4-hydroxylase for metabolic channeling in phenylpropanoid biometabolism[J].Plant Cell,2004,16(11):3098-3109.
[8] Chen Hao,Jiang HanXiao,Morgan J A.Non-natural cinnamic acid derivatives as substrates of cinnamate4-hydroxylase[J].Phytochemistry,2007,68(3):306-311.
[9] Schneider K,Hö vel K,Witzel K,et al.The substrate specificity-determining amino acid code of4-coumarate:CoA ligase[J].Proc Natl Acad Sci USA,2003,100(14):8601-8606.
[10] Hamberger B,Hahlbrock K.T he4-coumarate:CoA ligase gene family in Arabidopsis thaliana comprises one rare,sinapate-activating and three commonly occurring isoenzy mes[J].Proc Natl Acad Sci USA,2004,101(7):2209-2214.
[11] Reimold U,Kröger M,Kreuzaler F,et al.Coding and 3′noncoding nucleotide sequence of chalcone sy nthase mRNA and assignment of amino acid sequence of the enzyme[J].EMBO J,1983,2(10):1801-1805.
[12] Ferrer J L,Jez J M,Bowman M E,et al.Structure of chalcone synthase and the molecular basis of plant polyketide biometabolism[J].Nat Struct Biol,1999,6(8):775-784.
[13] Boland M J,Wong E.Purification and kinetic properties of chalcone-flavanone isomerase from soya bean[J].Eur J Biochem,1975,50(2):383-389.
[14] Mehdy M C,Lamb C J.Chalcone isomerase cDNA cloning and mRNA induction by fungal elicitor,wounding and infection[J].EM BO J,1987,6(6):1527-1533.
[15] Druka A,Kudrna D,Rostoks N,et al.Chalcone isomerase gene from rice(Oryza sativa)and barley(Hordeum vulgare):physical,genetic and mutation mapping[J].Gene,2003,302(1-2):171-178.
[16] Lukacin R,Britsch L.Identification of strictly conserved histidine and arginine residues as part of the active site in Petunia hybrida flavanone 3β-hydroxylase[J].Eur J Biochem,1997,249(3):748-757.
[17] Lukacin R,G röning I,Schiltz E,et al.Purification of recombinant flavanone 3β-hydroxylase from Petunia hybrida and assignment of the primary site of proteolytic degradation[J].Arch Biochem Biophys,2000,375(2):364-370.
[18] Owens D K,Crosby K C,Runac J,et al.Biochemical and genetic characterization of Arabidopsis flavanone 3β-hydroxylase[J].Plant Physiol Biochem,2008,46(10):833-843.
[19] Lo Piero A R,Pug lisi I,Petrone G.Gene characterization,analy sis of expression and in vitro synthesis of dihydroflavonol4-reductase from Citrus sinensis(L.)Osbeck[J].Phytochemistry,2006,67(7):684-695.
[20] Petit P,Granier T,d′Estaintot B L,et al.Crystal structure of grape dihydroflavonol4-reductase,a key enzyme in flavonoid biometabolism[J].J Mol Biol,2007,368(5):1345-1357.
[21] Pelletier M K,Murrell J R,Shirley B W,Characterization of flavonol synthase and leucoanthocyanidin dioxy genase genes in Arabidopsis.Further evidence fo r differential regulation of“early” and “late” genes[J].Plant Physiol,1997,113(4):1437-1445.
[22] Shimada S,Inoue Y T,Sakuta M.Anthocyanidin synthase in non-anthocyanin-producing Caryophy llales species[J].Plant J,2005,44(6):950-959.
[23] Seitz C,Eder C,Deiml B,etal.Cloning,functional identifica-tion and sequence analy sis of flavonoid 3′-hydroxylase and flavonoid 3′,5′-hydroxylase cDNAs reveals independent evolution of flavonoid 3′,5′-hydroxylase in the Asteraceae family[J].Plant Mol Biol,2006,61(3):365-381.
[24] Seitz C,Ameres S,Forkmann G.Identification of the molecular basis for the functional difference between flavonoid 3′-hydroxylase and flavonoid 3′,5′-hydroxylase[J].FEBS Lett,2007,581(18):3429-3434.
[25] Nakayama T,Suzuki H,Nishino T.Anthocyanin acyltransferases:specificities,mechanism,phylogenetics,and applications[J].J M ol Cataly sis B:Enzymatic,2003,23(2-6):117-132.
[26] Suzuki H,Nakayama T,Yonekura-Sakakibara K,et al.cDNA cloning,heterolog ous expressions,and functional characterization of malonyl-coenzyme A:anthocy anidin 3-O-glucoside-6″-O-malonyltransferase from dahlia flowers[J].Plant Physiol,2002,130(4):2142-2151.
[27] Jonsson L M V,Aarsman M E G,Poulton J E,et al.Properties and genetic control of four methyltransferases involved in methylation of anthocyanins in flowers of Petunia hybrida[J].Planta,1984,160(2):174-179.
[28] Gerats A G M,Vrijlandt E,Wallroth M,et al.T he influence of the genes An1,An2,and An4 on the activity of the enzyme UDP-g lucose:flavonoid 3-O-glucosyltransferase in flowers of Petunia hybrida[J].Biochem Genet,1985,23(7-8):591-598.
[29] Ford C M,Boss P K,Hφ j P B.Cloning and characterization of Vitis vinif era UDP-glucose:flavonoid 3-O-glucosyltransferase,a homologue of the enzyme encoded by the maize Bronze-1 locus that may primarily serve to glucosylate anthocyanidins in vivo[J].J Biol Chem,1998,273(15):9224-9233.
[30] Yamazaki M,G ong ZhiZhong,Fukuchi-Mizutani M,et al.M olecular cloning and biochemical characterization of a novel anthocyanin 5-O-glucosyltransferase by mRNA differential display for plant forms regarding anthocyanin[J].J Biol Chem,1999,274(11):7405-7411.
[31] Fukuchi-Mizutani M,Okuhara H,Fukui Y,et al.Biochemical and molecular characterization of a novel UDP-glucose:anthocyanin 3′-O-glucosyltransferase,a key enzyme for blue anthocyanin biometabolism,from gentian[J].Plant Phy siol,2003,132(3):1652-1663.
[32] Kogawa K,Kato N,Kazuma K,et al.Purification and characterization of UDP-glucose:anthocyanin 3′,5′-O-glucosyltransferase from Clitoria ternatea[J].Planta,2007,226(6):1501-1509.
[33] Hrazdina G,Wagner G J,Siegelman H W.Subcellular localization of enzymes of anthocyanin biometabolism in protoplasts[J].Phytochemistry,1978,17(1):53-56.
[34] Wurtele E S,Thayer S S,Conn E E.Subcellular localization of a UDP-glucose:aldehyde cyanohydrin β-glucosyl transferase in epidermal plastids of Sorghum leaf blades[J].Plant Physiol,1982,70(6):1732-1737.
[35] Chen JianYe,Wen PengFei,Kong WeiFu,et al.Changes and subcellular localizations of the enzymes involved in phenylpropanoid metabolism during grape berry development[J].J Plant Physiol,2006,163(2):115-127.
[36] Burbulis L E,Winkel-Shirley B.Interactions among enzy mes of the Arabidopsis flavonoid biosy nthetic pathway[J].Proc Natl Acad Sci USA,1999,96(22):12929-12934.
——矮牵牛

