白花树SRAP—PCR反应体系的建立与优化
柳新红,李 楠,李因刚,徐 梁,盛炜彤,惠刚盈,郑勇奇
(1. 中国林业科学研究院林业研究所,北京 100091;2. 浙江省林业科学研究院,浙江 杭州 310023)
白花树SRAP—PCR反应体系的建立与优化
柳新红1,2,李 楠2,李因刚2,徐 梁2,盛炜彤1,惠刚盈1,郑勇奇1
(1. 中国林业科学研究院林业研究所,北京 100091;2. 浙江省林业科学研究院,浙江 杭州 310023)
以白花树叶片的总DNA为模板,通过正交试验设计,确定了适合白花树的SRAP-PCR的20 µL最佳反应体系为:24 ng模板DNA,2.0 mM的Mg2+,0.5 mM的dNTPs,1.25uM的引物,1.5 U的Taq DNA聚合酶,2 µL的10× PCR缓冲液。经琼脂糖凝胶电泳检测,特征性条带清晰,信息量大,稳定。
白花树;SRAP-PCR;反应体系;正交设计
白花树(Styrax tonkinensis)又名越南安息香、滇桂野茉莉、白背安息香,属安息香科安息香属落叶乔木,是热带和亚热带树种,分布于我国的云南、广东、广西、贵州、福建、湖南、江西、重庆等省(市、区)以及越南、老挝等国[1]。该树种生长迅速,成材期短,萌芽力强[2],是制浆、造纸等短周期工业原料林的首选树种之一;同时该树种结果量大,早实丰产,种子含油率高,也是材、油兼用的优良生物质能源树种。此外,白花树树脂“安息香”,含较多香脂酸,是贵重药材[3],有防腐、消炎、祛痰、行气血之效,并可制造高级香料。
相关序列扩增多态性(Sequence-related amplified polymorphism,SRAP)标记是基于聚合酶链式反应(PCR)的新型分子标记技术[4]。该标记根据基因外显子中GC含量丰富的内含子、启动子中AT含量丰富的特点,设计一对适宜引物进行PCR扩增,因不同个体的内含子、启动子与间隔区的长度差异而产生多态性。SRAP标记与RFLP(Restriction Fragment Length Polymorphism)、RAPD(Random Amplified Polymorphic DNA)、ISSR(Inter-Simple Sequence Repeat)、AFLP(Amplified Fragment Length Polymorphism)、SSR(Simple Sequence Repeat)等分子标记技术相比,具有技术简单、快速、引物设计简单、多态性和信息量丰富、在基因组中分布均匀、便于克隆测序目标片段、实验成本低等优点,尤其是与ISSR、RAPD等以随机引物为基础的分子标记相比,SRAP可检测基因的可译框或调控区域,扩增结果可能与基因的编码区域或调控区域直接相关,因而获得的差异扩增DNA条带将直接与基因关联,从而将个体的性状差异与特异基因连锁[5~10],在遗传学和育种学领域具有广泛的应用前景。
目前,还没有分子标记技术在白花树上应用的研究报道,相对阻碍了白花树遗传多样性、遗传图谱构建和基因定位等方面的研究。因此,本研究拟先通过优化白花树的SRAP-PCR反应体系,为进一步开展白花树分子遗传学与标记辅助育种等研究奠定基础,以期推动这一优良树种的研究利用。
1 材料与方法
1.1 实验材料
实验材料采集于白花树分布相对集中的云南、广西、广东、福建、江西、湖南、重庆和贵州8个省(区、市)和越南等地,共20份种质,296份材料。
试验所用Taq DNA聚合酶、10×PCR buffer、Mg2+、dNTPs均购自TaKaRa公司;SRAP引物采用的组合[4,11~12]见表1、表2,由上海生工生物工程技术服务有限公司合成。

表1 SRAP正向引物及其序列Table 1 Sequcnccs(5'-3’)of SRAP forward primers used in this research

表2 SRAP反向引物及其序列Table 2 Sequences(5’-3’)of SRAP reverse primers used in this research
1.2 实验方法
1.2.1 白花树总DNA的提取 采用CTAB 法提取白花树叶片的总DNA,DNA粗提物再经Magabio核酸纯化试剂盒(Bioer,杭州)进行纯化。纯化后的DNA样品分别用1.5%的琼脂糖凝胶电泳和DNA/RNA紫外分光光度计(GeneQuant Pro,GE Healthcare)进行定性和定量检测。
1.2.2 引物筛选 SRAP的反应程序参考Li和Quiros等的方法[13],并在此基础上有所改进,将35个循环中的退火温度由50℃提高到52℃,进行引物的初步筛选,所得引物用于反应体系优化实验。扩增反应在博日PCR仪上进行,扩增程序为:94℃预变性5 min;94℃变性1 min,35℃退火1 min,72℃延伸1 min,5个循环;94℃变性1 min,52℃退火1 min,72℃ 延伸1 min,35个循环;最后72℃延伸10 min,4℃保存。本研究采用20 µL基本PCR反应体系,反应体系见表3。PCR产物采用1.5%的琼脂糖凝胶电泳检测。

表3 白花树SRAP-PCR原始反应体系(20µL)Table 3 The parameters of SRAP-PCR of S. tonkinensis(20 µL)
1.2.3 SRAP-PCR反应因素水平的确定与正交表的设计 为了确定 PCR反应中5个因素的最佳水平,采用正交设计L16(45)在 4个水平上进行试验。参与PCR反应的因素水平见表4,L16(45)正交设计方案见表5。
1.2.4 扩增产物的检测 PCR产物用1.5%的琼脂糖凝胶电泳,观察分析条带,得到白花树的SRAP-PCR反应5个因素的最佳水平及组合。
1.2.5 SRAP-PCR反应体系的确立 以上述确定的条件,用初筛的引物组合对随机选取的DNA模板分别进行扩增,对优化过的SRAP反应体系的稳定性进行检测。

表4 白花树SRAP-PCR反应因素水平Table 4 Factors and levels of SRAP-PCR reaction in S. tonkinensis

表5 白花树SRAP-PCR反应因素水平L16(45)正交设计Table 5 Orthogonal design for SRAP-PCR optimization in S. tonkinensis
2 结果与分析
2.1白花树基因组DNA的提取
用CTAB法提取DNA,电泳检测结果显示,获得的DNA样品量大,条带清晰(图1)。经DNA/RNA紫外分光光度计进行定性和定量检测,仅OD260/OD280 >1.8的DNA样品方可用于下面的PCR反应。

图1 20个白花树样品DNA电泳结果Figure 1 The electrophoresis results of twenty DNA from S. tonkinensis
2.2 引物筛选试验
从20份种源中随机各挑选一个DNA样品,共20个PCR扩增模板,进行引物初筛。从9个SRAP正向引物和22个反向引物组合成的198个组合中,筛选出适于不同白花树种源遗传分析的引物8对(表6)。从筛选的引物中选取条带适中且清晰的Me1/Em8引物组合(图2),进行SRAP-PCR反应体系优化实验。

表6 适于不同白花树种源SRAP分析的引物Table 6 Primers suitable for SRAP analysis in S. tonkinensis
2.3 PCR反应体系的优化
2.3.1 正交实验结果分析 按照表5正交试验设计的16个反应体系进行扩增,随机选择1个白花树DNA模板进行试验。L16(45)正交试验PCR产物电泳结果见图3,扩增体系是否适合,关键是扩增结果所反应的信息量,即凝胶电泳图片中的有效条带数量和清晰度,按照遗传多样性分析要求,对PCR扩增结果进行分析,以条带数量丰富、清晰度高作为评价标准。初步选出9号和12号处理,并用于反应体系的再次筛选。其他处理表现出条带模糊或缺失,或是出现非特异性扩增,都不符合预期的目标。
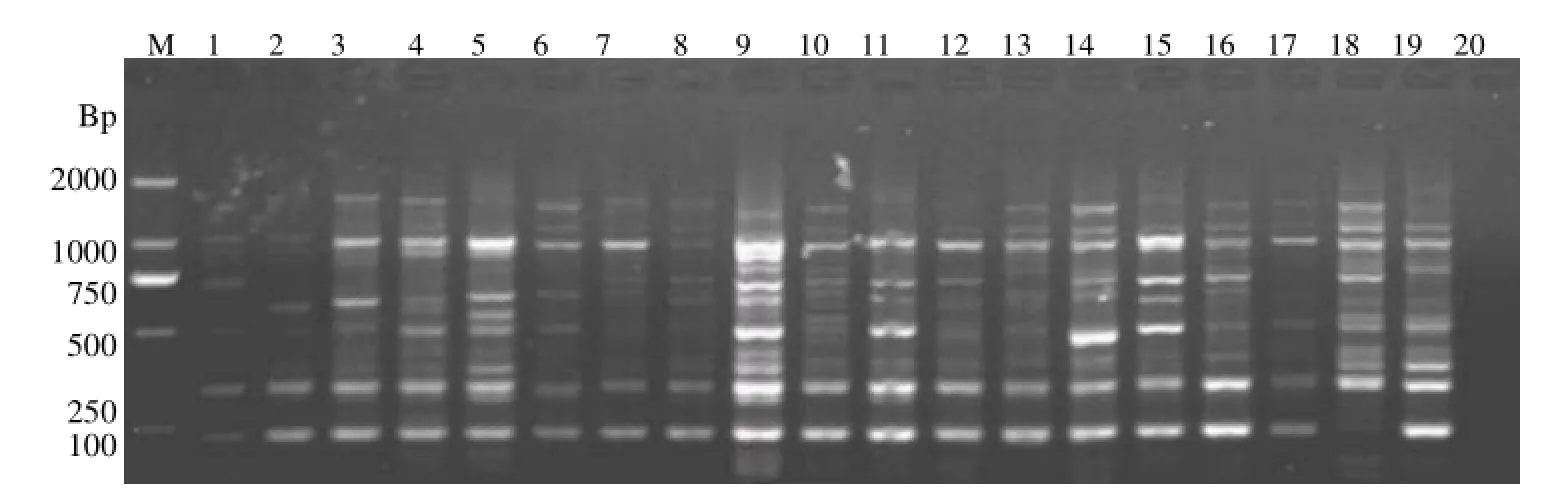
图2 Me1/Em8引物组合PCR扩增结果Figure 2 The SRAP amplified result by the primer pair Me1/Em8
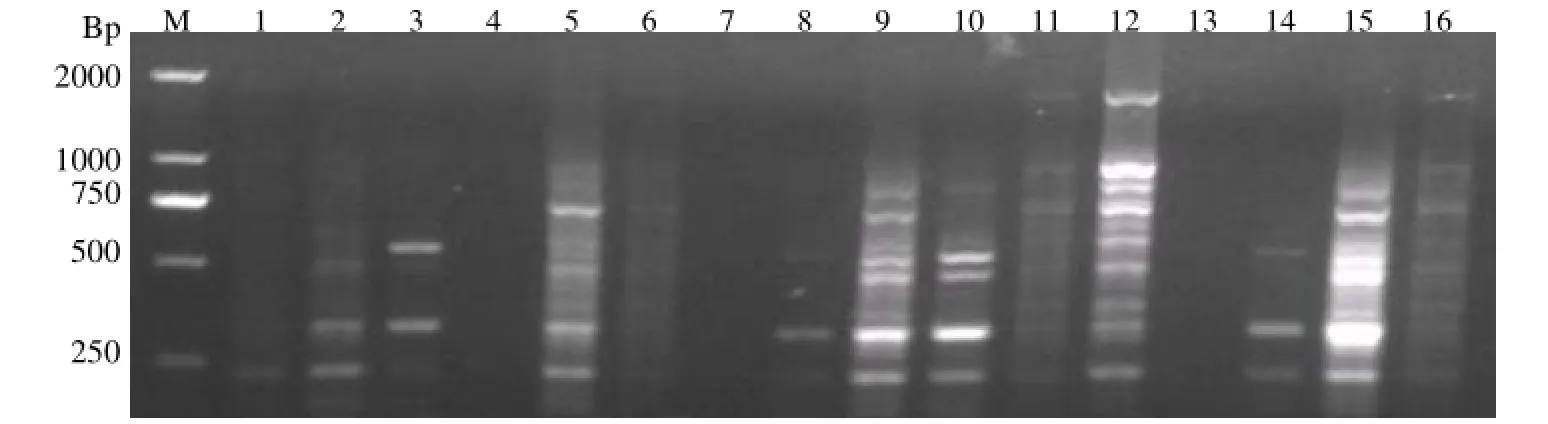
图3 SRAP-PCR扩增正交试验电泳结果Figure 3 The results of SRAP-PCR system with orthogonal design
2.3.2 PCR反应体系的筛选 从20个白花树种源中再随机挑选8个DNA样品,进行反应体系的再次筛选,并将原始反应体系设为对照。从图4可以看出,每个样品的3个处理,12号相比9号和对照来说,条带数量清晰且较丰富,每个泳道都有条带扩增。PCR反应中对模板浓度要求的范围较宽,一般每个反应中5 ~ 500 ng DNA都能提供好的结果,但不同物种的DNA进行PCR扩增时,适合的浓度不同[14]。结合实验分析结果和节约实验材料角度综合考虑,本试验选择20 µL体系终浓度24 ng为模板最佳浓度水平。
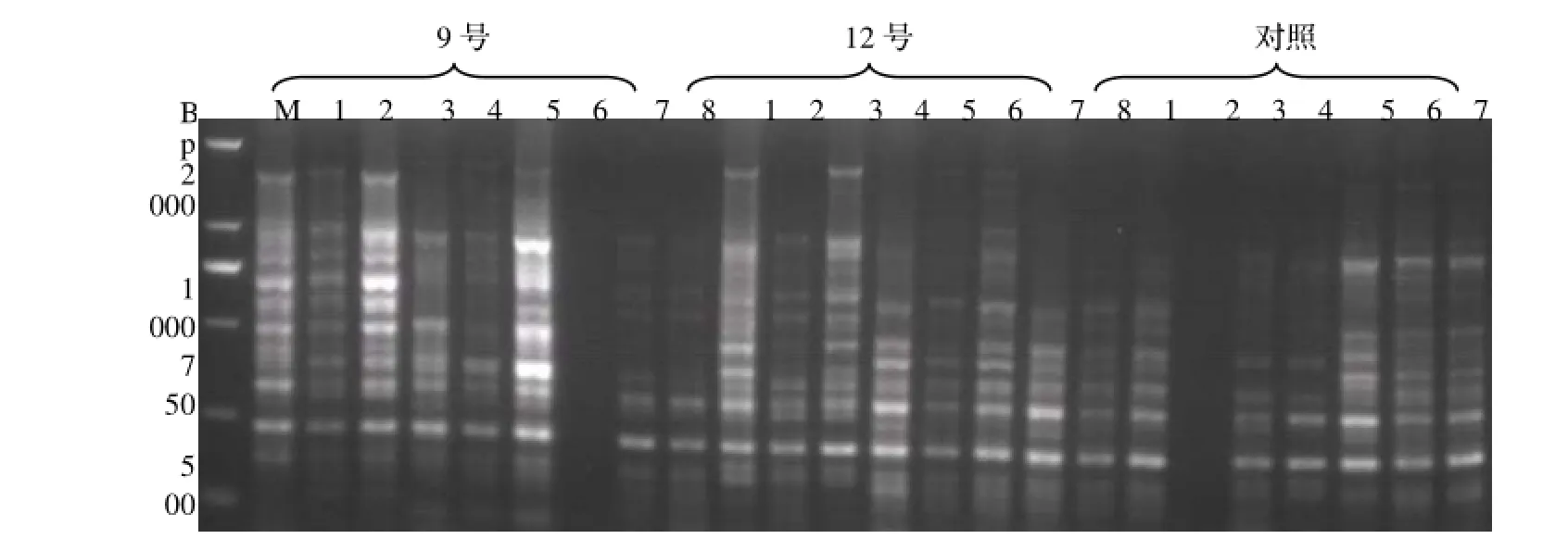
图4 9号、12号处理和对照的SRAP-PCR产物电泳结果Figure 4 The influence of different treatments on amplified result
2.3.3 优化体系的应用 以优化的体系,用Me3/Em8、Me4/Em8、Me6/Em16引物组合分别对随机选取的白花树DNA样品进行扩增(图5),电泳检测扩增谱带清晰、多态性强、稳定性好,该体系适合白花树SPAP-PCR反应。

图5 3个引物组合在部分DNA样品上的扩增Figure 5 The influence of 3 primers on amplified product
3 结论与讨论
SRAP已被成功应用于多种植物的遗传多样性分析、遗传图谱构建、重要性状标记以及相关基因克隆等,如响叶杨×银白杨(Populus adenopoda×P. alba)[15]、柿树(Diospyros kaki)[16]、红松(Pinus koraiensis)[17]、青葙(Celosia argentea)[18]、甘蓝型油菜(Brassica napus)[19]、菊花(Chrysanthemum morifolium)[20]、甜瓜(Cucumis melo)[21]等。SRAP分子标记是基于PCR的标记,其反应条件易受各种因素的干扰,对实验结果产生影响。本研究首次构建了白花树的SRAP-PCR反应体系,并对模板DNA浓度、Mg2+浓度、dNTPs浓度、引物浓度、Taq DNA聚合酶等因子进行了优化,建立了一套适合白花树的SRAP-PCR反应体系。通过研究,确定了白花树SRAP-PCR 20 µL反应体系中,用24 ng模板DNA,2.0 mM的Mg2+,0.5 mM的dNTPs,1.25 uM的引物,1.5 U的Taq DNA聚合酶,2 µL的10×PCR缓冲液为最优的SRAP-PCR反应组合。此反应体系能扩增出多态性强,条带清晰,重复性好的结果,能够应用于白花树的分子标记分析,该体系为进一步应用于白花树的遗传多样性分析、遗传格局研究和种质鉴别奠定了基础。
[1] 中国科学院中国植物志编辑委员会. 中国植物志:第六十卷:第二分册[M]. 北京:科学出版社, 2004.
[2] 柳新红,李因刚,何小勇. 白花树研究进展[J]. 浙江林业科技,2008,28(5):61-65.
[3] 国家药典委员会. 中华人民共和国药典(Ⅰ部)[M]. 北京:化学工业出版社, 2005.
[4] G. Li C F Q. Sequence-related amplified polymorphism (SRAP), a new marker system based on asimple PCR reaction:its application to mapping and gene tagging in Brassica[J]. Theor Appl Genet, 2001(103):455-461.
[5] 曹亮,魏宝阳,李顺祥. SRAP和SCAR分子标记应用于中药材研究进展[J]. 科学导报,2010(12):104-109.
[6]杨迎花,李先信,曾柏全,邓子牛. 新型分子标记SRAP的原理及其研究进展[J].湖南农业科学,2009(5):15-17, 20.
[7] 徐操,赵宝华. SRAP分子标记的研究进展及其应用[J]. 生命科学仪器,2009(4):24-27.
[8] 邱文武,孙伟生,窦美安. 基于PCR的新型分子标记——SRAP研究进展[J]. 江西农业学报,2007,19(8):26-28.
[9] 李建军,刘志坚,肖层林,等. SRAP技术在遗传的研究进展[J]. 现代生物医学进展,2007,7(5):783-786.
[10] 唐玉海,郭春芳,张木清. 相关序列扩增多态性(SRAP)标记及其应用研究进展[J]. 生物技术通报,2006(C00):237-241.
[11] Ferriol M, Pico B, Nuez F. Genetic diversity of a germplasm collection of Cucurbita pepo using SRAP and AFLP markers[J]. Theor Appl Genet. 2003(107):271-282.
[12] Li G, Gao M, Yang B,et al. Gene for gene alignment between the Brassica and Arabidopsis genomes by direct transcriptome mapping[J]. Theor Appl Genet. 2003, 107(1):168-180.
[13] Li G, Quiros C F. Sequence-related amplified polymorphism(SRAP),a new marker system based on a simple PCR reaction:Its application to mapping and gene tagging in Brassica[J]. Theoretical and Applied Genetics103. 2001, 103(2-3):455-461.
[14] 杨迎花,李先信,曾柏全,等. 新型分子标记SRAP的原理及其研究进展[J]. 湖南农业科学,2009(5):15-17.
[15]Yuanxiu W, Xiaoyan S, Tan B,et al. A genetic linkage map of Populus adenopoda Maxim.× P.alba L.hybrid based on SSR and SRAPmarkers[J]. Euphytica. 2010(173):193-205.
[16] Da-Long G, Zheng-Rong L. Genetic relationships of some PCNA persimmons (Diospyros kaki Thunb.) from China and Japan revealed by SRAP analysis[J]. Genetic Resources and CropEvolution. 2006(53):1 597-1 603.
[17] Min-Min C, Fujuan F, Xin S,et al. Construction of a framework map for Pinus koraiensis Sieb.et Zucc. using SRAP, SSR and ISSR markers[J]. Trees, 2010(24):685-693.
[18] Na F, Qie X, Qinghua G,et al. Genetic Diversity and Population Structure of Celosia argentea and Related Species Revealed by SRAP[J]. Biochem Genet, 2009(47):521-532.
[19] Zudong S, Zining W, Jinxing T,et al. An ultradense genetic recombination map for Brassica napus, consisting of 13551 SRAP markers[J]. TAG Theoretical and Applied Genetics, 2007, 114(8):1 305-1 317.
[20] Fei Z, Sumei C, Fadi C,et al. Genetic analysis and associated SRAP markers for flowering traits of chrysanthemum (Chrysanthemum morifolium) [J]. Euphytica, 2011, 177(1):15-24.
[21] Jianshe W, Jianchun Y, Wei L. Construction of a molecular map for melon ( Cucumis melo ) based on SRAP[J]. Acta Horticulturae Sinica. 2007, 34(1):135-140.
Optimization of SRAP-PCR System for Styrax tonkinensis
LIU Xin-hong1,2,LI Nan2,LI Yin-gang2,XU Liang2,SHENG Wei-tong1,HUI Gang-ying1,ZHENG Yong-qi1
(1. Institute of Forestry Research, Chinese Academy of Forestry, Beijing 100091, China; 2. Zhejiang Forestry Academy, Hangzhou 310023, China)
The reaction system was established based on total DNA extracted from Styrax tonkinensis leaves, by using orthogonal design of L16(45),which is the first report of S. tonkinensis SRAP-PCR reaction system. The optimal PCR system is as follows: 24 ng genomic DNA templates,1.0 mM Mg2+,1 mM dNTPs,0.75 uM primer,1.5 U/20µL Taq DNA polymerase and 2µL 10×PCR buffer in 20 µL reaction system. The clear, stable and abundant polymorphism bands were obtained by using above reaction systems. The results can be lay a foundation for studying genetic differentiation and construction of genetic map from different S. tonkinensis.
Styrax tonkinensis; orthogonal design; SRAP-PCR; system optimization
S718.43
A
1001-3776(2011)06-0030-05
2011-06-09;
2011-09-20
浙江省重大科技专项“食用油料与能源植物定向培育及开发利用研究”(2008C12019),浙江省院合作林业科技项目“早实高产白花树生物柴油能源定向培育与制取工艺研究”(2010SY05),浙江省创新团队建设与人才培养项目(20102F0014)
柳新红(1967-),男,浙江武义人,研究员,从事森林培育与育种研究。

