利用 16S-23S rDNA间隔区快速鉴定猪链球菌和马链球菌兽疫亚种
刘广锦,陈绵绵,商可心,范 洁,张 炜,姚火春,陆承平
(南京农业大学动物医学院,江苏南京,210095)
猪链球菌病(Porcine streptococcal disease)是由多种溶血性链球菌感染引起的猪的一种多型性病,包括急性型的败血症和慢性型的关节炎、心内膜炎、淋巴结脓肿、哺乳仔猪下痢和孕猪流产等。通过流行病学调查,本病在我国各地均有发生,猪链球菌与马链球菌兽疫亚种是我国猪源链球菌病的两种主要病原[1]。猪链球菌(Streptococcus suis,S.suis)是 1987年由 Kilpper-Balz等提议建立的1个新种[2],能引起猪和其它家畜及人类的多种临床疾病,按照其荚膜抗原的差异,目前分为 35个血清型(1-34,1/2)和相当数量无法定型的菌株[2],其中猪链球菌 1型、1/2型、2型(S.suis serotype 2,SS2)、7型、9型属于在世界上流行较广的致病性血清型[3],对养猪业及公共卫生均构成严重威胁。马链球菌兽疫亚种(Streptococcus equi subsp.zooepidemicus,S.zooepidemicus)属于兰氏分群的C群链球菌,其没有宿主专嗜性,可感染牛、猪、猴子和羊[4~6],人类因食用污染的食物或与发病动物密切接触后,亦可引发该菌感染[7,8],是一种重要的人兽共患病的病原。1977年四川地区发生过该病的大流行,造成 30多万头猪死亡,迄今为止仍呈地方流行[9]。因此,建立快速、敏感、特异的检测方法来鉴别这两种病原菌,对防制猪链球菌病具有重要作用。
在 16S rDNA与 23S rDNA之间,有一段连接序列,称为 16S-23S rDNA间隔区(16S-23S rDNA gene intergenic spacer region,ISR),是非功能区,所处的选择压力很小,进化速度是 16S rDNA的 10倍,Schlege等[10]报道了 ISR分析是一个鉴定链球菌属内的种(包括猪链球菌)的很有力的工具。因此,笔者就从 16S-23S rDNA ISR的高进化率特征入手设计引物,使用 PCR方法区分导致猪源链球菌病的两种重要病原——猪链球菌 2型和马链球菌兽疫亚种。
随着当前生物信息学的快速发展,许多重要的人畜共患病病原的全基因组序列和 16S rDNA,16S-23S rDNA ISR,23S rDNA序列都已公布,可以在美国国立生物技术信息中心(NCBI)和英国基因组研究中心(Sanger)免费下载。同时,还可利用 NCBI中 Primer-BLAST功能,以 GenBank中收录的大量核苷酸序列作为模板进行“虚拟 PCR”,为设计正确的 PCR引物和检测引物的特异性提供了优越的平台。本实验就是先下载大量序列进行分析比对,分别在 16S rDNA末端和 23S rDNA前端保守区设计了上下游引物,在Primer-BLAST中验证引物的特异性,然后再进行PCR的操作,确保实验的真实可靠性。通过PCR,发现实验中的 21株猪链球菌均扩增出长度约为 1 100 bp的片段,8株马链球菌兽疫亚种均扩出约1 300 bp的片段,其他属菌株和阴性对照无结果。这样,由PCR产物长度的不同就可快速区分猪链球菌和马链球菌兽疫亚种。最后,将代表株(HA9801,ATCC 35246)的PCR产物进行测序,进一步验证了实验的准确性,同时也发现猪链球菌和马链球菌兽疫亚种 ISR最大的差异表现为猪链球菌有一段 172 bp基因的缺失。
1 材料与方法
1.1 材料
1.1.1 菌株 本实验室保存的12株猪链球菌 2型;6株猪链球菌9型;猪链球菌 1型,1/2型,7型各 1株;8株马链球菌兽疫亚种;其他菌株:金黄色葡萄球菌,大肠杆菌Top10,嗜水气单胞菌 W 1,嗜水气单胞菌 J1。共 33株细菌(表 1)。

表 1 菌株列表
1.1.2 试剂 PCR Mix,200 bp DNA Ladder Marker和细菌DNA抽提试剂盒和 PCR产物回收试剂盒为TIANGEN产品;pMD19-T连接载体,DL2000Marker购自 Takara公司;核酸染料Goldview购自赛百盛生物科技公司;THB培养基为美国BD公司出品;PCR仪采用Biometra公司产品,凝胶成像系统出品于 Bio-RAD。
1.1.3 引物 本次研究利用 NCBI(www.ncbi.nlm.nih.gov)和 Sanger(www.sanger.ac.uk)中心上公布的基因序列,下载猪链球菌中 3株(05ZYH 33,98HAH 33,P1/7)已完全测序的全基因组序列,7株猪链球菌 2型和 3株马链球菌兽疫亚种的 16S-23S rDNA间隔区序列,在 DNASTAR软件 MegAlign中进行基因比对,使用Primer Prem ier 5.0结合 Oligo 6.0引物设计软件分别在 16S rDNA的 3'端和 23S rDNA 5'端保守区设计上下游引物,扩增 16S-23S rDNA ISR完整序列,扩增片断长度约为1 100~1 300bp。上下游引物序列分别为KISR1:5'-AAG CCA GTC TCA GTT CG-3';K-ISR 2:5'-TAG GGT TGG GAG ATG GT-3'。
1.2 方法
1.2.1 细菌 DNA的提取 金黄色葡萄球菌、大肠杆菌 Top 10,嗜水气单胞菌W 1,J1株接种于LB培养基,其余链球菌均接种于 THB培养基,37℃培养过夜后按TIANGENE公司细菌(革兰氏阳性菌或阴性菌)基因组DNA提取试剂盒操作说明进行抽提。DNA样品 -20℃保存备用。
1.2.2 PCR反应 PCR反应在 25μL体系中进行,2×PCR Mix 12.5μL,ddH2O 9.5μL,细菌 DNA样品 1 μL,引物各 1μL。PCR循环参数:94℃预变性 5min;然后 94℃ 1min,50℃ 30 s,72℃ 1.5m in,循环扩增30次;最后 72℃延伸 10min,于 4℃结束反应。
1.2.3 PCR产物电泳及回收 配制含Goldwiew的琼脂糖质量浓度为 10 g/L的凝胶,5 V/cm条件下电泳30min后,在凝胶成像系统中观察条带,按 TIANGENE公司 PCR产物回收试剂盒说明对HA9801,ATCC 35246两个样品进行 PCR产物回收,准备连接PMD19-T载体。
1.3 基因克隆与鉴定
1.3.1 感受态细胞的制备 接种活力菌E.coliTop10置 37℃摇床上 200 r/min震摇培养 2~3 h,测得菌液 OD590约为0.375。拿出菌液在冰上放置 10m in使之停止生长,吸取 1.5mL菌液于EP管中,4℃ 5 000 r/m in离心 10min,弃去上清,在沉淀中加入预冷的 0.1mol/L CaCl2600μL,轻悬菌体,在冰上放置 30min。然后,再次 4℃ 5 000 r/min离心 10 m in,弃去上清,在沉淀中加入预冷的 0.1mol/L CaCl2200μL,轻轻重悬菌体,4℃存放 12 h后进行转化。
1.3.2 目的基因连接 T载体 利用T-A连接,将纯化的PCR产物直接连接于pMD19-T载体中,连接体系为:Solution I 5 μL,PCR回收产物 4μL,PMD 19-T载体 1μL。16℃水浴 1 h后转化到 Top10感受态细胞中。
1.3.3 转化 取连接产物4μL,感受态细胞 200μL混合均匀,冰上放置 30min。42℃水浴热休克 90 s后,迅速取出放置冰上 2min。向混合液中加入新鲜 LB培养液 800μL,置 37℃摇床上 100 r/m in震摇培养 1h,4 000 r/min离心 5min。弃去 800μL上清,用剩余的液体重悬菌体,涂布含有 Amp的抗生素平板,置 37℃温箱培养过夜。
1.3.4 鉴定 挑选单菌落,接于含有 Amp的 LB培养基培养 6 h,进行 PCR鉴定,确定已连接到pMD19-T载体中。
1.3.5 测序及序列分析 挑选HA 9801,ATCC 35246菌株的阳性克隆送上海英骏生物工程公司进行测序。使用 NCBI中的 BLAST功能与 GenBank中收录的核苷酸序列进行同源性分析,并利用DNASTAR分析软件将测序所得 2株细菌的基因核苷酸序列进行比对。
2 结果与分析
2.1 PCR扩增结果
以提取的各株细菌基因组为模板,以 K-ISR1和 K-ISR2为引物扩增,所有各型猪链球菌的扩增片断均相同,约为1 100 bp,8株马链球菌兽疫亚种扩增的片段也均在大约 1 300bp,这样在片断长度上就区分了猪链球菌和马链球菌兽疫亚种。其他菌株和阴性对照均无结果(图 1~图 3)。
2.2 测序结果
挑选 HA 9801,ATCC 35246菌株的阳性克隆经 DNA双向测序后,与 GenBank上相关序列(CP000408,DQ204516)比较,同源性分别为 99%,100%,进一步确定了实验的准确性。在 DNASTAR软件MegAlign中将 HA 9801,ATCC 35246进行基因比对,结果见图 4。

图 1 SS2和 S.zooepidemicus为模板的 PCR扩增结果
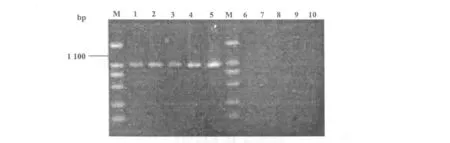
图 2 SS9和其他属细菌为模板的PCR扩增结果

图 3 SS1,SS1/2,SS7和S.zooepidemicus为模版的 PCR扩增结果

图 4 HA 9801和 ATCC 35246序列比对结果
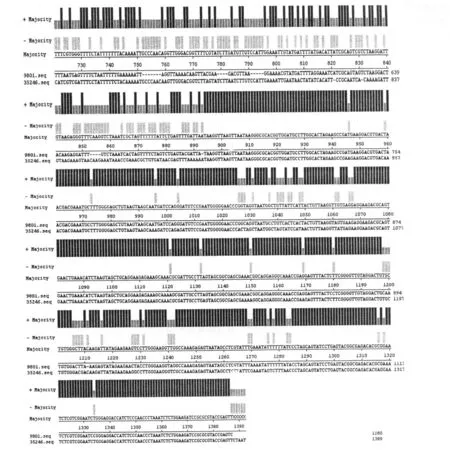
续图 4 HA 9801和 ATCC 35246序列比对结果
截去 16S rDNA末端和 23S rDNA前端的保守区,16S-23S rDNA ISR完整序列由第 294 bp至 892 bp,长度分别为:HA9801=423 bp,ATCC 352465=595bp。 2株菌的 16S-23S rDNA ISR均只编码 1个 tRNAAla基因,在 tRNAAla基因前序列变化不大,但HA 9801比ATCC 35246缺失了约 172个碱基。tRNAAla序列基本一致,在 tRNAAla基因之后基因插入和突变较多。
2.3 Primer-BLAST
利用 Primer-BLAST功能,将使用的引物在 NCBI中进行虚拟 PCR,K-ISR1与 K-ISR2这对引物还与肺炎链球菌 CGSP14株同源性为 100%,但预计扩增片断大小约在 940 bp,从长度上与猪链球菌和马链球菌兽疫亚种有明显区别。
3 讨 论
关于细菌的分类鉴定标准很多,常见的细菌分类方法有表型(phenotype)分型法和基因(genotype)分型法[11]。表型分型法中有生化分型法、血清分型法、抗菌谱分型法、荚膜分型法和噬菌体分型法等。其中的一些方法操作较简单,临床结合密切,被广泛应用,但多有粗糙、费时费力,不能够较好的区分流行菌株及散发菌株,强毒株与弱毒株。例如根据荚膜抗原的差异将 S.suis分为 35个血清型,但仍有相当数量无法定型的菌株,且荚膜血清型并不能反映菌株的毒力。此外,S.suis生化特性很不稳定,生化培养法常无法准确地定型而出现假阴性。随着分子生物学的快速发展,基因分型法逐渐在细菌分类中占有主导地位,在S.suis中主要有核糖体基因法、聚合酶链反应(PCR)法、随机扩增核酸片段多态性分析(RAPD)[12],脉冲场电泳(PFGE)[13]、扩增片断长度多态性(AFLP)[14]、基因测序等。每种方法分类的范围不同,RAPD,PFGE,AFLP等多用于确定种属的菌株分型鉴定,区分率高,但数据分析量大,需要借助计算机,费时费力,仪器也较昂贵。
赵冉等[15]针对 SS2毒力因子基因溶菌酶释放蛋白(MRP)、胞外因子(EF)、猪溶血素(SLY)设计三重引物进行多重PCR扩增,建立了 SS2强毒株的 PCR鉴定方法,但多重 PCR试验操作和反应条件的优化较为复杂。有文献研究表明[16],某些SS2没有这些毒力基因却一样具有致病性,有关的致病机理还有待于研究,且其他型的发病率也在不断上升。范红结等[17]根据S.zooepidemicus的毒力因子类M蛋白来鉴定菌株,但并未见用 16S-23S rDNA ISR来同时鉴别S.suis和 S.zooepidemicus的报道。由于 16S rDNA基因分子结构上的高度保守性、分布的普遍性及其所含的大量信息,使16S rDNA基因成为了一个较为理想的基因分类靶序列,但是不同种链球菌的 16S rRNA核苷酸的不同主要集中在某一个很小的高变区[18]。23S rDNA分子与 16S rDNA一样具有高度保守性,Harland等[19]利用 23S rDNA进行乳房链球菌和副乳房链球菌的鉴定,但 23S rDNA相对分子量较大,其基因数据库也不如16S rDNA齐全。本次试验利用16S-23S rDNA ISR长度和序列的多态性,结合16S rDNA和 23S rDNA的保守性,分别在 16S rDNA末端和 23S rDNA前端保守区设计上下游引物,巧妙地在序列长度上区分了S.suis和S.zooepidemicus,结果可直接在PCR核酸电泳图中看到,达到了快速鉴定两种病原的目的。
对 HA 9801和ATCC 35246的测序结果可见,碱基的插入造成了 16S-23S rDNA ISR长度的多态性。16S-23S rDNA ISR序列在编码 tRNAAla基因序列之前,HA 9801比 ATCC 35246缺失了约172个碱基,在 NCBI中 BLAST发现此缺失部分是马链球菌兽疫亚种特有序列,说明此部分反映了种间的差异,也为设计马链球菌兽疫亚种的鉴定引物提供了基础。两株代表菌的 tRNAAla基因序列完全一致,这与 Brain等[20]提出 16S-23S rDNA ISR的 tRNAAla基因序列在链球菌属中较为保守的结论相一致。在对其他细菌的16S-23S rDNA ISR研究中发现,革兰氏阳性细菌多编码 tRNAAla或 tRNAIla基因,革兰氏阴性菌根据 ISR的片断长短不等编码 tRNAAla,tRNAGla,tRNALla,tRNAVla,tRNAIla,还有部分 ISR根本不含tRNA基因[21~23]。在编码tRNAAla基因序列之后二株菌表现出较多碱基的不同,作为可变区。
在进行本试验前,从NCBI和Sanger中心下载了大量的基因序列,在比对中发现,rDNA在许多细菌基因组中都是有多个拷贝,说明rDNA在基因组中占有重要地位。同时拷贝数也不尽相同,如在猪链球菌,肺炎链球菌中是 4个,化脓链球菌中为 6个,嗜热链球菌则达到 7个。
[1] 陆承平.猪链球菌病与猪链球菌 2型[J].科技导报,2005,23(9):9-10.
[2] KILPPER-BALZ R,SCHLEIFER K H.Streptococcus suis sp.nov.rev[J].Int Syst Bacteriol,1987,37:160-162.
[3] STAATS JJ,FEDER I,OKWUMABUA O,etal.Streptococcus suis:Past and Present[J].Vet Res Commun,1997,21:381-407.
[4] SHARP M W,PRINCE M J,GIBBENS J.Streptococcus zooepidemicus in fection and bovine mastitis[J].Vet Rec,1995,137:128-129.
[5] SALASIA S I,WIBAWAN IW,PASARIBU F H,et al.Persistentoccurrence of a single S.zooepidem icus clone in the pig andmonkey population in Indonesia[J].Vet Sci,2004,5(3):263-265.
[6] ALFONSO Las Heras,ANA Vela,ELENA Fernández,etal.Unusual outbreak of clinicalmastitis in dairy sheep caused by S.zooepidemicus[J].Clin Microbiol,2002,40(3):1 106-1 108.
[7] URALO,TUNCER I,DIKICIN,et al.Streptococcus zooepidem icusmeningitis and bacteraemia[J].Scand InfectDis,2003,35(3):206-207.
[8] KORMAN TM,BOERS A,GOODING TM,et al.Fatal case of toxic shock-like syndrome due to group CStreptococcus associated with superantigen exotoxin[J].JClin Microbiol,2004,42:2 866-2 869.
[9] 陆承平.兽医微生物学[M].第三版.北京:中国农业出版社,2001.
[10] SCHLEGE L,GRIMONT F,GRIMONT P A,et al.Idencation ofmajor streptococcal species by rna-amPlified ribosomal DNA restriction analysis[J].JClin Microbiol,2003,41:657-666.
[11] VANDAMME P,POT B,GILLISM,etal.Polyphasic Taxonomy,a Consensus Approach to Bacterial Systematics[J].Microbiological Reviews,1996,60(2):407-438.
[12] CHATELLIER S,GOTTSCHALK M,R Higgins,et al.Relatedness of Streptococcus suis serotype 2 isolated from different geographic origins as evaluated bymolecular fingerprinting and Phenotyping[J].Clin Microbiol,1999,37:362-366.
[13] BERTHELOT-HERAULT F,MAROIS C,GOTTSCHALK M,et al.Genetic Diver-Sity of Streptococcus suis Strains Isolated from Pigs and Humans as Revealed by pu lsed-field Gel Electrophoresis[J].Clin Microbiol,2002,40(2):615-629.
[14] REHM T,BAUMSC G,STRONUNENGER B,et al.Amplified fragment length polymorphism of Streptococcus suis strains correlateswith their profile of virulence-associated genes and clinical background[J].Med Microbiol,2007,56(1):102-109.
[15] 赵冉,孙建和,陆承平.多重 PCR鉴定猪链球菌 2型毒力菌株[J].上海交通大学学报:农业科学版,2006,24(6):503-506,528.
[16] BERTHELOT-HERAULT F,MORVAN H,KERIBIN A M,et al.Production of Muraminidase-Released Protein(MRP),Extracellular Factor(EF)and Suilysin by field isolates of Streptococcus suis Capsular type 2,1/2,9,7and 3 isolated from swine in Frence[J].Vet Res,2000,3l:473-479.
[17] 范红结,陆承平,唐家琪.马链球菌兽疫亚种类M蛋白的基因克隆、序列分析及其在猪源链球菌的检测[J].微生物学报,2004,44(5):617-620.
[18] FORSMAN P,TILSALA-TIMISJARVIA,T Alatossava.Identication of Staphylococca l and Streptococca l causes of Bovine mastitis using 16s-23s rRNA spacer regions[J].Microbiology,1997,143:3 491-3 500.
[19] HARLAND N M,LEIGH JA,COLLINSM D.Development of gene p robes for the specific iden fitication of Streptococcus uberis and Streptococcus parauberisbased upon large subunit rRNA gene sequence[J].App l Bacteriol,2000,74:526-531.
[20] BERRIDGEB R,FULLER JD,AZAVEDO J de,etal.Development of Specific Nested Oligonucleotide PCR Primers for the Streptococcus iniae 16S-23S Ribosomal DNA Intergenic Spacer region[J].J Clin Microbiol,1998,36(9):2 778-2 781.
[21] APPUHAMY S,LOW JC,PARTON R,et al.Specific PCR primers from the 16S-23S rRNA spacer region for the rapid detection and identification of Actinobacillus seminis[J].Journal of Appiled Microbiology,1998,85:941-948.
[22] CHUN J,HUQ A,COLWELL R R.Analysis of 16S-23S rRNA Intergenic Spacer Region of Vibrio cholerae and Vibrio mim icus[J].Applied and EnvironmentalMicrobiology,1992:2 202-2 208.
[23] SRIPRAKASH K S,GARDINER D L.Lack of polymorphism within the rRNA operons of group A Streptococci[J].Mol Gen Genet,1997,255:125-130.
(责任编辑:朱宝昌)
——和田盘羊
- 河北科技师范学院学报的其它文章
- 金黄色葡萄球菌 isdB基因克隆、表达及其抗原性鉴定
- 肺外感染的非结核分枝杆菌

