绵羊毛性状EST-SSR标记聚合效应分析方法的建立
王遵宝,张文祥,赵宗胜,余鹏,吴洪宾
(石河子大学动物科技学院,石河子,832003)
绵羊毛性状EST-SSR标记聚合效应分析方法的建立
王遵宝,张文祥,赵宗胜,余鹏,吴洪宾
(石河子大学动物科技学院,石河子,832003)
为了实现绵羊毛性状的多基因聚合,对绵羊不同 EST-SSR标记基因型的性状表型值进行了聚类分析,以期建立一种适用于动物育种的多基因聚合分析方法。结果显示:6个标记与毛性状存在关联性,其彼此间的连锁程度较低,属于独立遗传,可用于多基因聚合的候选标记(D′小于0.5,r2小于0.3);通过标记基因型间性状相似性的聚类分析,获得了自然长度、拉伸长度、纤维直径方面及绵羊毛性状综合指标的4个最佳基因型类群,其不仅可作为绵羊毛性状选育的一种育种方案,而且有助于动物多基因聚合育种的研究。
绵羊;基因聚合;毛性状;聚类分析
羊毛是绵羊重要的经济性状,采用多基因聚合技术对毛性状进行综合选择,从而加快毛性状的育种进展,实现性状的整体改良提高,这是绵羊分子育种的必然趋势。多基因聚合是通过传统动物杂交技术结合分子信息,将有利基因聚合到同一个基因组,在分离世代,通过分子标记选择含有多个目标基因的个体,再从中选出优良生产性状的个体,该技术在植物上已获得了成功并在生产实践中开展应用[1-4]。然而在动物育种上目前还没有见到基因聚合成功的报道,仅见简单的合并基因型选择育种,其主要原因是对基因功能、调控网络及基因互作的研究报道较少,基因型与表型关联性和聚合分析方法不够成熟,因此有必要建立一种研究动物多基因聚合分析方法[5]。
利用分子标记技术在杂交和回交后代中检测获得目标基因聚合体是动物基因聚合采取的主要策略,而标记间的连锁互作关系会直接影响多基因的聚合效果。本研究分析了绵羊皮肤源多态 ESTSSR标记与毛性状的关联性,据此选出了对毛性状有影响的位点[9]。
本试验拟通过分析位点间的连锁互作关系,选出其中可用于多基因聚合的位点,并基于不同基因型间性状相似性对不同基因型进行聚类分析,最终获得针对不同性状及毛性状整体的最佳标记基因型组合,从而建立适用于动物多基因聚合的分析方法。本研究不仅可为后期绵羊毛性状育种计划的制定提供参考,而且有助于推动绵羊毛性状的多基因标记辅助选择技术的发展。
1 材料与方法
1.1 试验动物
本研究选用新疆兵团农九师165团某羊场德国美利奴羊×中国美利奴羊级进杂交 F2代196只3-6周岁基础母羊群体为供试群体,采集毛样及对应全血,ACD抗凝,-20℃。
1.2 EST-SSR位点选择
选用前期实验获得的9个多态性丰富的 ESTSSR标记位点用于聚合性状分析,相关引物由上海生工生物技术有限公司合成,具体信息见表1。

表1 所选EST-SSR引物序列及退火温度Tab.1 Primer sequence of EST-SSR markers and reference annealing temperature
1.3 羊毛性状测定
采集毛样时在绵羊肩胛部测定毛丛自然长度。参照文献[10]中的方法测定羊毛拉伸长度,每份样品的测定数量不少于300根。使用CU-I型纤维细度分析仪测定羊毛纤维直径,记录羊毛细度表型值。
1.4 基因组DNA提取及PCR扩增、产物检测
采用常规酚-氯仿法提取绵羊全血基因组DNA,用于PCR反应,其产物经聚丙烯酰胺凝胶电泳分离和银染后成像,根据标准MarkerⅠ读出扩增片段的大小,确定个体基因型[11]。
1.5 统计分析
本研究所用供试群体为同场同类群母羊,仅年龄有差异,据此建立固定效应模型,调用SPSS13.0软件的GLM过程分析基因型效应对性状测定值的影响,并进行多重比较(LSD法)。统计分析模型为:

上式中:Yij为性状测定值,μ为群体均值,Gi为基因型效应,Fj为年龄效应,(GF)ij为基因型与年龄的交互效应,Eij为随机残差效应。
根据关联性分析结果选出影响绵羊毛性状的标记,用Shesis软件对6个位点进行连锁不平衡检验,分析其间连锁互作程度;计算位点上不同基因型的毛性状均值,并基于性状间的欧几里德距离,运用SPSS 13.0软件对基因型进行聚类分析。
2 结果与分析
2.1 羊毛性状测定结果
毛性状测定结果(表2)表明该实验群体的3项毛性状指标符合正态分布(P>0.05),纤维直径均值为23.89μm,自然长度和拉伸长度的均值分别为9.64 cm和13.82 cm。
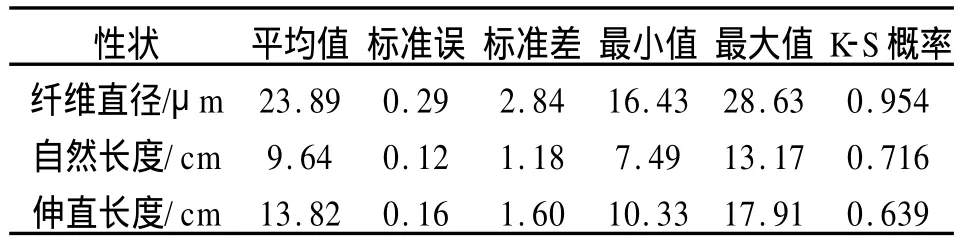
表2 羊毛性状统计分析结果Tab.2 The results of wool traits analysis
2.2 EST-SSR标记位点与毛性状的关联性分析统计
根据关联性分析结果(表3),筛选获得了不同基因型在羊毛性状表现上存在差异显著的6个位点。其中影响羊毛自然长度、纤维直径的位点各有5个,影响羊毛拉伸长度的位点有4个;SER012、SER016、SER018 3个位点对3个性状均有影响,而位点SER011专一性地影响了羊毛直径。

表3 影响羊毛不同性状的位点统计Tab.3 The summary of wool traits correlative loci
2.3 EST-SSR标记位点的连锁不平衡分析
结果见表4。

表4 6个EST-SSR位点间连锁不平衡分析Tab.4 The analysis of Linkage disequilibrium among six EST-SSR markers
连锁不平衡分析的结果(表4)显示,位点间的D′均小于 0.5(最大 0.303),r2均小于 0.3(最大0.016),表明这些位点的连锁程度很低,属于独立遗传,可用于性状的多基因聚合研究[12]。
2.4 不同位点基因型聚类结果及类群间毛性状均值的多重比较
根据不同基因型间性状的欧式距离,分别进行了单性状基因型和多性状基因型聚类分析,以此绘制了毛性状基因型聚类树图(图1~4),并参考其结果对所有基因型进行了分类,即每个性状分为5个类群,用于计算每个类群的平均表型值,筛选最佳基因型组合。
在自然长度方面,SER09AB、SER010CC、SER09CD、SER010AC、SER012AD、SER018AD、SER018BC 7个基因型聚为一类,其自然长度均值达(10.383±0.139)cm,为最佳组合;拉伸长度方面,SER009AB、SER018AD 2个基因型聚为一类,其拉伸长度均值达(15.5300±0.6505)cm,为最长类型;纤维直径方面,SER010CC、SER012EE、SER011AC、SER018AE、SER011BC 5 个基因型聚为一类,纤维直径最细,为(21.5900±0.4048)μm;对绵羊毛性状的3个指标整体的基因型聚类分析结果 显 示,SER09CD、SER018AD、SER011BC、SER010CC、SER011AC、SER018AE 6个基因型聚为一类,并表现出最佳的毛用性状,其自然长度达(10.1467±0.0.3578)cm,拉伸长度达(14.6667±0.7092)cm,平均直径为(22.0267±0.8643)μm。针对各性状类群间的多重比较结果表明,各类群间差异均达显著水平(P<0.05)。详细结果见表5和表6。

图2 拉伸长度基因型聚类图Fig.2 The cluster results for stretched length

图1 自然长度基因型聚类图Fig.1 The cluster results for natural length
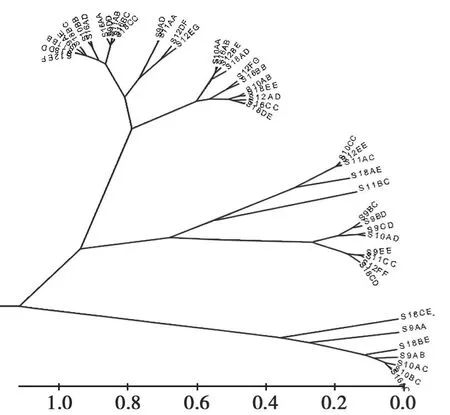
图3 纤维直径基因型聚类图Fig.3 The cluster results for fiber diameter

图4 综合毛性状基因型聚类图Fig.4 The cluster results for comprehensive traits
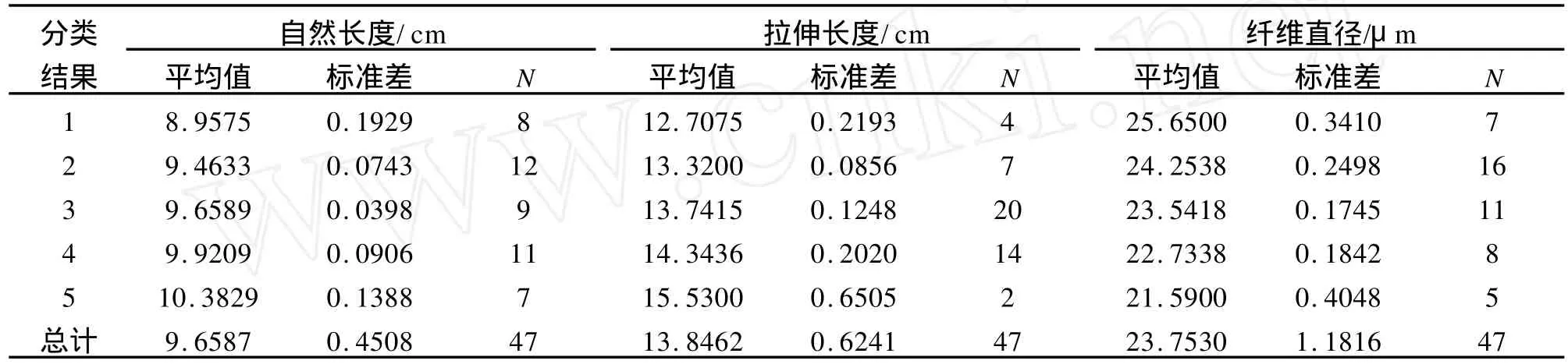
表5 不同性状的基因型聚类结果Tab.5 The cluster results of genotypes among different traits

表6 毛性状整体的基因型聚类结果Tab.6 The cluster results of genotypes for comprehensive wool traits
3 讨论
基因聚合最早由 Yadav等[13]在研究芥菜抗病和抗逆性状改良时提出,就是将分散在不同个体、品种或品系中的理想基因聚合到同一个基因组中。分子标记辅助选择是当前基因聚合最普遍、有效的方法。DNA分子水平上分子标记技术具有数量多、多态性丰富、不受表型影响、共显性等特点,可为优良基因型的选择和聚合提供了技术支撑。目前,国内外已在植物育种中进行了大量的研究,并且已经在水稻、棉花、小麦、大麦等农作物育种上取得了成功并得到应用[1-4]。然而在动物育种中目前还没有见到基因聚合成功的报道,仅见简单的标记辅助育种及多基因聚合效果预测。PIC(猪种改良公司)是最早应用标记辅助育种技术的公司,其最先将10个DNA标记用于猪的分子育种;2003年PIC公司在西班牙成功培育了能够抵御大肠杆菌F18所引发的仔猪腹泻的新品系,成为在猪中利用分子标记进行抗病育种的范例[14]。Cotswold公司从1986年开始通过基因检测技术剔除群体中氟烷基因,组建母系集团式核心群,已成功培育成了无应激猪品系[15]。李宁[16]选择可提高猪产仔数的 ESR和FSH合并基因型,设计开发了合并基因型DNA诊断试剂盒,并获得美国专利,其极大地带动了国内动物多基因聚合育种的研究。李学余等[17]通过分析白耳鸡群体PRL和POU1F1标记基因各基因型与产蛋数的关联,确定了 PRL和 POU1F1的有利单基因型(AA和CC),单基因聚合后获得的AACC基因型的72周龄产蛋数显著高于其他8组聚合基因型合个体。付言峰等[18]研究了猪 PPARγ2、VEGF、IL-8 3个基因突变位点多态性与产仔数的关系,并对各位点基因型进行聚类分析,结果表明VEGF基因的P4引物位点与 IL-8基因 P5引物位点聚类在第一内层,如将这2个基因位点进行基因聚合,将会显著提高猪产仔数。但以上这些研究对两位点间的连锁关系未作详细分析,其间的基因互作很可能会影响聚合效果。为了推动绵羊毛性状的标记辅助选择及多基因聚合育种的发展,本研究前期设计开发了9对多态性丰富的皮肤EST-SSR标记,结合与性状的关联性分析最终筛选出6个对毛性状有影响作用的位点。连锁不平衡分析结果显示,这6个位点属于独立遗传,可用于多基因聚合研究。通过聚类分析,分别获得了针对羊毛自然长度、拉伸长度、纤维直径及综合性状的最佳基因型聚合组合。在自然长度最佳类群里包含了 SER09、SER010和SER018 3个位点的2个基因型,这表明该性状可能在SER09和SER018位点存在较大的杂交效应,而SER010位点的C等位基因可能使自然长度的关键基因;拉伸长度方面,SER009AB、SER018AD 2个基因型聚为一类;纤维直径方面,提示SER011的C等位基因可能是纤维直径的有利基因;对绵羊毛性状的3个指标整体的基因型聚类分析结果显示 ,SER09、SER010、SER011、SER018 等4个位点的6个基因型聚为一类,并表现出最佳的毛用性状,这4个位点的最佳基因型组合可作为后期绵羊毛性状基因聚合育种的候选基因组合,但其真实的聚合效应有待进一步验证。
[1]巴沙拉特,丁效华,曾列先,等.水稻抗白叶枯病基因的聚合育种[J].分子植物育种,2006,4(4):493-499.
[2]夏贤仁,姬广海,张世光.基因聚合品种对云南水稻白叶枯病的抗性分析[J].石河子大学学报:自然科学版,2004,22(增刊):24-26.
[3]董章辉,石玉真,张建宏,等.棉花纤维长度主效QTLs的分子标记辅助选择及聚合效果研究[J].棉花学报,2009,21(4):279-283.
[4]董建力,惠红霞,王敬东,等.小麦 YrX、Pm21基因和Dx5优质基因聚合体的分子标记选择[J].西北农业学报,2008,17(4):58-61.
[5]黄冬维,张元跃,蒋隽,等.基因聚合在猪育种中的应用现状[J].猪与禽,2008,20(3):60-62.
[6]Ellis J R,Burke J M.EST-SSRs as a resource for population genetic analyses[J].Heredity,2007,99:125-132.
[7]Adelson D L,Cam G R,De Silva U,et al.Gene expression in sheep skin and wool(hair)[J].Genomics,2003,83:95-105.
[8]尹俊,李金泉,张燕军,等.内蒙古成年绒山羊皮肤基因表达的研究[J].畜牧兽医学报,2005,36(10):996-1000.
[9]王遵宝,努尔力·阿不力孜,赵宗胜,等.基于绵羊皮肤组织ESTs开发新型微卫星标记[J].中国农学通报,2010,26(3):42-45.
[10]赵有璋.羊生产学[J].北京:中国农业出版社,2002.
[11]刘云芳,高剑峰,潘晓亮,等.绵羊全血中DNA的微量提纯[J].石河子大学学报:自然科学版,1997,1(2):136-138.
[12]Pritchard J K,Przeworski M.Linkage disequilibrium in humans:models and data.American Journal of Human[J].Genetics,2001,69(1):1214.
[13]Yadav R D S,Singh S B,Singh A,et al.Gene pyramiding and horizontal resistance to diara stress in Mustards[J].National Academy Science Letters-India,1990,13(9):325-327.
[14]刘榜.国内外猪基因组与分子育种研究进展[J].猪业科学,2008,25(1):66-67.
[15]Liu X M,Smith C M,Gill B S.Identification of microsatellite markers linked to Russian wheat aphid resistance genes Dn4 and Dn6[J].Theor Appl Genet,2002,104:1042-1048.
[16]李宁.基因组学技术在动物遗传育种中的应用[J].华南农业大学学报,2005,26(增刊):12-19.
[17]张学余,李国辉,韩威,等.PRL和 POU1F1基因及基因聚合对白耳鸡产蛋数的效应分析[J].安徽农业大学学报,2010,37(2):249-252.
[18]付言峰,孔路军,邓慧,等.影响猪繁殖性状基因多态位点聚类分析[A].第十五次全国动物遗传育种学术讨论会论文集[C].杨凌,2009:155.
Establishment of Analytical Method to Evaluate the Effect of Gene Pyramiding in Ovine EST-SSR Markers about Wool Traits
WANG Zunbao,ZHANG Wenxiang,ZHAO Zongsheng,YU Peng,WU Hongbin
(College of Animal Science and Technology,Shihezi University,Shihezi,832003,China)
In order to achieve aggregation of multiple favorable genes on wool traits,this study carried out a cluster analysis on character phenotypic values between different genotypes of EST-SSR markers in sheep.Finally,we found 6 markers correlated with wool traits.There was no linkage disequilibrium among 6 loci,that is to say,they could be used as candidate markers for their independently hereditary in gene pyramiding(D′is less than 0.5,r2is less than 0.3).The results of cluster analysis based on traits similarity among genotypes revealed that the best four genotype groups were obtained in allusion to natural length,stretch length,fiber diameter and overall target of wool traits respectively.The results not only can be used for wool trait selection breeding programs,but also are helpful to animal breeding of gene pyramiding.
sheep;gene pyramiding;wool traits;cluster analysis
S813.3
A
1007-7383(2011)02-0179-05
2010-12-10
科技部国际科技合作项目(DF2007DFB30420),石河子大学研究生创新基金项目(YJCX2010-Y01)
王遵宝(1984-),男,硕士生,专业方向为动物遗传育种与繁殖;e-mail:wangzunbao@163.com。
赵宗胜(1969-),男,副教授,从事动物遗传育种与繁殖研究;e-mail:zhaozongsh@shzu.edu.cn。

