一种糖尿病动物模型基因芯片的聚类分析
沈青松, 黄文佳, 吕玉龙, 王翼飞
(上海大学 理学院,上海 200444)
一种糖尿病动物模型基因芯片的聚类分析
沈青松, 黄文佳, 吕玉龙, 王翼飞
(上海大学 理学院,上海 200444)
建立一种基因表达谱的聚类分析模型,通过信噪比处理、聚类结果的分析比较、相应标记的寻找,为芯片数据的后续分析以及寻找差异基因提供一种有效的方法.对一种糖尿病动物模型的小鼠基因表达谱进行实际分析,获取了有意义的结果,从而为糖尿病的快速和早期临床医学诊断提供有效的技术支撑.
基因表达谱;过程模型;聚类分析;差异基因
Abstract:Thispaper introduces a model of cluster analysis on the gene expression data.By processing signal-to-noise ratio,comparing the cluster analysis results and searching the corresponding markers,it offers an efficient method to post-analyze the gene exp ression data and discover different genes.Gene expression data of a diabetic mouse are analyzed,and significance of this analysis obtained,which facilitates technical support to rapid and early clinical diagnosisof diabetes.
Key words:gene exp ression profiles;p rocessmodel;cluster analysis;different gene
基因芯片,又称 DNA芯片 (DNA chip)或 DNA微阵列 (DNA microarray),是随着“人类基因组计划”(human genome p roject,HGP)的实施而发展起来的一项新技术,可广泛应用于基因序列分析、基因突变检测、多态性分析以及疾病的基因诊断等许多领域[1-2].
目前,在发达国家糖尿病已经成为导致人口死亡的第四大疾病.资料显示,全球糖尿病患者约 2亿人,其中中国约有 5 000万人受到糖尿病的困挠,患病率居世界第二位,并且以每天至少 3 000人的速度增加,每年增加人数超过 120万[3],因此,糖尿病已经成为严重的公共卫生问题之一.由于高密度基因芯片实验一次就能同时检测出成千上万个基因的表达,因此,该技术的出现为肿瘤及糖尿病等复杂性疾病的研究提供了一种新的实验手段和研究方法,并为这类疾病的诊断与医学治疗提供了有效的技术支撑,现在已经引起了社会各界广泛的重视.利用基因表达谱对肿瘤等复杂性疾病进行分类检测正逐步形成生物信息学的一个重要研究领域.但由于基因表达数据存在维数高、噪音大、样本数量小以及基因表达之间存在很大相关性等特点,深入而准确地挖掘DNA序列中蕴含的信息具有极大困难[4].自从Golub等以急性白血病基因表达谱数据为分类样本,提出基于权重表决的基因选择算法以来,许多机器学习方法被广泛地应用于肿瘤等高通量数据的两分类问题的研究.这些方法所做的主要工作就是降维、去噪和剔除冗余基因,目的就是提取信息基因 (即具有显著特性或影响其他基因变化的基因)或抽取综合属性信息,并采用合适的聚类方法,最大限度地把具有相同或类似作用的基因聚类到一块[4].
聚类分析方法把一个没有类别标记的样本集按照某种准则划分成若干个子集 (类),使相似的或者具有相近性质的样本尽可能归为一类,而不相似的样本尽可能分到不同的类中.然后,再对类中的数据进行分析与比较,看是否真的具有良好的聚类效果与生物学意义.
1 材料与方法
本研究以糖尿病动物模型Mouse C57BL6的小鼠基因芯片数据作为研究对象,相关数据下载于美国斯坦福大学 (http:∥smd.stanford.edu/cgi-bin/tools/display/listM icroArrayData.pl?tableName=publication).
本研究的目的是查明 CD4+T细胞在生长发育过程中非肥胖型糖尿病小鼠基因表达的变化情况.利用基因芯片基因表达分析中的胰腺淋巴细胞受体基因和正常小鼠在不同年龄的基因表达的比较,验证了α-干扰素对正常小鼠和病态小鼠的不同影响,从而为推迟或者降低小鼠糖尿病的发病、增强小鼠抵抗能力等,作出了相应的分析与阐述.
本研究的聚类分析问题的关键是:①如何根据基因表达谱,从基因空间中选择与糖尿病疾病有关的基因集合,或抽取有分类特征的综合属性;②如何根据信息基因或综合属性获得好的聚类效果.
1.1 芯片数据聚类分析过程模型
由于基因芯片数据的聚类分析是模式分类识别方法在生物医学领域的一个具体应用,因此,芯片数据聚类分析过程模型与模式分类识别过程模型具有相似性,主要分为基因表达谱数据获取、基因表达数据预处理和归一化、信息基因选择或综合属性抽取、样本聚类分析模型建立及测试与聚类结果评估.本研究得到的芯片数据聚类分析模型如图1所示.

图1 芯片数据聚类分析过程模型Fig.1 M odel of chip data cluster analysis processing
基因表达谱芯片数据的获取是一个相当复杂的实验操作过程,所得到的数据往往存在噪声大、质量较低等现象,某些数据还不在同一量纲范围.另外基因表达数据存在重复、缺失等现象,因此,要对基因表达谱芯片数据进行预处理和归一化,这是在分析基因表达谱数据之前一个非常重要的环节[5].本研究所涉及的微阵列数据预处理工作主要有:
(1)采用样本加权平均补齐基因表达数据.
(2)计算理想的非配对值 (ideal mismatch,IM),然后从完全配对 (perfectmismatch,PM)强度中减去 IM,对校正后的 PM进行对数转换.
包含配对 (mismatch,MM)探针的原因是提供一个估计非特异性杂交和其他影响 PM的偏离信号的值.计算理想的非配对值共分三种不同的情况,具体计算公式见文献[2].
当计算得到每个探针的 IM后,探针值为 Vi,j=PMi,j-IMi,j,对数转换后的探针值 (p robe value,PV)为PVi,j=log2(Vi,j).
(3)经对数转换后的值进行稳健性均数估计,然后进行反对数转换,对信号值进行标准化.
1.2 基因选择或综合属性抽取
由于基因表达谱中基因数量非常大,少则几千,多则上万,并且其中多数基因的表达与肿瘤无关,这为基因选择和综合属性抽取带来了很大困难,很难采用某一种简单的方法完成这一任务.因此,通常要对原始数据进行基因筛选,所选择的基因数量由实验来确定,其数量选择的合理性以及所选择基因的聚类效果也由最终与肿瘤作用相关的基因来评估[6],主要方法如下:
(1)应用 t-检验 (t-test)与设定阈值相结合的方法,对从几万个数据中筛选出的几千个基因数据进行聚类分析;
(2)应用信噪比方法 (signal to noise),本研究中的记分函数表示为
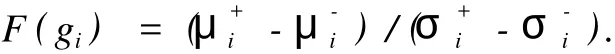
采用这些方法所选择的基因相互之间仍存在高度相关性,还必须采取某种策略精选出聚类能力更强的具有差异信息的基因,并进一步剔除冗余基因.
1.3 样本聚类分析模型建立
本研究对筛选出的基因号数据进行如下处理与假设规定.
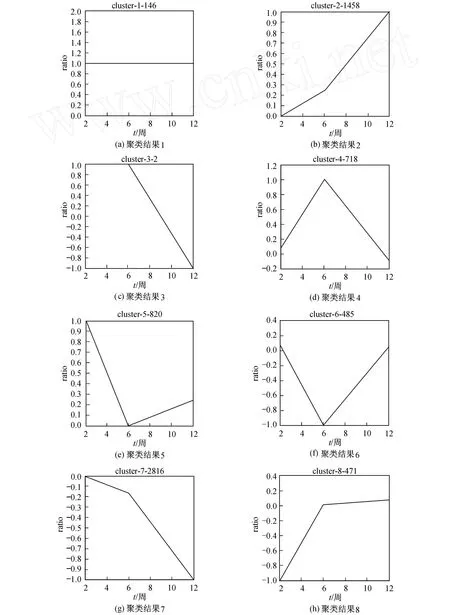
(1)对原数据实验组/对照组取对数值后得到的各时间节点的数据比值进行取值.若基因显示存在 (prensent),则当比值 r≥1时 ,r取 1;当 -1 (2)对基因号进行对应的数据聚类分析处理:①对数据 (基因)进行大 (较粗糙)的聚类;②对步骤①得到的数据进行再次分类,使得显著性变化的基因有更强的相似性;③利用计算机程序寻找到各时间点具有显著变化的标记 (marker,具有显著性变化的基因);④对寻找到的标记与信噪比散点图进行对比分类,结合步骤①和②进行最终的聚类. 1.4 测试与聚类结果评估 聚类的相似程度与准确程度是评判聚类效果与性能的一个非常重要的指标,但不是唯一的评价指标.评价整个聚类性能的优劣还需要由聚类前的基因选择或特征抽取过程是否具有生物学含义来判定[7-8]. 本研究把整个聚类过程是否具有生物学含义作为评判聚类结果是否优劣的另一个重要指标.一个聚类结果得到以后,是否影响肿瘤等复杂性疾病的作用时间点,应该给出可信度和聚类模型的过程,使实验人员或医务人员能够对结果理解和信服[9-10]. 图2 信噪比散点图Fig.2 Scatter plot of signal to noise ratio 2.1 实验结果 本研究使用芯片数据聚类分析模型方法对小鼠糖尿病细胞数据进行了预测和分析.从得到的上万个数据中筛选出 6 916个基因,在利用信噪比方法处理以后,得到信噪比散点图 (见图2). 图2说明基因的信噪比比值 90%以上都处于0~5之间,大于5的不到10%.本研究的芯片数据共分为 3个时间记录点,分别为第 2周、第 6周、第 12周.之所以选择这 3个时间点作为记录点,是因为在这 3个时间点上,基因表达数据显著性变化较为明显.分别记录下这 3个时间点所对应的信噪比比值,对以上 6 916个基因进行聚类分析,将其分为 8大类,得到的聚类结果分别为 146,1 458,2,718,820,485,2 816,471(各类中所含基因的数目). 本研究选取各组具有显著性变化的基因表达谱的聚类结果进行比较分析,结果如图3所示. 图3(a)类中的基因表达值在第 2周、第 6周、第 12周 3个时间点都维持在一个上升变化较高的水平,这种类型的基因占 2.11%;图3(b)类中的基因表达值在第 2~6周没有太大的上升,而第 6~12周发生显著性上升,这类基因占 21.08%;图3(c)类中的基因表达值在第 2~6周一直保持在一个显著性上升的状态,而第 6~12周开始显著性下降,这类基因占 0.03%;图3(d)类中的基因表达值在第 2~6周显著性上升,而第 6~12周基因表达值未发生明显变化,这类基因占 10.38%;图3(e)类中的基因表达值在第 2~6周由显著性上升趋向于平稳,而第6~12周基因表达值未发生明显变化,这类基因占11.86%;图3(f)类中的基因表达值在第 2~6周显著性下降,而第 6~12周基因表达值未发生明显变化,这类基因占 6.94%;图3(g)类中的基因表达值在第 2~6周未发生明显变化,而第 6~12周基因表达值显著性下降,这类基因占 40.72%;图3(h)类中的基因表达值在第 2~6周显著性下降,并趋向于平稳,而第 6~12周基因表达值未发生明显变化,这类基因占6.81%. 图3 聚类结果比较Fig.3 Compar ison of cluster results 对以上得到的聚类结果进行再次分类,留下第2周、第 6周、第 12周 3个时间点中至少有 2个时间点发生显著变化的基因,剔除仅有 1个时间点发生显著变化的基因.在 6 916个基因中,只留下 1 395个基因.这 1 395个基因在 8大类的基础上又分成了17小类,每类的基因数分别为 146,342,2,66,50,198,33,19,51,365,41,2,9,9,21,18,23,这使得每一小类的基因在表达上具有更强的相似性. 本工作的研究对象 (基因)通过α-干扰素对小鼠的基因表达谱进行诱导,来识别第 2周、第 6周和第 12周的时间点上各基因表达谱的变化,最终确定是否由于这些基因表达谱的变化导致了小鼠糖尿病的发生与形成.通过以上聚类分析的方法,把发生类似显著性变化的基因分在同一类中,为进一步的生物实验与早期临床实验提供可靠的依据与技术支撑. 通过对所有的数据进行聚类分析处理,寻找到了标记 (见表 1). 表 1 聚类寻找到的标记Table 1 M arker s of cluster ing results 对表格 1中的 25个基因与分类结果进行比对,得到结果如下:属于第 7小类的基因有 XM_134383,NM_018764,NM_145435;属于第 12小类的基因有NM_175479,NM_007967;属于第 13小类的基因有NM_028990,NM_146260,NM_178774,NM_145603,XM_484178,AK129101,NM_152839,J00453,NM_080560;属于第 14小类的基因有 NM_146261,NM_021276,XM_127023,NM_145155,XM_489160,NM_139140,NM_009119,NM_010059,XM_129721. 按照之前聚类后得到的 8类结果进行分类,把基因号与注释结果 (NCB I数据库)相比对,其中基因NM_175479,NM_018764,NM_139140,NM_009119,NM_028990,NM_146260,NM_145603与小鼠糖尿病的发生与病变有直接或间接的关系[11].找到这些基因可为后续的生物学实验研究提供了有用的科学依据,为进一步深入研究糖尿病疾病的形成机理,研发基因药物提供重要的信息. 2.2 实验结果比较 通过对以上差异基因的搜索查询 (www.ncbi.nlm.nih.gov),由差异基因的功能注释与分析,寻找到的差异基因的有效性达到了 88%,这比用一般的Gene Cluster软件和 Treeview软件的系统聚类(hierarchical clustering)方法和 K均值方法,无论是在精确度上,还是寻找到的差异基因的有效性上,都有明显的提高.通过对照检索比较,寻找到的基因都具有显著的代表性与差异功能. 基因表达谱的聚类分析是基于实际问题的一种分析过程,因此对聚类结果的解释也是一个非常重要的环节,这就涉及到聚类趋势和聚类有效性等问题的研究.聚类分析过程模型是一个大的分析系统,只有系统中的各部分协调工作,才有可能获得好的聚类分析结果[12].本研究所建立的芯片数据聚类分析模型对小鼠糖尿病发病的预测与控制就是一种有效的方法. 基于基因表达谱的肿瘤及糖尿病等复杂性疾病的聚类结果的检测,一个关键问题就是基因选择或综合属性抽取,但由于基因表达谱数据集的高维性和小样本等特点,使得这个问题最为棘手.信息基因选择算法或者综合属性抽取算法的优劣程度,除了根据其分类性能作为评估标准外,还没有其他统一的评估准则来评判各种分类方法的优劣,希望能尽快指定一个合理的聚类方法评估体系[12].通过聚类分析的研究方法,本研究最后所筛选出的基因也是由基因性能来判断,而没有一个统一的标准来衡量聚类效果的好坏,因此,评判聚类效果一直未有统一的标准.而基于基因表达谱建立的聚类分析模型是一个非常有应用价值的工具,也是理论研究与实际应用紧密结合的尝试,很有希望应用服务于医学临床实践和医学制药等领域,使得预测医学和个性化医学成为可能[13]. 进一步的研究工作包括设计更有效的信息基因选择算法以及研究聚类分析模型中参数的选择等问题.同时,尽可能多地发现与糖尿病密切相关的标记基因,为糖尿病的快速和早期临床精确诊断带来帮助. [1] 王翼飞,史定华.生物信息学:智能化算法及其应用[M].北京:化学工业出版社,2006:221-243. [2] 李瑶.基因芯片数据分析与处理[M].北京:化学工业出版社,2006:162-180. [3] Hua Mei Information.2007年中国血糖仪市场研究报告[R].北京:北京富奥华美信息咨询有限公司,2007:1-16. [4] EZZIANE Z. Applications of artificial intelligence in bioinformatics:a review [J]. Expert Systems with Applications,2006,30(1):2-10. [5] D’HAESELEER P.How does gene exp ression clustering work[J].Nature Biotechnology,2005,23(12):1499-1501. [6] YANG Y H,M ICHAEL J B,DUDOIT S,et al.Comparison of methods for image analysis on cDNA microarray data[J]. Journal of Computational and Graphical Statistics,2002,11(1):1-29. [7] KOKS S,FERNANDES C,KURRIKOFF K,et al.Gene expression profiling reveals upregulation of Tlr4 recep tors in Cckb receptor deficient mice[J].Behavioural Brain Research,2008,188(1):62-70. [8] ALON U,BARKA IN.Broad patterns of gene exp ression revealed by clustering analysisof tumor and normal colon tissues probed by oligonucleotide arrays[J]. Cell Biology Proc Natl Acad SciUSA,1999,11(96):6745-6750. [9] NAKAMURA T,FIDLER I J,COOMBES K R.Gene expression profile of metastatic human pancreatic cancer cells depends on the organ microenvironment[J].Cancer Reserch,2007(1):139-148. [10] YOO C K,LEE I B,VANROLLEGHEMA P A.Interpreting patterns and analysis of acute leukemia gene expression data by multivariate fuzzy statistical analysis[J].Computers and Chemical Engineering,2005,29(6):1345-1356. [11] L IQ,XU B H.Interferon-α initiates type 1 diabetes in nonobese diabetic mice[J].Proc Natl Acad Sci USA,2008,105(34):12439-12444. [12] 王树林,陈火旺,王戟.基于基因表达谱的肿瘤分类研究进展[C]∥生物信息学中的智能计算理论与方法研究.合肥:中国科学技术大学出版社,2007:56-64. [13] TUSHER V G. Significance analysis of microarrays applied to the ionizing radiation response[J].PNAS,2001,4(9):5116-5121. (编辑:刘志强) Cluster Analysis on Gene Chips of a D iabetic An imal M odel SHEN Qing-song, HUANGWen-jia, LÜ Yu-long, WANG Yi-fei O 235 A 1007-2861(2010)04-0409-06 10.3969/j.issn.1007-2861.2010.04.016 2009-03-27 国家自然科学基金资助项目 (30871341);上海市重点学科建设资助项目 (S30104);上海市教委重点学科建设资助项目(J50101) 王翼飞 (1948~),男,教授,博士生导师,研究方向为生物信息学.E-mail:yifei_wang@staff.shu.edu.cn
2 实验结果分析与比较

3 结 束 语
(College of Sciences,ShanghaiUniversity,Shanghai200444,China)

