布氏田鼠封闭群遗传结构的随机扩增多态DNA分析
施海霞,潘思丹,梁 虹,宋铭晶
(中国医学科学院实验动物研究所 北京协和医学院比较医学中心卫生部人类疾病比较医学重点实验室,北京 100021)
研究报告
布氏田鼠封闭群遗传结构的随机扩增多态DNA分析
施海霞,潘思丹,梁 虹,宋铭晶
(中国医学科学院实验动物研究所 北京协和医学院比较医学中心卫生部人类疾病比较医学重点实验室,北京 100021)
目的使用随机扩增多态DNA标记建立标准化的布氏田鼠封闭群遗传质量控制分子标记库。方法使用高盐沉淀法从鼠尾中提取布氏田鼠基因组DNA。采用40条 PRAD引物对布氏田鼠封闭群进行 PCR扩增,琼脂糖电泳分离条带,参考标准分子量标记计算条带大小,并使用多态位点数、单态位点数以及多态位点比率评价种群的遗传多样性。结果筛选出8个能获得清晰稳定扩增条带的 RAPD标记。这8个RAPD标记检测到的多态位点数存在明显差异。8个引物得到的遗传多态位点的数据之和能揭示种群的遗传结构。结论本实验建立了检测布氏田鼠封闭群遗传结构的RAPD标记。
布氏田鼠;实验动物化;随机扩增多态DNA;封闭群
利用实验动物进行生命科学研究是假定动物个体间在遗传上是均质的,没有差异的。但是实际上由于遗传变异,不同个体在同一座位上形成许多不同的等位基因形式,根据变异发生位点的不同可以分为两种情况:同一基因座位内,同一位点上发生突变形成的一系列等位基因称为同点等位基因(homoallele);在同一个基因座位内不同位点上发生突变形成的一系列等位基因称为异点等位基因(heteroallele)。动物群体内一个基因座位的等位基因的成员可以多达三个以上,这些等位基因被称为复等位基因[1]。因此,个体间的遗传差异是普遍存在的,动物品系中同一近交系内个体间遗传差异最小,而封闭群内个体间的遗传差异较大。我们2007年从中国农业大学引入了布氏田鼠(Lasiopodomys brandtii)普通级实验室种群,通过对该种群的系统药物净化和同胞选择筛选,2010年建立了连续繁殖5代的清洁级布氏田鼠封闭群[2]。国内一些实验室进行了布氏田鼠种群的繁殖、生理、生化、免疫以及遗传结构等方面的一系列研究[3,4],但对该物种的医学实验动物化研究和应用还基本上是空白。我们建立该布氏田鼠封闭群标准化的遗传质量控制分子标记,明确种群内不同个体的遗传背景,对促进布氏田鼠的实验动物化研究具有重要意义。
随机 扩 增 多 态 DNA(randomlyamplified polymorphic DNA,RAPD)标记技术所用的随机引物同基因组DNA序列有其特定的结合位点,这些结合位点在基因组的分布如符合PCR扩增条件,即在模板的两条链上都有互补位置,且3’端相距在 PCR扩增长度范围之内,就可扩增出片段。如果基因组在这些区域发生 DNA片段的插入、缺失或碱基突变,就导致这些结合位点的分布发生相应的变化。通过对RAPD扩增产物的检测就可以快速地检测到基因组DNA在这些区域内的多态性,并且所需模板DNA的量较少,操作简单,实验成本低,在动植物品系鉴定和遗传多样性的分析中已得到广泛的应用[5,6]。本课题利用 RAPD技术,检测布氏田鼠封闭群的遗传结构,建立遗传质量控制分子标记库,以便于我们就能监测和控制布氏田鼠封闭群的遗传多样性,并依据布氏田鼠的遗传背景和实验的目的为科学实验提供实验动物,保证实验结果的可重复性和准确性。
1 材料和方法
1.1 实验动物
布氏田鼠饲养在独立送排风净化动物笼(individually ventilated cage,IVC),饲养室温度22℃~25℃,湿度50%~70%,光照条件14L:10D,自由摄食和饮水。
1.2 DNA提取
布氏田鼠基因组 DNA来自自清洁级布氏田鼠雌雄各25只,采用高盐沉淀法提取,具体步骤如下:取布氏田鼠尾部组织置于1.5 mL离心管中,加入500 μL裂解液和10 μL蛋白酶K(20 mg/mL),55℃消化过夜,待鼠尾组织完全溶解仅有少许尾骨残骸后,加入300 μL 6 mol/L饱和 Nacl混匀,冰上静置15 min,12000 r/min离心15 min后取上清,加入与上清等体积的异丙醇沉淀 DNA,70%乙醇清洗两次,然后倒掉乙醇自然风干,100 μL TE溶解沉淀,琼脂糖凝胶电泳检测 DNA浓度,调整浓度至 50 ng/μL。
1.3 仪器和试剂
水平电泳仪 (BIO-RAD)、高速离心机(Beckman)、PCR仪(BIO-RAD)、凝胶图像分析系统(Tannon)。蛋白酶K购自SIGMA公司,Taq DNA聚合酶、10×buffer、MgCl2、dNTP以及 DNA Marker购自宝生物有限公司(TaKaRa)。引物在上海英骏生物技术有限公司合成。
1.4 引物的筛选
用40条RAPD引物分别对随机选取的6只布氏田鼠(雌雄各半)基因组DNA进行PCR扩增,每个个体分别使用25 ng和50 ng进行PCR扩增,选择那些在调节好适宜的模板浓度后在所有个体均能获得稳定重复的扩增产物的引物用于布氏田鼠种群的RAPD实验,筛选出的引物和扩增产物情况见表1。
1.5 PCR反应条件与电泳
20 μL反应体系含10×buffer 2 μL,25 mmol/L Mgcl21 μL,10 mmol/L dNTP 0.4 μL,50 mmol/L引物1 μL,1U Taq DNA聚合酶。扩增程序为:94℃预变性3 min,然后进入37个循环,包括94℃变性1 min,37℃退火30 s,72℃延伸1 min。最后一个循环增加72℃延伸10 min。RAPD产物行1.5%琼脂糖凝胶(含0.5 μg/mL的溴化乙锭)电泳分离,电泳电压30V,5 h。紫外线下观察拍照。
1.6 统计分析
每个条带都记做一个位点的基因,某位点上有条带的个体的等位基因标记为1,无条带的记为0,所有个体在该位点都有条带,即等位基因的频率等于1,记为单态位点。多态位点是指等位基因的频率小于或等于0.99的位点。多态位点比率是指其等位基因的频率小于或等于0.99的位点占本实验中统计的所有位点的比率。
2 结果
本研究用8个RAPD引物对50只布氏田鼠进行了PCR扩增,共扩增出 66条带,都位于 250~2000bp之间,其中多态片段37条(表1)。部分扩增图谱见图1~3。

表1 布氏田鼠封闭群遗传结构分析的RAPD引物及扩增情况Tab.1 Sequences of primers used in RAPD assay for closed colony of Brandt's vole
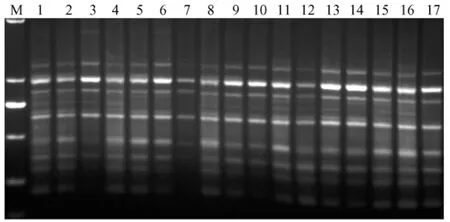
图1 引物OPT01对布氏田鼠的RAPD扩增产物Fig.1 The RAPD products of the genomic DNA of Lasiopodomys brandtii amplified by OPT01
3 讨论
RAPD扩增产物的差异可以归为三类:第一类是来自个体某座位在引物结合区的突变,造成某片段有无的差异;第二类是个体某高变区的插入和缺失突变,造成个体间扩增片段大小的差异;第三种是显性纯合子与杂合子造成片段扩增条带浓度的差异,但是这种产量的差异也可能是在扩增时产物随机竞争产生的,无法据此判定该条带是显性纯合还是杂合,因此,本研究中无论是弱带还是强带都作为有条带处理,在该基因的位点标记为1。
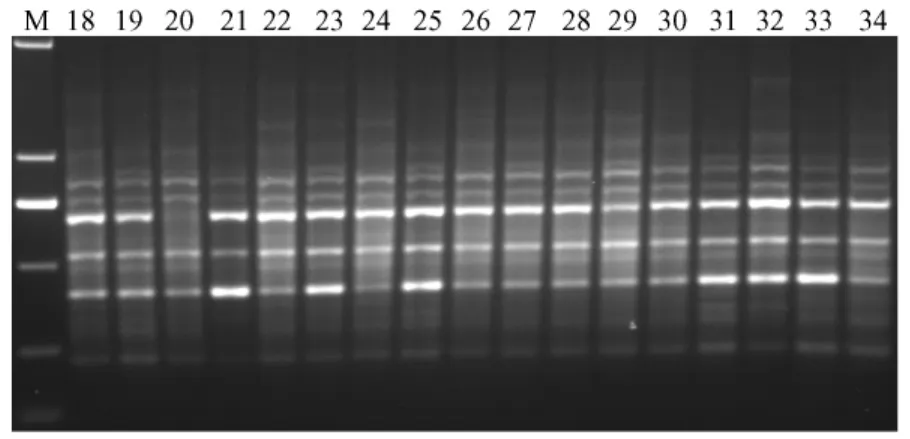
图2 引物OPT07对布氏田鼠的RAPD扩增产物Fig.2 The RAPD products of the genomic DNA of Lasiopodomys brandtii amplified by OPT07
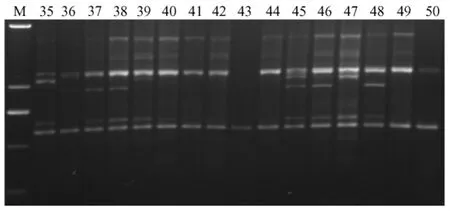
图3 引物OPZ03对布氏田鼠的RAPD扩增产物Fig.3 The RAPD products of the genomic DNA of Lasiopodomys brandtii amplified by OPZ03
结果显示不同引物检测到大仓鼠种群的多态位点率明显不同,引物OPT15、OPT-07和OPT-13检测到的多态位点比率较低,分别是0.364、0.375和0.375。而引物 OPT16和 OPZ03,OPZ07检测到的多态位点率较高,在0.70以上。因为RAPD标记所用的引物是由10个核苷酸组成的寡核苷酸序列,与模板DNA可能有的结合位点位点很多,一套引物可以检测整个基因组[7],如果与引物互补的DNA区段是保守区段,那么不同个体间一般呈现相同的扩增产物;如果与高变区段互补,那么扩增产物则表现出个体间的丰富多态,所以本研究不同引物检测到的多态位点率的差异,可能源于不同引物所扩增的模板DNA的区域不同。引物OPT-16扩增出的片段只有9条,但多态片段达到8条,多态位点比率为0.889,推测其扩增区域为高变区。而引物 OPT15扩增出的片段为11条,但多态片段最少,只有4条,多态位点比率为0.364,推测其扩增区域为保守区。结果显示利用不同引物对这个布氏田鼠封闭群进行RAPD扩增,个体间的带型有的比较相似,如OPT-01的扩增结果(图1)。有的带型基本一致,只有少部分个体的部分位点扩增出多态片段,如OPT-07的扩增结果(图2)。有的带型存在明显差异,如OPZ-03的扩增结果(图3)。引物扩增条带的相似程度反映种群内不同个体间遗传背景的相似程度。任何一条引物得到的遗传变异信息都有限,不能区分出所有个体,但是8条引物得到的遗传多态位点的数据库能揭示种群内个体间的遗传差异和种群的遗传多样性。
[1] 赵寿元,乔守怡.遗传学[M].北京:高等教育出版社,2002: 257-289,290-327.
[2] 梁虹,潘思丹,施海霞,等,布氏田鼠封闭群建立及繁殖特性研究[J].中国卫生检验杂志,2009,19(12):2767-2769.
[3] Li FH,Zhong WQ,Wang ZX,et al.Rank in a food competition test and humoralimmunefunctionsin male Brandt'svoles (Lasiopodomys brandtii)[J].Physiol Behav,2007,90:490-495.
[4] Cai XQ,Yang M,Zhong WQ,et al.Humoral immune response suppresses reproductive physiology in male Brandt's voles (Lasiopodomys brandtii)[J].Zoology,2009,112:69-75.
[5] 宋国华,刘田福 中国地鼠 RAPD分析体系的优化[J].中国比较医学杂志,2005,15(2):84-87.
[6] 王洪,岳秉飞,王钜,等,长爪沙鼠与实验大小鼠 RAPD图谱比较研究[J].中国比较医学杂志,2006,16(6):353-355.
[7] 蔡月琴,屠珏,余佳,等,RAPD标记技术用于WHBE兔近交系培育中的遗传分析[J].中国实验动物学报,2009,17(5): 326-329.
RAPD Analysis of the Genetic Structure for Closed Colony of Brandt's Vole
SHI Hai-xia,PAN Si-dan,LIANG Hong,SONG Ming-jing
(Key Laboratory of Human Diseases Comparative Medicine,Ministry of Health;Institute of Laboratory Animal Science,Chinese Academy of Medical Sciences(CAMS)&Comparative Medicine Centre,Peking Union Medical College(PUMC),Beijing 100021,China)
ObjectiveTo establish the he genetic structure for cultivation population of Brandt's vole (Lasiopodomys brandtii)by RAPD(randomly amplified polymorphic DNA)assay.Method Genomic DNA was extracted from tail tip following high-concentration-salt precipitation methods.40 RAPD primers were PCR amplified to analyze the DNA structure of Brandt's vole population.RAPD markers were separated by agarose gel electrophoresis.Alleles were determined according to a size marker.Monomorphic site,polymorphic site and the proportion of polymorphic loci,were used to describe the genetic variation of Brandt's vole population detected by RAPD。ResultsThe rates of variation of RAPD loci by 8 primers used in this study are different.The combination of the PCR bandings by eight RAPD primers can provide sufficient statistical power to assess the genetic variations in Brandt's vole population。ConclusionWe have successfully established genetic methods for analyzing genetic structure of closed colony of Brandt's vole.
Brandt's vole;Cultivation;RAPD;Closed colony
R394.5
A
1671-7856(2010)07-0049-04
2010-03-31
高等学校博士学科点专项科研基金(20070023119),中央级公益性科研院所基本科研业务费专项基金(DWS200702),以及国家科技支撑计划课题(2009BAI83B01)子课题资助。
施海霞,女,初级实验技师。
宋铭晶。E-mail:songmj@cnilas.org

