氧及活性氧代谢相关基因在大鼠肝脏再生中的作用*
蔺 芳,张家洋,皮川真,徐存拴
(1.新乡学院生命科学与技术系,河南新乡453000;2.华兰生物工程股份有限公司,河南新乡453000;3.河南省-科技部共建细胞分化调控重点实验室,河南新乡453000)
氧为机体代谢所必需。氧的一些代谢产物,如活性氧(reactive oxygen species,ROS)有重要生理作用[1]。氧及活性氧代谢包括氧化反应、谷胱甘肽结合反应、过氧化氢代谢及超氧化物代谢等[2]。正常情况下,细胞有清除活性氧、维持细胞氧化还原内环境稳定功能,但是,当活性氧产生过多或细胞保持氧化还原稳定机制发生紊乱时,会造成活性氧在体内积累,并损伤核酸、蛋白质、脂类等生物大分子,导致机体衰老和疾病,如肿瘤、心脑血管病等。研究表明,肝脏有很强再生能力[3-4]。部分肝切除(partial hepatectomy,PH)[5]后,残留的肝细胞增生以补偿丢失的肝细胞,并且可精确感知再生肝的大小,适时停止再生[6-7],这个过程称为肝再生(liver regeneration,LR)。肝再生包括肝细胞数量增多和肝脏体积增大。涉及细胞激活、去分化、增殖及其调控、再分化、组织结构和功能重建等生理生化过程[8],受到包括氧及活性氧在内的多种因素调控。通常根据细胞生理活动将肝再生分为启动(PH后0.5 h~4 h)、G0/G1过渡(PH后4 h~6 h)、细胞增殖(PH后6 h~66 h)、细胞分化和组织结构功能重建(PH后72 h~168 h)等四个阶段。根据时间进程分为早期(PH后0.5 h~4 h)、前期(PH后6 h~12 h)、中期(PH后16 h~66 h)、后期(PH后72 h~168 h)等四个时期。为在基因转录水平[9]了解氧及活性氧代谢相关基因在大鼠肝再生中作用,用含184个上述代谢相关基因的Rat Genome 230 2.0芯片[10]检测了大鼠2/3肝切除后再生肝基因表达情况。在此基础上,进一步分析了其在肝再生中的表达动态、与肝再生的相关性和作用。
1 材料与方法
1.1 材料
1.1.1 实验动物 试验用SD纯系大鼠由河南师范大学实验动物中心提供,体重200 g~250 g。
1.1.2 主要试剂与仪器 GeneChip全自动洗涤工作站450和高分辨芯片扫描仪3000为美国Affymetrix公司产品;Trizol试剂盒为Invitrogen公司产品;RNeasy mini试剂盒为QIAGEN公司产品;其他试剂为分析纯。
1.2 方法
1.2.1 样品制备 将264只大鼠随机分组,每组6只。按Higgins G M等[5]方法切除大鼠肝左叶和中叶,之后残留肝细胞通过细胞增殖由基本不生长状态转变为快速生长状态,以补偿丢失、损伤的肝组织和恢复肝脏的生理功能,这些在切除剩余肝叶上重新生长出的部分肝组织即为再生肝。并于0.5、1、2、4、6、8、12、16、18、24、30、36、42、48、54、60、66、72、96、120、144、168 h的相应恢复时间颈椎脱臼处死动物,切取再生肝,置4℃PBS中涮洗3次,从右叶再生肝中部取100 mg~200 mg组织,将每组的来源于6只大鼠的样品混合,总肝量为(0.1~0.2)g×6=0.6 g~1.2 g,于—80℃保存备用。对照组材料为正常成年大鼠肝。假手术(sham-operation,SO)组除不切除肝叶外,其他同部分肝切除组。试验中严格遵循中国动物保护法。
1.2.2 总RNA提取与纯化 总RNA提取按Invitrogen公司的Trizol试剂盒操作程序进行[11]。总RNA纯化按QIAGEN公司的RNeasy mini试剂盒操作程序进行[12]。用琼脂糖凝胶电泳(180 V,0.5 h)检测总RNA的28 S和18 S比例;其亮度约为2∶1;在260/280 nm波长下测定总RNA浓度和纯度[13]。
1.2.3 cDNA、cRNA的合成和纯化 cDNA、cRNA合成按Affymetrix公司方法进行。合成cDNA时,模板量为1 μ g~8 μ g总RNA。合成生物素标记的cRNA时,取12 μ L上述cDNA溶液作模板。cDNA、cRNA纯化按基因芯片分析样品纯化操作程序进行。两者的浓度、纯度和质量检测方法同上。
1.2.4 cRNA片断化和芯片检测 取15 μ L浓度为1 μ g/μ L的cRNA,加6 μ L 5×片断化缓冲液、9 μ L无RNA酶水混匀,94℃温浴35 min,得到长度为35 bp~200 bp的cRNA片断。按Affymetrix公司提供的配方配制杂交液,然后将杂交液加至经预杂交处理的Rat Genome 230 2.0芯片中,于45℃、60 r/min杂交16 h,吸去杂交液,用GeneChip全自动洗涤工作站450(Affymetrix公司,USA)洗涤和染色芯片。用高分辨芯片扫描仪3000(Affymetrix公司,USA)扫描芯片,获得基因表达信号值。
1.2.5 芯片检测参数的获取 用GCOS1.2软件读取、处理信号值数据。获得归一化后的信号值、信号检出(P,A,M)以及试验和对照组的比值。
1.2.6 芯片检测参数的校正 为减少芯片分析误差,用Rat Genome 230 2.0芯片对每个时点再生肝重复检测3次。将3次检测的平均值视为矫正值,然后用Gene Math,Gene Spring(Silicon Genetics,San Carlos,CA),Microsoft Excel(Microsoft,Redmond,WA)等分析软件对各组数据进行统计和聚类分析[14-15]。
1.2.7 肝再生相关基因的确认 根据GENEONTOLOGY(www.geneontology.org)网站的生物活动分类,将具体的氧及活性氧代谢一词输入NCBI(www.ncbi.nlm.nih.gov)和RGD(rgd.mcw.edu)等网站查找大鼠、小鼠和人的相关基因,并根据GENMAPP(www.genmapp.org)、KEGG(www.genome.jp/kegg/pathway.html)和BIOCARTA(www.biocarta.com/genes/index.asp)等网站的氧及活性氧代谢途径图汇总上述活动相关基因。然后查阅相关文献资料对上述基因进行进一步确认。除大鼠基因外,将现在认为存在于人/小鼠、在大鼠肝再生中发生有意义表达变化的基因视为大鼠同源基因。最后把3次检验结果相同或相似、至少在部分肝切除后一个表达上调或下调两倍以上、部分肝切除组与假手术组差异显著(0.01<P<0.05)或极显著(P<0.01)的大鼠基因和大鼠同源基因视为肝再生相关基因。
2 结果
2.1 氧及活性氧代谢相关基因在大鼠肝再生中表达变化
查NCBI、GENMAPP、KEGG、BIOCARTA、RGD等网站资料表明,178个基因参与氧及活性氧代谢。查Rat Genome 230 2.0芯片资料表明,该芯片含上述的150个基因。其中81个基因至少在部分肝切除后一个时点发生了有意义的表达变化,并且部分肝切除组与假手术组有显著或极显著差异、三次Rat Genome 230 2.0芯片检测结果具有可重复性。因此,初步确认这些基因与肝再生相关。其中,33个基因表达上调,20个基因表达下调,28个基因在有的时点表达上调、有的时点表达下调(简称上/下调表达)。它们的上调范围为对照的2倍~60倍,下调范围为对照的2倍~10倍(表1)。
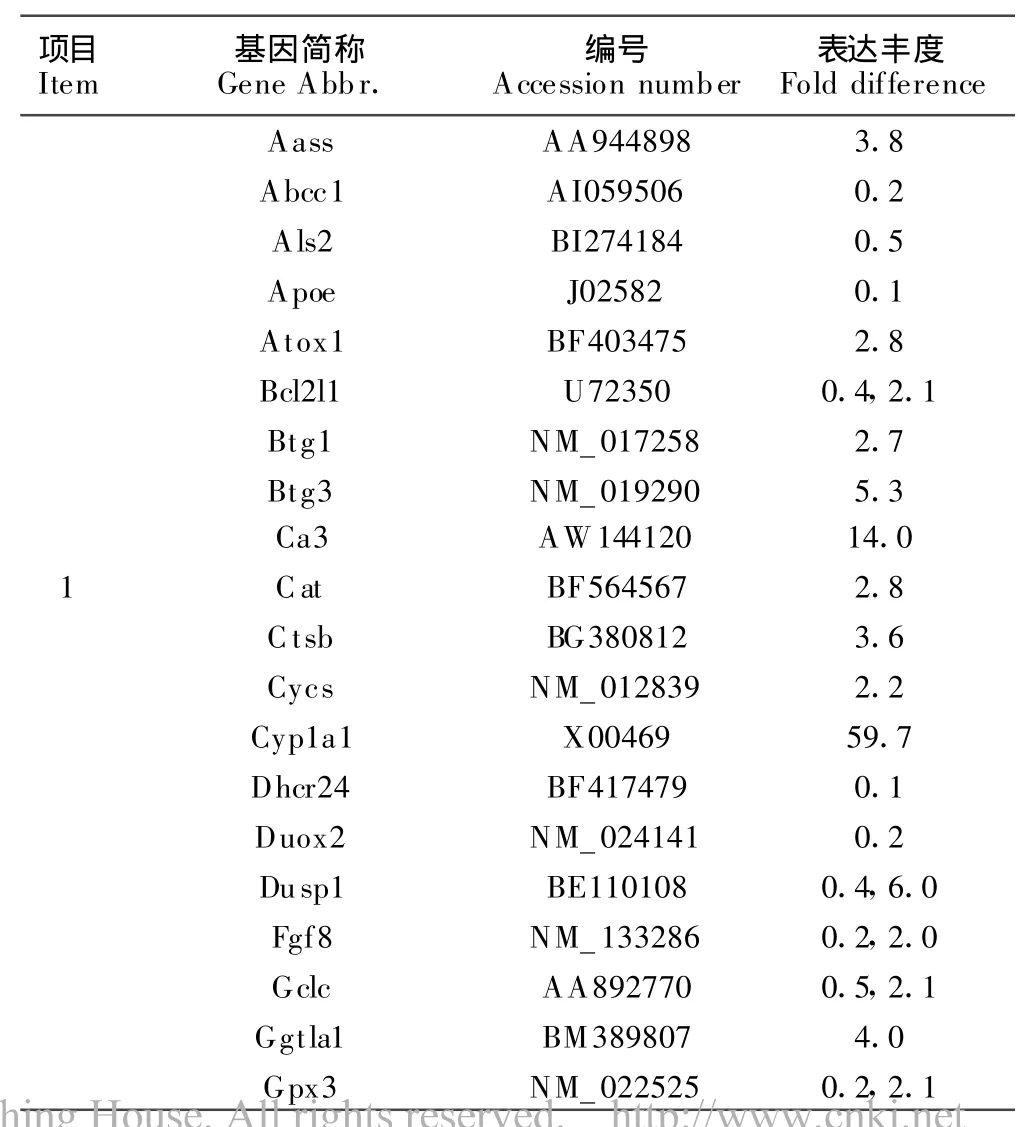
表1 81个氧及活性氧代谢相关基因在大鼠肝再生中表达丰度Table 1 Ex pression abundance of 81 oxygen and reactive oxygen species metabolism-associated genes during rat liver regeneration

(续表)

(续表)
2.2 氧及活性氧代谢相关基因在大鼠肝再生中起始表达及总表达情况
分析表明,肝再生各时点起始上调和下调及总表达的基因数为:0.5 h时均为13和4;1 h时12和4,23和6;2 h时4和2,18和4;4 h时1和2,17和4;6 h时3和3,21和7;8 h时0和1,18和8;12 h时0和4,11和11;16 h时3和5,13和9;18 h时3和4,18和11;24 h时1和1,15和10;30 h时3和1,11和11;36 h时0和1,9和6;42 h时0和1,12和5;48 h时0和0,17和9;54 h时0和0,14和8;60 h时1和0,13和8;66 h时2和0,16和7;72 h时0和0,12和6;96 h时0和0,12和6;120 h时0和1,18和9;144 h时0和0,9和5;168 h时0和0,7和7(图1)。肝再生中基因起始表达的总体情况是:46个基因起始表达上调,35个基因起始表达下调。其中,在肝再生启动阶段(PH后0.5~4 h)30个基因起始上调,12个基因起始下调;G0/G1过渡阶段(PH后4~6 h)4个基因起始上调,5个基因起始下调;细胞增殖阶段(PH后6 h~66 h)16个基因起始上调,21个基因起始下调;细胞分化和组织结构功能重建阶段(PH后72 h~168 h)0个基因起始上调,1个基因起始下调。肝再生中基因表达的总趋势是:共表达上调317次,下调161次。其中,肝再生启动阶段(PH后0.5 h~4 h)上调71次、下调18次;G0/G1过渡阶段(PH后4 h~6 h)上调38次、下调11次;细胞增殖阶段(PH后6 h~66 h)上调188次、下调110次;细胞分化和组织结构功能重建阶段(PH后72 h~168 h)上调58次、下调33次(图1)。基因表达贯穿于整个肝再生过程。在0.5~2、30和60~66 h起始表达上调基因占优势,在4、8~18、36~42和120 h起始表达下调基因占优势,其他时点不再有基因起始表达。
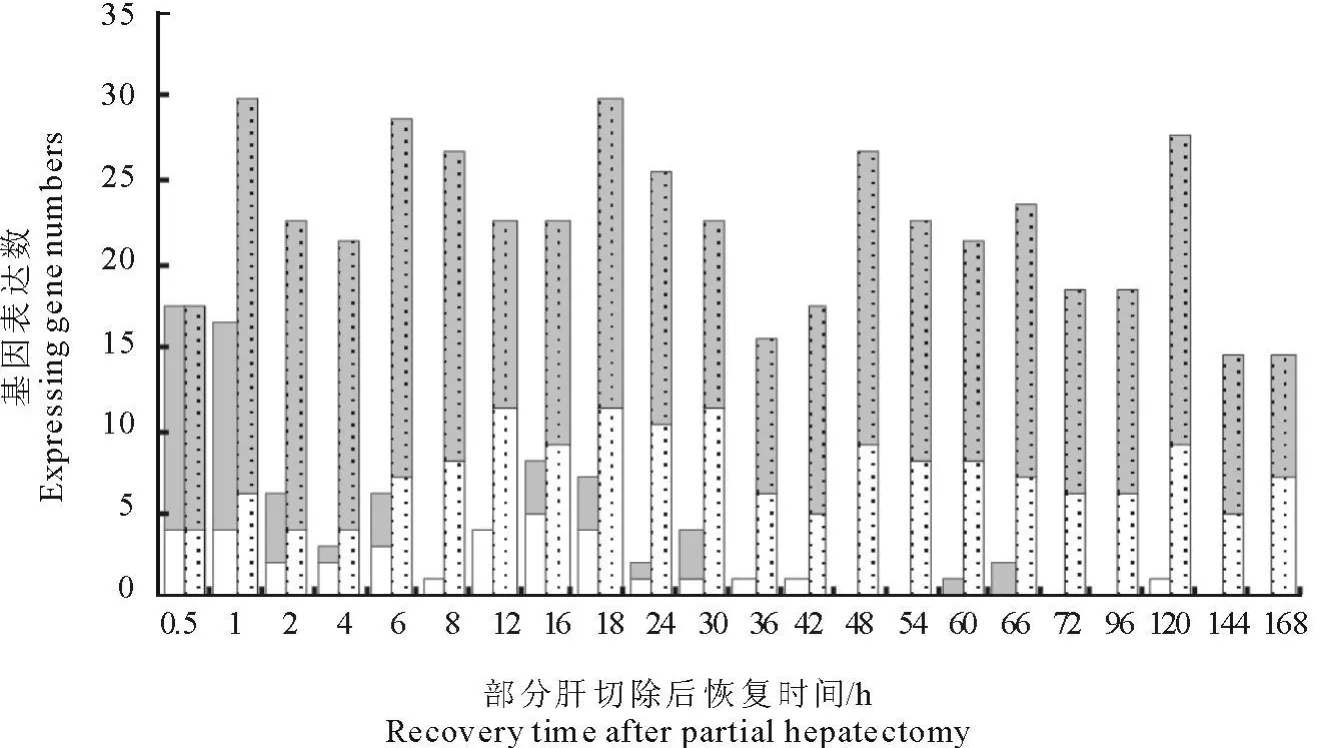
图1 81个氧及活性氧代谢相关基因在大鼠肝再生各时点起始表达及总表达情况Fig.1 The initial and total expression profiles of 81 genes associated with oxygen and reactive oxygen species metabolism at each time point of rat liver regeneration
2.3 氧及活性氧代谢相关基因在大鼠肝再生中表达方式
根据功能和在肝再生中变化将上述81个基因的表达方式分为6类(图2)。①参与氧化反应(根据参与氧化反应中基因表达的相似性又分为1-a~1-g共7小类)。23个基因上调,15个下调,17个上/下调;②参与谷胱甘肽结合反应。4个上调,2个上/下调;③参与过氧化物合成。1个下调;④参与过氧化物分解。2个上调,1个下调,1个上/下调;⑤参与超氧化物释放。2个下调,3个上/下调;⑥参与超氧化物清除。2个上调。
3 讨论
在氧化反应相关基因中,促进活性氧合成的cyp1a1等3个基因几乎在整个肝再生中表达上调,通过抑制硫氧还蛋白的活性来调节氧化应激的txnip等20个基因在肝再生早期表达上调,保护细胞抗氧化的gsr等27个基因在肝再生中期表达上调,维持氧化还原内环境稳定的btg1等3个基因在肝再生后期表达上调,推测几乎整个肝再生中氧化反应加强。其中,cyp1a1在部分肝切除后0.5~24、36、48~72、120 h表达上调,12 h达到高峰,是对照的59.7倍,推测它在再生肝的氧化反应中起关键作用。
在谷胱甘肽结合反应相关基因中,催化谷胱甘肽结合反应并有解毒作用的gstm4等6个基因几乎在整个肝再生中表达上调。推测谷胱甘肽结合反应在肝再生中表达增强。其中,gstm4在1、18、48 h表达上调,1 h达到高峰,是对照的7.5倍。推测它在再生肝的谷胱甘肽结合反应中起关键作用。

图2 81个氧及活性氧代谢相关基因在大鼠肝再生中表达方式Fig.2 Expression patterns of 81 genes associated with oxygen and reactive oxygen species metabolism during rat liver regeneration
在过氧化氢代谢相关基因中,催化过氧化氢合成的duox2在肝再生早期和前期表达下调,相应时期分解过氧化氢的cat等4个基因表达上调。推测肝再生早期和前期过氧化氢代谢加强。
在超氧化物代谢相关基因中,促进超氧化物释放的alox12等5个基因和与过氧化物清除有关的mt3主要在肝再生早期和前期表达上调。推测肝再生相应时期超氧化物代谢增强。其中,nox1在0.5~8、18、120 h表达上调,2 h达到高峰,是对照的8.6倍。推测它在再生肝的超氧化物释放中起关键作用。
综上所述,本试验从较长时间和较多时点入手,用高通量基因表达谱芯片从基因转录水平分析了部分肝切除后氧及活性氧代谢相关基因在肝再生中表达变化,初步证实肝再生早期和前期过氧化氢和超氧化物代谢加强;几乎在整个肝再生中氧化反应和谷胱甘肽结合反应增强。然而,从基因→mRNA→蛋白质→功能等受包括蛋白互作在内的多种因素影响,今后将进一步用Northern blot、蛋白质芯片、RNA干扰、蛋白互作等技术验证上述结果。
[1] Lakshmi S V,Padmaja G,Kuppusamy P,et al.Oxidative stress in cardiovascular disease[J].Indian J Biochem Biophys,2009,46(6):421-440.
[2] 李建喜,杨志强,王学智.活性氧自由基在动物机体内的生物学作用[J].动物医学进展,2006,27(10):33-36.
[3] Kandilis A N,Koskinas J,Tiniakos D G,et al.Liver regeneration:focus on cell types and topographic differences[J].Eur Surg Res,2010,44(1):1-12.
[4] Pathikonda M,Munoz S J.Acute liver failure[J].Ann Hepatol,2010,9(1):7-14.
[5] Higgins G M,Anderson R M.Experimental pathology of the liver:restoration of the liver of the white rat following partial surgical removal[J].Arch Pathol,1931,12:186-202.
[6] Kung J W,Forbes S J.Stem cells and liver repair[J].Curr Opin Biotechnol,2009,20(5):568-574.
[7] Xu C S,Shao H Y,Du B.Study on correlation of signal molecule genes and their receptor-associated genes with rat liver regeneration[J].Genome,2009,52(6):505-523.
[8] Zhao Q,Ren H,Zhu D,et al.Stem/progenitor cells in liver injury repair and regeneration[J].Biol Cell,2009,101(10):557-571.
[9] Xu C,Lin F,Qin S.Relevance between lipid metabolism-associated genes and rat liver regeneration[J].Hepatol Res,2008,38(8):825-837.
[10] Collins J F.Gene chip analyses reveal differential genetic responses to iron deficiency in rat duodenum and jejunum[J].Biol Res,2006,39(1):25-37.
[11] Nuyts S,Van Mellaert L,Lambin P,et al.Efficient isolation of total RNA from Clostridium without DNA contamination[J].J Microbiol Meth,2001,44(3):235-238.
[12] Schippert R,Schaeffel F,Feldkaemper M P.Microarray analysis of retinal gene expression in Egr-1 knockout mice[J].Mol Vis,2009,15:2720-2739.
[13] Li L,Roden J,Shapiro B E,et al.Reproducibility,fidelity,and discriminant validity of mRN A amplification for microarray analysis from primary hematopoietic cells[J].J Mol Diagn,2005,7(1):48-56.
[14] Kundel A,Zarnegar R,Kato M,et al.Comparison of microarray analysis of fine needle aspirates and tissue specimen in thyroid nodule diagnosis[J].Diagn Mol Pathol,2010,19(1):9-14.
[15] Werner T.Cluster analysis and promoter modelling as bioinformatics tools for the identification of target genes from expression array data[J].Pharmacogenomics,2001,2(1):25-36.
- 动物医学进展的其它文章
- 猪流感病毒H1N1广东分离株HA基因的克隆与进化分析

